Revisão
|
|
| Avanços em técnicas ômicas na descoberta e no estudo de produtos naturais Advances in omics techniques for the discovery and study of natural products |
|
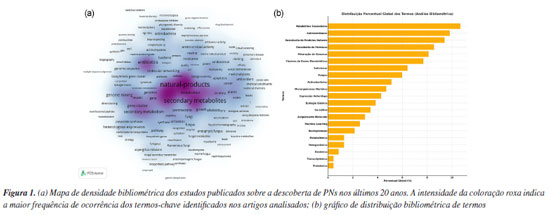
Júlio C. J. BarbosaI I. Instituto de Química, Universidade Estadual de Campinas, 13083-970 Campinas - SP, Brasil Recebido: 03/02/2025 *e-mail: taicia@gmail.com; camilavic@usp.br Humanity has historically turned to nature to treat diseases, with records dating back to the Neanderthals, who already used medicinal plants. To this date, biologically active natural products (NP) remain indispensable sources in drug development for several therapeutic areas, due to their vast structural diversity and unique ability to interact with specific biological targets. With the advancement of omics sciences, NP research has been revolutionized by omics approaches such as genomics, transcriptomics, proteomics, and metabolomics. These techniques are widely applied to identify biosynthetic pathways and search for new therapeutic targets, utilizing plants, microorganisms, and other natural sources. The integration of these approaches into multi-omics strategies has proven to be highly effective for the discovery of new NPs, offering significant advantages and avoiding the re-isolation of already known molecules. Furthermore, they provide a comprehensive view of the molecular mechanisms involved in NP production, enabling more targeted and efficient exploration. This combination of multidisciplinary approaches is crucial for the development of more selective and effective drugs, contributing to the advancement of safer and more innovative treatments. This review will contextualize the main omics techniques used in NP discovery, while providing examples of their use and new perspectives in NP discovery. INTRODUÇÃO Ao longo de sua extensa história, a humanidade frequentemente recorreu à natureza em busca de alternativas para tratar e aliviar doenças, uma prática que precede o entendimento científico detalhado sobre as patologias.1 Um dos registros mais antigos do uso de plantas para fins medicinais data aproximadamente 60.000 a.C., conforme revelado no sepultamento de um homem Neandertal no sítio arqueológico de Shanidar (Iraque). Estudos paleoantropológicos na caverna de Shanidar evidenciaram depósitos de pólen provenientes de plantas com propriedades medicinais.2 Além de plantas, animais e fungos também foram reconhecidos como valiosas fontes naturais de compostos bioativos.3 Os produtos naturais (PNs) compreendem uma diversidade de moléculas bioativas oriundas de organismos vivos e representam grande parte dos antibióticos, imunossupressores, antitumorais, antifúngicos e antimaláricos em uso.4-6 Essenciais em diversas funções ao longo dos séculos, os PNs não apenas tiveram um papel central na medicina tradicional, mas continuam sendo uma das fontes mais relevantes para a descoberta e desenvolvimento de fármacos.6-9 Um exemplo é a penicilina, um dos primeiros e mais utilizados antibióticos no mundo.10 A penicilina foi descoberta acidentalmente em 1928 por Alexander Fleming, através da constatação que culturas de Staphylococcus aureus não se desenvolviam em áreas contaminadas pelo fungo Penicillium chrysogenum. Essa observação levou-o à identificação da substância com propriedades antibacterianas, que mais tarde seria chamada de penicilina. Embora Fleming tenha feito essa descoberta, foi Boris Ernst Chain, bioquímico britânico, que determinou a estrutura química da penicilina e desenvolveu métodos para sua produção em larga escala.11 Além disso, Howard Florey, patologista australiano, foi o responsável por conduzir os primeiros ensaios clínicos que demonstraram a eficácia da penicilina no tratamento de infecções bacterianas. Em reconhecimento às suas contribuições fundamentais para a descoberta e o desenvolvimento da penicilina, Fleming, Chain e Florey receberam o Prêmio Nobel de Fisiologia ou Medicina em 1945.12 Até hoje, a penicilina continua sendo um antibiótico essencial no tratamento de diversas infecções bacterianas. Outro exemplo é a artemisinina, um importante fármaco antimalárico. Sua descoberta foi realizada por Youyou Tu,13 farmacologista chinesa, que recebeu o Prêmio Nobel de Fisiologia ou Medicina em 2015 por suas contribuições fundamentais no tratamento da malária com esse terpeno. A artemisinina é um sesquiterpenóide, encontrado majoritariamente na planta Artemisia annua. A pesquisadora Youyou Tu liderou o projeto 523, uma iniciativa do governo chinês para buscar alternativas para um tratamento eficaz da malária, doença que afetava gravemente os soldados durante a Guerra do Vietnã.14 Sua eficácia rápida e potente contra o parasita Plasmodium falciparum reduziu significativamente a mortalidade associada à malária. A artemisinina permanece como uma molécula crucial para o combate à malária e como alternativa para síntese de novos medicamentos antimaláricos.15 As abordagens tradicionais para a descoberta de PNs baseavam-se principalmente no isolamento dos metabólitos por meio de longos ciclos de fracionamento e triagem biológica, seguidos pela caracterização dos compostos de interesse por técnicas espectroscópicas e espectrométricas. Atualmente, as técnicas ômicas, tais como genômica, transcriptômica, proteômica e metabolômica, representam o estado da arte na pesquisa em PNs. As técnicas ômicas representam uma revolução na biologia e na biotecnologia, proporcionando um entendimento profundo e abrangente dos sistemas biológicos.16-18 Historicamente, essas técnicas evoluíram significativamente desde o advento do sequenciamento do ácido desoxirribonucleico (DNA) nas décadas de 1970 e 1980, passando por avanços importantes com o Projeto Genoma Humano no início dos anos 2000, até a atual era de big data e análise de sistemas biológicos complexos.19-22 Uma pesquisa bibliométrica foi conduzida para identificar estudos relevantes sobre a descoberta de PNs utilizando abordagens ômicas e estratégias multiômicas. A seleção dos artigos foi realizada com base em uma busca sistemática nas principais bases de dados científicas, incluindo Web of Science, PubMed, e Scopus, utilizando os seguintes termos-chave: "natural products", "omics approaches", e "multi-omics strategies". Para garantir a abrangência da pesquisa, foram aplicados filtros que selecionaram majoritariamente artigos publicados nos últimos 20 anos, refletindo as tendências mais atuais no campo da descoberta de PNs. A análise bibliométrica contemplou um conjunto representativo de termos-chave extraídos dessas bases de dados, compilando os estudos relevantes, conforme ilustrado na Figura 1a.
A distribuição temática revelou que os tópicos mais recorrentes foram "metabólitos secundários" (25,6%), "descoberta de produtos naturais" (22,6%) e "antimicrobianos" (20,5%), refletindo o predomínio das pesquisas científicas voltadas para a identificação e caracterização de compostos bioativos, Figura 1b. Outros temas de destaque incluíram "descoberta de fármacos", "mineração de genomas" e "clusters de genes biossintéticos", indicando a integração crescente de abordagens genômicas na bioprospecção de novas moléculas. Apesar da importância das técnicas ômicas na biotecnologia moderna, estas representaram apenas 4,27% da frequência total dos termos, sendo "Genômica", "Metagenômica" e "Metabolômica" as ferramentas mais mencionadas. Esta baixa representatividade reforça que, embora as abordagens ômicas estejam em ascensão, a pesquisa em PNs ainda se baseia majoritariamente em metodologias tradicionais, ainda fundamentais, para a descoberta e caracterização de metabólitos. No geral, os resultados evidenciam uma tendência consolidada de combinação entre métodos clássicos e tecnologias emergentes na área de bioprospecção, apontando para oportunidades futuras de integração mais ampla das tecnologias ômicas. Neste contexto, no campo da medicina, as técnicas ômicas permitem a identificação de biomarcadores específicos e o desenvolvimento de terapias direcionadas, otimizando tratamentos conforme a genética individual do paciente.23-25 Na agricultura, a análise ômica possibilita a criação de culturas mais resistentes e nutritivas, através do entendimento profundo dos mecanismos genéticos e metabólicos das plantas.26 Por outro lado, na biologia sintética, as técnicas ômicas são essenciais para a engenharia de microrganismos capazes de produzir novos PNs a partir de templates conhecidos.27 Por fim, a integração dessas técnicas na exploração de PNs abre caminhos para inovações, contribuindo significativamente para a saúde humana, segurança alimentar e sustentabilidade ambiental.28
TÉCNICAS ÔMICAS NO ESTUDO E DESCOBERTA DE PNS As técnicas "ômicas" compõem um campo de estudo das ciências biológicas que está em constante desenvolvimento e expansão. O objetivo dessas técnicas é analisar uma grande quantidade de dados, a fim de identificar, quantificar e caracterizar as moléculas biológicas envolvidas na estrutura, função e dinâmica de células, tecidos ou organismos.17 Em estudos relacionados à Química dos PNs, as técnicas ômicas, como genômica, transcriptômica, proteômica e metabolômica, são importantes para o avanço da compreensão dos processos envolvidos no metabolismo secundário de microrganismos, plantas e animais marinhos, que sintetizam moléculas bioativas.16
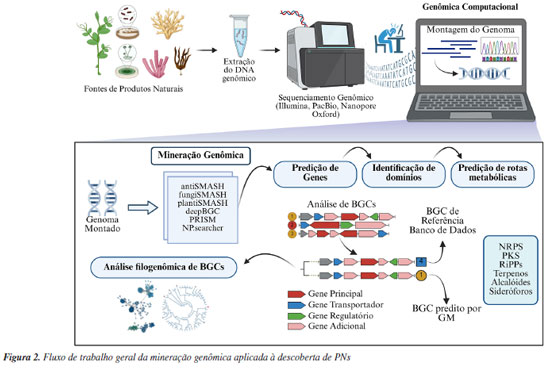
GENÔMICA A genômica é o campo da biologia que estuda e analisa o conjunto de informações que envolvem o DNA de um organismo, incluindo a montagem de genomas completos, a predição de genes, a identificação de domínios formadores de proteínas, funções genéticas e vias metabólicas em que os produtos desses genes estão envolvidos.29 As tecnologias de sequenciamento de nova geração (NGS, do inglês next-generation sequencing),30-32 tais como Illumina®, PacBio® e Oxford Nanopore®, têm revolucionado a investigação de PNs, ao permitir a montagem de genomas de alta qualidade de uma vasta gama de organismos, incluindo plantas e microrganismos, e outras fontes de PNs.33 A técnica mais utilizada para o sequenciamento de genomas de fontes de produtos naturais é a técnica Illumina®. Essa técnica se beneficia nos sequenciamentos genômicos por empregar um alto número de sequências (reads), apresentando tamanhos de 75 a 300 pares de base (pb).31 Enquanto as tecnologias de sequências longas, como PacBio® e Oxford Nanopore® podem obter sequências de montagem maiores que 15.000 pb e 700 pb, respectivamente.30,32 Para realização da mineração genômica (GM, do inglês genome mining), técnica genômica a ser aprofundada no próximo tópico, cada tecnologia de sequenciamento apresenta vantagens e desvantagens. A tecnologia Illumina® é precisa e produz muitos reads a um custo menor, mas pode gerar muitos contigs pequenos, dificultando a montagem de certas regiões.21 Enquanto as tecnologias de longas leituras, como PacBio® e Oxford Nanopore®, oferecem montagens de reads maiores, porém a precisão de sequenciamento ainda é baixa, o que dificulta no procedimento de predição de genes. Para melhorar a montagem e reduzir erros, combinar Illumina® (reads curtos) com PacBio® ou Nanopore® (reads longos) tem se mostrado uma abordagem eficaz.34,35 No genoma dos organismos vivos, encontram-se genes que codificam enzimas fundamentais para diferentes vias metabólicas. Essas vias se dividem em primárias, diretamente ligadas ao crescimento e reprodução, e secundárias, associadas às funções ecológicas dos organismos.33 As vias metabólicas secundárias são as principais responsáveis pela maioria dos PNs conhecidos até hoje.5,26,36,24 Os genes responsáveis por essas vias são frequentemente colocalizados no genoma, formando agrupamentos de genes biossintéticos - BGCs (do inglês, biosynthetic gene clusters). Além dos genes enzimáticos biossintéticos, os BGCs incluem genes que codificam transportadores e reguladores transcricionais.37 Com o surgimento da bioinformática, interligando a computação e a biologia, o campo da genômica, assim como o das demais técnicas ômicas, passou a desempenhar um papel central na conversão de dados biológicos - como sequências de DNA, ácido ribonucleico (RNA), proteomas, e perfis metabólicos - em informações aplicáveis para a descoberta de PNs.38 Dentro desse espectro, a mineração genômica emerge como um importante campo da bioinformática que abrange o desenvolvimento de ferramentas para o processamento e análise de sequências genômicas em busca de genes biossintéticos. Inicialmente, o GM se apoiava em ferramentas como o BLAST (do inglês, basic local alignment search tool) e de bancos de dados de proteínas, como o SwissProt, e o banco de dados de domínios conservados (CDD, do inglês conserved domain database).39-41 Com o tempo, abordagens baseadas no BLAST foram gradualmente substituídas por ferramentas de genômica computacional que utilizam detecção de domínios de proteína através de modelos probabilísticos, como os modelos ocultos de Markov (HMMs, do inglês hidden Markov models). Com isso, entre 2007 e 2012, diversas ferramentas de predição de BGCs baseadas em HMM foram desenvolvidas, a maioria voltada para a anotação putativa e predição de estruturas de sintases de policetídeos do tipo I (T1PKS) e de sintetases de peptídeos não ribossomais (NRPS).42,43 Em 2015, Yamada et al.44 demonstraram que ferramentas baseadas em modelos HMM possibilitaram a expansão do número de sintases de terpeno (TS) nos bancos de dados, de 140 para 262 TSs bacterianas. Em destaque, o antiSMASH, introduzido por Blin et al.,45 é uma plataforma de bioinformática amplamente utilizada para a predição de BGCs em sequências genômicas, possibilitando a anotação e análise abrangente de uma ampla variedade de classes de PNs, como: policetídeos (PKS), peptídeos (NRPS), terpenos, sideróforos, alcaloides, peptídeos sintetizados ribossomalmente e modificados pós-traducionalmente (RiPPs), entre outros. Essa ferramenta genômica realiza a anotação de genes principais da biossíntese de metabólitos secundários, de genes regulatórios, genes ligados à resistência e genes biossintéticos adicionais presentes em BGCs. Ao longo do tempo, outras ferramentas complementares surgiram, como o NPSearcher,46 deepBGC,25 PRISM47 (prediction informatics for secondary metabolomes), BiG-SCAPE/CORASON,27 e o BiG-SLiCE.48 Essas plataformas de GM contam com bancos de dados que reúnem diversas informações sobre BGCs envolvidos na biossíntese de produtos naturais. O MIBiG49 (minimum information about a biosynthetic gene cluster), por exemplo, é um banco de dados de BGCs que está interligado com o antiSMASH, contando com mais de 2.500 BGCs depositados até o ano de 2024. Importante mencionar que o uso das tecnologias de sequenciamento que resultam em sequências longas não são adequadas para o uso na predição de BGCs, uma vez que as ferramentas disponíveis ainda apresentam baixa acurácia quando se trata de sequências desta dimensão.50 A genômica tem sido cada vez mais utilizada na descoberta de PNs. O fluxo geral de trabalho que envolve a mineração de genomas para estudo de PNs segue três etapas principais (Figura 2). A primeira etapa é o sequenciamento do DNA genômico através de técnicas NGS. A segunda etapa é o uso dos dados de sequenciamento genômico para predição de BGCs através das diferentes plataformas de GM, como antiSMASH. Essa etapa é importante para a predição de classes de PNs relacionadas com os BGCs e de estruturas de PNs potencialmente relacionadas aos agrupamentos gênicos, através dos bancos de dados como o MIBiG. A última etapa é a identificação de domínios, funções e rotas metabólicas dos genes contidos nos BGCs analisados. Essa etapa tem como principal função a análise das sequências codificantes de enzimas responsáveis pela biossíntese de PNs, além disso, tem como objetivo agregar informações com relação a motivos proteicos e domínios conservados nos genes de biossíntese principais. A partir disso, é possível prever classes metabólicas e realizar análises filogenômicas dos BGCs identificados. Após as análises genômicas é possível aplicar outras técnicas ômicas, como a proteômica, transcriptômica e metabolômica, para guiar o isolamento e elucidação da estrutura de um novo PN.
Os avanços em GM têm possibilitado cada vez mais que os pesquisadores utilizem genomas de referência para estudos relacionados à descoberta de novos BGCs. Um exemplo, é o uso da mineração genômica para investigar a relação entre a evolução de espécies e a conservação de BGCs em 248 espécies de Burkholderia.51 O gênero de bactérias gram-negativas Burkholderia apresenta em seu genoma um grande número de BGCs que codificam metabólitos secundários, incluindo metabólitos antibacterianos, antitumorais, compostos herbicidas e inseticidas.52 A mineração genômica via antiSMASH permitiu a identificação de vários BGCs conservados entre as espécies de Burkholderia, como os de biossíntese de terpenos, NRPS, entre outros. Além disso, as avaliações de BGCs indicaram que as suas distribuições estão intimamente relacionadas à variação filogenética, permitindo traçar estratégias mais precisas para identificação de novas moléculas medicinais desse gênero.52 As ferramentas baseadas em modelos HMM se tornaram valiosas na identificação de BGCs envolvidos na biossíntese de PNs. No entanto, como qualquer metodologia, essas ferramentas apresentam limitações que podem impactar, por exemplo, na descoberta de BGCs não-canônicos. O principal motivo é que o modelo HMM é treinado com sequências de domínio proteico conhecidas que compõem os BGCs. Ao mesmo tempo, os bancos de dados são limitados e enviesados em torno de microrganismos mais estudados, como actinomicetos, dificultando o treinamento de novos modelos baseados em BGCs não-canônicos.53,54 As isocianidas são uma classe de metabólitos secundários de bactérias e fungos. Esses compostos são produzidos por BGCs não-canônicos, o que dificulta sua detecção por softwares de GM. Nickles et al.55 hipotetizaram que os genes de isocianidas são membros centrais em diversos BGCs fúngicos e apresentaram o primeiro pipeline de GM, montado a partir de ferramentas pré-existentes para identificação de isocianidas fúngicas. Com esse novo pipeline, os pesquisadores identificaram 4.341 genes de isocianidas em 3.300 genomas, representando 1.329 espécies únicas. Portanto, o novo método de GM permitiu a descoberta de genes biossintéticos de isocianidas em uma ampla variedade de fungos. Através da genômica, é possível descobrir diversos PNs, como exemplificado no estudo de Chen et al.56 Neste trabalho, os pesquisadores utilizaram diferentes ferramentas baseadas em GM para a descoberta de derivados de glutarimida. As glutarimidas são antibióticos policetídicos conhecidos amplamente por suas propriedades antitumorais e antifúngicas, produzidos principalmente por organismos do gênero Streptomyces, incluindo lactimidomicina (LTM) e migrastatina (MGS). Através das análises de bioinformática do genoma de Burkholderia gladioli ATCC 10248, um BGC críptico foi identificado. Esse BGC codificava a produção de um policetídeo por uma trans-AT PKS. Este BGC foi então ativado por meio da inserção de um promotor constitutivo, seguido de expressão heteróloga, resultando na produção de sete novos derivados de glutarimida, incluindo cinco compostos inéditos, as gladiofunginas D-H (1-5). O uso de ferramentas de mineração genômica também tem se mostrado importante para o estudo de terpenóides fúngicos. Tang e Matsuda57 desenvolveram uma estratégia baseada em GM global, focada na busca por BGCs que codificam enzimas, cujos domínios não são detectáveis. Estas enzimas, muitas vezes negligenciadas durante as análises de GM, podem desempenhar importantes papéis na biossíntese de triterpenos e outras classes de PNs. Neste estudo, o uso dessa estratégia permitiu a descoberta de onoceroides, triterpenóides inéditos com potencial terapêutico. Além disso, o estudo de BGCs que codificam para terpeno ciclases da família Pyr4 levou à identificação de novos compostos triterpênicos bioativos. Entre os compostos isolados, destacam-se onoceroide A (6), fumionoceroide A (7), fumionoceroide B (8), allionoceroide A (9), e allionoceroide C (10), todos com atividades biológicas significativas. Os pesquisadores também realizaram testes de atividade antitumoral para o composto onoceroide A, fumionoceroide A e fumionoceroide B, que mostraram potencial anticâncer. A expressão heteróloga desses BGCs em Aspergillus oryzae possibilitou a produção e caracterização desses compostos, que poderiam ser explorados como novos agentes terapêuticos no tratamento de doenças como câncer e doenças cardiovasculares.57 Além das análises convencionais de genomas, a mineração de metagenomas emergiu como uma abordagem inovadora na busca por novos PNs, permitindo a descoberta e catalogação de novas fontes desses compostos. Em um estudo realizado por Huang et al.,50 a metagenômica de leitura longa de microrganismos marinhos revelou BGCs crípticos, correspondentes a alguns metabólitos secundários. Neste estudo, os pesquisadores utilizaram metagenomas sequenciados por técnicas de leitura longa. Isso possibilitou a determinação de 339 BGCs envolvidos na biossíntese de PNs e permitiu a catalogação de filos bacterianos não cultivados.50
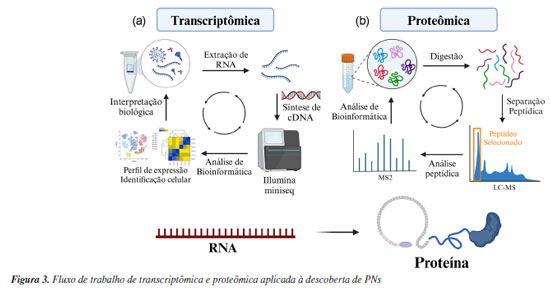
TRANSCRIPTÔMICA E PROTEÔMICA O transcriptoma distingue-se do genoma por sua natureza altamente dinâmica e complexa, refletindo as variações da expressão de genes induzidas por processos de desenvolvimento celular, bem como por influências bióticas e abióticas. Essa variabilidade é acentuada também pelo contexto temporal em que os sinais são registrados, resultando assim em perfis de expressão gênica que podem mudar de maneira significativa durante diferentes condições ambientais e durante diferentes estágios de desenvolvimento.58 Enquanto o genoma de um organismo é estático, o transcriptoma é um retrato variável da expressão gênica, composta não só por mRNA (RNA mensageiro), mas também incluindo microRNAs (miRNAs), RNAs não codificantes longos (lncRNAs) e RNAs circulares (circRNAs). Esses diversos tipos de transcritos desempenham papéis cruciais em numerosos processos biológicos, incluindo a regulação da expressão gênica e a modulação da resposta celular a diferentes estímulos.59 Neste sentido, a análise transcriptômica visa identificar e quantificar as moléculas de RNA transcritas a partir do genoma de um organismo em um determinado momento. Antes da popularização das técnicas de NGS, os microarranjos de RNA (mRNA microarrays) eram utilizados como uma ferramenta experimental fundamental para a construção de transcriptomas de plantas medicinais, com o objetivo de identificar genes responsáveis pela biossíntese de PNs. Além disso, a técnica de microarranjos de RNA é frequentemente empregada em estudos de biossíntese, permitindo a avaliação dos níveis de expressão gênica dos principais genes envolvidos na produção de compostos naturais.60,61 Atualmente, as técnicas transcriptômicas evoluíram substancialmente, destacando-se o sequenciamento de RNA em alta resolução (RNA-seq), que permite a identificação e quantificação detalhada das moléculas de RNA. Além disso, a análise de splicing alternativo e a transcriptômica de célula única (single cell sequencing) oferecem insights sobre a complexidade e a dinâmica da expressão gênica, abrindo novas fronteiras na pesquisa relacionada à biossíntese de PNs.62 Dentro da busca por PNs, o RNA-seq apresenta vantagens em relação aos demais métodos, pois fornece informações mais abrangentes, incluindo a conexão entre éxons, a localização dos limites de transcrição, sítios de splicing alternativo, genes de fusão e variações de sequência.63 Amplificar a capacidade de identificar regiões codificantes (exons) e limites de transcrição significa permitir uma compreensão detalhada da estrutura dos genes principais envolvidos na biossíntese de PNs e seus genes flanqueadores. Em plantas, o estudo de splicing alternativo é útil para identificar a eficiência de tradução, localização subcelular, funções biológicas e/ou interações de proteínas traduzidas.64 Por fim, a identificação de variações de sequência é crucial para entender como a diversidade genética pode influenciar a produção de PNs com diferentes atividades biológicas. Um exemplo é o uso de RNA-seq para montagem de novo de transcriptomas e para análises comparativas de genes biossintéticos entre Physalis alkekengi e Physalis peruavia.65 A análise transcriptômica envolve três etapas principais (Figura 3a). A primeira etapa é a extração do RNA do organismo de interesse. Em seguida, esse RNA é convertido para DNA de fita complementar (cDNA) através da ação de enzimas de transcriptase reversa. Esse DNA é sequenciado através de técnicas NGS e os dados são analisados por ferramentas de bioinformática, como o HISAT2 (hierarchical indexing for spliced alignment of transcripts) (University of Maryland, EUA), Trinity (Broad Institute, EUA), SOAPdenovo-Trans (BGI, China), entre outros.66,67 Essas ferramentas permitem a montagem de novo de transcriptomas, a identificação de isoformas de splicing e regiões transcritas complexas, além de ser útil na identificação de genes diferenciais em determinadas condições, sendo importante na pesquisa de PNs, pois possibilita a identificação dos genes envolvidos na biossíntese de novos compostos bioativos de interesse.
A transcriptômica surgiu como uma ferramenta poderosa no estudo de genes biossintéticos, em particular sendo importante na elucidação de vias biossintéticas de PNs. Essa abordagem já foi aplicada com sucesso em várias plantas, como a Catharanthus roseus e Danshen (Salvia miltiorrhiza).68,69 No primeiro estudo,68 a transcriptômica de C. roseus revelou uma variedade de alcaloides indólicos, como a vincristina (11) e vimblastina (12), amplamente utilizados em terapias contra o câncer. Nos estudos sobre a S. miltirrhiza, a montagem do transcriptoma revelou a presença de importantes classes de PNs, como os diterpenos e os ácidos fenólicos, compostos essenciais para suas propriedades medicinais, que incluem o tratamento de doenças vasculares.69 Os metabólitos secundários (PNs) em plantas e organismos endofíticos desempenham papéis amplamente reconhecidos em resposta a fatores bióticos e abióticos. Nos microrganismos patogênicos, esses compostos assumem uma função crucial na patogenicidade. Nesse contexto, a transcriptômica surge como uma ferramenta valiosa para o estudo de genes biossintéticos potencialmente envolvidos na virulência de patógenos. Um exemplo notável da aplicação da transcriptômica na descoberta de novos PNs é a identificação de meroterpenóides envolvidos na virulência do patógeno Rhynchosporium commune, agente causador da escaldadura da cevada, uma das maiores ameaças à produção global deste grão. Através de análises de expressão diferencial dos genes de R. commune WAI453, tanto in planta quanto in vitro, foi observada a indução da expressão de um cluster gênico biossintético híbrido terpeno-policetídeo (BGC), denominado rhy, que contém o gene de fator de transcrição tipo mieloblastose (rhyM). A superexpressão de rhyM em condições axênicas levou à produção de uma série de novos meroterpenóides, denominados rhynchospenos A-E (13-17). A infiltração de rhynchospenes em folhas de cevada resultou em necrose acentuada, com rhynchospeno B apresentando a maior fitotoxicidade e provocando necrose em uma concentração mínima de 50 ppm. Além disso, o silenciamento do gene rhyM confirmou o papel dos rhynchospenes como fatores de virulência no patossistema.70 A proteômica, por outro lado, é um campo abrangente que estuda as proteínas em detalhe, incluindo suas estruturas, funções, interações e padrões de expressão sob diversas condições biológicas.71 Essa técnica, voltada para o estudo de PNs, busca estudar as enzimas e proteínas resultantes da tradução do mRNA, abrangendo uma ampla gama de organismos, como plantas, bactérias e fungos. Utilizando principalmente tecnologias de espectrometria de massas de alta resolução (HRMS, do inglês high-resolution mass spectrometry), a proteômica permite uma análise detalhada do proteoma - o conjunto total de proteínas produzidas por um organismo, tecido ou célula -, revelando como variações no proteoma podem ocorrer em resposta a estresses abióticos ou bióticos.72 Na busca por novas fontes de PNs, a proteômica é aplicada na identificação e validação de proteínas e enzimas chave que participam de vias biossintéticas secundárias, e pode contribuir no conhecimento de como essas vias são ativadas durante interações e/ou alterações no ambiente biológico.73 Atualmente, as análises de proteômica seguem a metodologia de shotgun MS (botton-up). Neste fluxo de trabalho (Figura 3b), o material proteico secretado pelo organismo (plantas, microrganismos, entre outros) passa por uma digestão proteolítica com tripsina. Os peptídeos são separados, fragmentados e analisados por espectrometria de massas de alta resolução acoplada à cromatografia líquida em tandem (LC-HRMS/MS, do inglês liquid chromatography coupled with high-resolution tandem mass spectrometry). A vantagem desse método de análise proteômica é o uso de equipamentos de espectrometria de massas de alta resolução, como do tipo quadrupolo-tempo de voo (QTOF, do inglês quadrupole time-of-flight) e Orbitrap, auxiliando na interpretação das unidades de aminoácidos que compõem as proteínas. Assim como as demais ômicas, a proteômica gera uma grande quantidade de dados, o que requer ferramentas de bioinformáticas robustas, como o MaxQuant74 (Max Planck Institute of Biochemistry, Alemanha), SWATH-MS,75 BLASTp (proteína-proteína) e UniProt.63,76 Embora as técnicas ômicas de transcriptômica e proteômica sejam amplamente aplicadas em estudos clínicos e de medicina personalizada,25 sua utilização isolada na busca por novos PNs ainda é limitada. Hoje, essas técnicas são utilizadas como ferramentas auxiliares no processo de descoberta de PNs. No entanto, ferramentas de transcriptômica já foram utilizadas no estudo de compostos da classe das tanshinonas por Bielecka et al.77 Estes compostos representam uma classe de pigmentos vermelhos encontrados em Danshen, uma erva medicinal de grande importância na medicina tradicional chinesa. Neste estudo os pesquisadores utilizaram a tecnologia de RNA-seq para montagem de novo de transcriptoma de duas espécies de Salvia perovskia. Com os transcriptomas montados e as análises de bioinformática, pôde-se anotar 134.443 transcritos pelo UniProt, sendo 56.693 atribuídos como pertencentes a plantas verdes (Viridiplantae). Por fim, as análises de enriquecimento de vias pelo KEGG78 (Kyoto Encyclopedia of Genes and Genomes) apontaram duas famílias de genes associados à biossíntese das tanshinonas nas duas espécies estudadas, em específico os genes correspondentes ao citocromo P-450 e a dioxigenases dependentes de 2-oxoglutarato. Com isso, o uso da transcriptômica comparativa gerou um conjunto de genes candidatos para auxiliar na elucidação da biossíntese das tanshinonas, além de demonstrar a diversidade da produção de PNs das espécies de Salvia.77 A transcriptômica também se revela uma ferramenta poderosa na descoberta e no estudo da biossíntese de PNs em plantas, uma vez que, diferentemente dos microrganismos, os genes frequentemente não estão organizados em operons ou agrupamento de genes biossintéticos.79
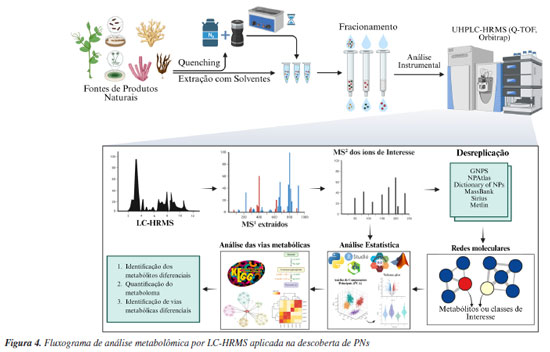
METABOLÔMICA A metabolômica é um campo multidisciplinar que se dedica ao estudo dos metabólitos de baixo peso molecular (< 2000 Da) presentes em sistemas biológicos.80 Entre os metabólitos, incluem-se tanto os primários, essenciais para processos básicos de crescimento e desenvolvimento, como os secundários, que desempenham papéis fundamentais na defesa das plantas e organismos, além de responderem a estímulos bióticos e abióticos.81 O metaboloma de um sistema biológico é vasto e altamente dinâmico, o que torna desafiadora a abordagem integral de seus componentes. Em função disso, a metabolômica utiliza uma variedade de metodologias analíticas, como a cromatografia, a espectrometria de massas, e a ressonância magnética nuclear com foco na descoberta de novas moléculas naturais, identificação de biomarcadores, estudos de quimiotaxonomia e na exploração dos mecanismos de defesa e resistência, especialmente em fontes PNs. A definição e os objetivos da metabolômica podem variar conforme o contexto de aplicação, mas seu principal objetivo continua sendo a caracterização detalhada do perfil metabólico de sistemas biológicos complexos.82-84 As análises metabolômicas podem ser realizadas de forma não direcionada (untargeted) e direcionada (targeted).85 A metabolômica direcionada envolve análises de grupos de metabólitos definidos.86 Já a metabolômica não direcionada fornece uma visão abrangente dos metabólitos presentes em um determinado sistema biológico, sendo a mais utilizada na busca por novos PNs.87,88 Atualmente, a abordagem mais amplamente utilizada e considerada altamente eficaz para a coleta de dados metabolômicos para a descoberta de PNs está associada a equipamentos de cromatografia líquida de ultra eficiência (UHPLC, do inglês ultra-high-performance liquid chromatography) acoplados a sistemas de espectrometria de massas de alta resolução (HRMS, do inglês high-resolution mass spectrometry).89 A LC-HRMS se estabeleceu dada sua eficiência e capacidade de fornecer uma análise detalhada e abrangente de extratos de diferentes fontes de PNs.85,90-94 A Figura 4 ilustra o fluxo de trabalho geral para análises metabolômicas baseada em espectrometria de massas para a busca de novos PNs.95
No fluxo de análises metabolômicas por LC-HRMS/MS, o processo inicia-se com a aquisição dos espectros de MS1. Em seguida, selecionam-se íons precursores alvos para a fragmentação, resultando em espectros de MS2 que refletem a estrutura química dos compostos originais. Esses espectros de fragmentação são então comparados a bibliotecas espectrais para auxiliar na anotação dos metabólitos presentes (Figura 4). Contudo, os dados de MS2 são utilizados principalmente para fins de identificação e, por si só, não fornecem informações quantitativas nem representam a totalidade do perfil químico da amostra. Diante dessas limitações, a espectrometria de massas é amplamente empregada na descoberta e anotação de metabólitos diferenciais e na inferência de vias metabólicas, mas deve ser complementada por outras abordagens para uma caracterização mais abrangente.96 Na busca por PNs, dentro do tratamento de dados metabolômicos, várias abordagens podem ser utilizadas (Figura 4). Uma destas abordagens inclui a anotação metabólica, que envolve a identificação da fórmula molecular, seguida pela pesquisa em bancos de dados de produtos naturais para determinar sua estrutura química através do perfil de fragmentação.97 Nesse âmbito, descobertas como as de Smith et al.,98 permitiram aos pesquisadores utilizarem o primeiro banco de dados de metabolômica analisadas por HRMS, o METLIN.99 Outras plataformas de acesso aberto são muito úteis para a anotação de PNs, como o GNPS (Global Natural Products Social Molecular Networking). A plataforma GNPS foi um grande marco na descoberta de PNs, pois revolucionou a metabolômica ao proporcionar uma plataforma de acesso aberto para o compartilhamento de dados de MS. Com o MassIVE como banco de dados principal, o GNPS consolidou-se como uma ferramenta essencial para a pesquisa de produtos naturais, promovendo colaboração e avanço no campo da metabolômica.100-102 Na análise metabolômica, a identificação de metabólitos é classificada em diferentes níveis de confiança, conforme sugerido pelas diretrizes do Metabolomics Standards Initiative (MSI).103 O nível 1 refere-se à identificação confirmada por padrão analítico comercial disponível, usando dados de tR (tempo de retenção), espectro de massa e, idealmente, espectro de fragmentação. O nível 2 é a anotação putativa baseada em similaridade espectral com bancos de dados de referência, sem validação com padrão químico. O nível 3 agrupa compostos em classes químicas (como flavonoides, terpenóides e etc.) sem identificação individual de metabólitos. O nível 4 refere-se a sinais desconhecidos, onde a presença do metabólito é detectada, mas não há informações suficientes para sugerir sua identidade ou classe.80 Essa estratificação é crucial para interpretar os dados com rigor e transparência na descoberta de novos compostos bioativos.95 Um exemplo do sucesso na aplicação da desreplicação em dados de LC-HRMS para a descoberta de PNs é a identificação de análogos das levesquamidas por LeClair et al.100 As levesquamidas A (18) e B (19) são PNs com atividade antituberculose que contêm o grupo isotiazolinona e sua biossíntese é realizada através de um mecanismo não colinear misto envolvendo NRPS e PKS.100 Através das análises de LC-MS/MS e a criação de redes moleculares pelo GNPS, os pesquisadores conseguiram identificar análogos das levesquamidas. Essas análises guiaram os pesquisadores para a realização de cultivos em larga escala da Streptomyces sp. RKND-216 com o objetivo de extrair e purificar os análogos da levesquamida A, o que resultou no isolamento de duas novas levesquamidas (C e D).100 Outra técnica baseada em MS amplamente utilizada na descoberta de PNs é a técnica de imageamento por espectrometria de massas (MSI, do inglês mass spectrometry imaging). Na busca por PNs, as técnicas de MSI, como o imageamento por ionização e dessorção a laser assistida por matriz (MALDI), apresentam vantagens com relação às técnicas tradicionais de análise, pois reduzem efetivamente o tempo de análise, utilização de solventes e fornecem informações sobre a distribuição espacial dos metabólitos secundários em plantas, microrganismos e outras fontes naturais.104 No entanto, o preparo de amostras para imageamento por MALDI é um processo complexo e demorado, sendo uma desvantagem da técnica. A técnica de MALDI-MSI é particularmente útil em análises de metabolômica não direcionada. Esta técnica consolidou-se como uma das técnicas ex vivo mais úteis na caracterização espacial de moléculas, permitindo a visualização da distribuição de metabólitos em diferentes tecidos, tornando-se uma ferramenta de ponta para pesquisadores biomédicos em estudos farmacocinéticos e até mesmo em estudos de metabolômica subcelular.105 Kuo et al.106 utilizaram uma estratégia integrada de MALDI-MSI guiado por redes moleculares no GNPS, com o objetivo de identificar e caracterizar os produtos naturais (PNs) presentes na madeira de ágar (agarwood), especificamente da espécie Aquilaria sinensis. A madeira de ágar ou "madeira Oud", é proveniente de árvores de floresta do nordeste das Índia, Butão e sudeste asiático e muito conhecida por sua resina. As análises das secções da madeira de ágar de Aquilaria sinensis possibilitaram a identificação inédita de vários metabólitos secundários, com a caracterização de três análogos de 2-(2-feniletenil)cromonas (20) através da associação entre MSI e montagem de redes moleculares.107 Além da desreplicação, facilitada por plataformas como o GNPS, a análise de espectros de fragmentação in silico pode expandir a busca por novos PNs.84 O SIRIUS108,109 (Friedrich Schiller Universität Jena, Bright Giant GmbH, Alemanha) é uma ferramenta que aprimora significativamente a identificação de fórmulas moleculares putativas através do CSI:FingerID, um método de aprendizado de máquina, que utiliza vetores de suporte, para comparar os padrões de fragmentação obtidos em espectros MS/MS com uma vasta base de dados de espectros preditos para estruturas químicas conhecidas. Assim, ao invés de buscar por correspondências exatas em bases de dados de MS/MS, o CSI:FingerID identifica os componentes estruturais mais prováveis que compõem a molécula desconhecida, e em seguida os busca em bancos de dados de estruturas químicas, permitindo uma identificação mais abrangente e detalhada.90-92 Esta ferramenta está revolucionando a previsão de estruturas por aprendizado de máquinas na busca por PNs. Um exemplo disso é o estudo de Lai et al.,110 que apontou uma estratégia de visualização de dados de MS utilizando o MCnebula111 (Cao-lab, School of Pharmacy, Zhejiang Chinese Medical University, China) integrado ao SIRIUS para avaliação metabólica de extratos de Plantaginis semen (PS). O P. semen é comumente utilizado na medicina tradicional chinesa para o tratamento de doenças renais. A combinação SIRIUS-MCnebula anotou putativamente oitenta e nove compostos nos extratos de P. semen, como isoacteosídeos, calceolariosídeos, 2'-acetilacteosídeos e plantainosídeos. Essa abordagem fornece uma nova estratégia de visualização de dados de MS através de ferramentas computacionais acopladas que utilizam aprendizado de máquina em sua programação.112 As análises estatísticas também são de grande importância nas análises metabolômicas por LC-HRMS visando a busca de novos PNs. O MetaboAnalyst, lançado em 2009 por Xia e colaboradores,113 é um software gratuito baseado em análises abrangentes de dados de metabolômica, incluindo análises estatísticas, identificação de metabólitos e mapeamento de vias metabólicas. Essa ferramenta, assim como o XCMS114 (Scripps Research Institute, EUA), MS-DIAL115 (Riken, Japão), entre outros, são estratégias utilizadas na busca por PNs diferenciais em interações planta-inseto, vírus-bactéria, fungo-planta, ou ainda organismo-meio ambiente que envolvem a produção de PNs.116 Análises univariadas e multivariadas associadas a estudos de redes moleculares permitiram a identificação de compostos inéditos produzidos durante a co-cultura entre 37 actinomicetos e Pyrrhoderma noxium. O P. noxium é um patógeno vegetal que induz a podridão e perda das raízes em várias espécies de árvores. Adra et al.,117 através dos experimentos de co-cultura, conseguiram identificar 15 actinomicetos isolados que apresentaram alta atividade antifúngica contra P. noxium. Entre os metabólitos anotados pode-se citar: as isoflavonas daidzeína (21) e genisteína (22), alguns antibióticos macrotetrolídeos como nonactina (23), monactina (24), dinactina (25), trinactina (26), bonactina (27), tetranactina (28), oligomicina A (29); as cumarinas 6-metilcumarina (30) e metoxsaleno (31), além de peptídeos como desferrioxamina E (32) e a dehidroxinocardamina (33). As análises de enriquecimento de vias, feitas no MetaboAnalyst, apontaram que as vias diferenciais estavam relacionadas à produção de policetídeos, classe já relacionada à atividade antifúngica dos actinomicetos analisados.117 Uma importante técnica para estudos metabolômicos de compostos de baixa polaridade é a cromatografia gasosa acoplada à espectrometria de massas de alta resolução (GC-HRMS, do inglês gas chromatography coupled with high-resolution mass spectrometry). Essa ferramenta combina a capacidade de separação da GC com a precisão da HRMS, permitindo a detecção e identificação de compostos voláteis e semi-voláteis, além de abranger uma gama de metabólitos que muitas vezes não são possíveis de serem analisados por LC-HRMS.118 Diferentes estratégias são utilizadas no preparo de amostras para análises via GC-MS, incluindo a microextração em fase sólida (SPME, do inglês solid phase microextraction), que reduz o uso de solventes orgânicos e o custo em relação às análises de LC-HRMS.119,120 Sachito e Oliveira,121 assim como Barbosa,122 utilizaram dados gerados por GC-MS para exploração do terpenoma de 3 diferentes cepas de Streptomyces (Streptomyces sp. CBMAI 2042, CBMAI 2043 e S. cyaneus) através de ferramentas de redes moleculares fornecidas pelo GNPS. Esses estudos forneceram informações da produção de sesquiterpenóides como cariofilenol (34), albaflavenol (35) e albaflavenona (36) com potencial atividade antimicrobiana, além de investigar a produção dos respectivos metabólitos sob diferentes condições de crescimento. Além disso, os pesquisadores utilizaram as ferramentas de GM e de criação de redes moleculares para auxiliarem na relação metabólito-gene.121,122 Embora a GC-HRMS seja altamente eficiente para a análise de compostos voláteis e semi-voláteis, a LC-HRMS se torna essencial para a análise de compostos de maior polaridade ou em matrizes mais complexas, ampliando o espectro de metabólitos analisados. A complexidade dos metabólitos - destacando-se a sua ampla diversidade estrutural e a variabilidade na sua concentração - ainda representa um desafio considerável para alcançar uma análise do metaboloma tão abrangente quanto as já estabelecidas para técnicas envolvendo estudos genômicos, transcriptômicos e proteômicos. Neste contexto, as técnicas de ressonância magnética nuclear (NMR, do inglês nuclear magnetic ressonance) podem conter informações preciosas sobre os compostos orgânicos, como a estrutura molecular, ambiente eletrônico, interações moleculares e isomeria.123 Apesar da técnica de NMR ser inerentemente menos sensível do que a MS, deve-se notar que hoje, equipamentos de NMR de alta resolução equipados com sondas criogênicas de baixo volume permitem a gravação de espectros de NMR ricos em informações com quantidades até a faixa de baixo μg. A propagação de tais informações estruturais estabelecidas, principalmente por meio de redes moleculares, permite uma melhor anotação de muitos análogos estruturais tipicamente presentes nos extratos naturais. Além disso, o desenvolvimento de ferramentas baseadas em NMR e aprendizado de máquina, como o SMART 2.1124 (small molecule accurate recognition technology), faz com que o uso dessa técnica seja cada vez mais utilizada em estudos metabolômicos visando a descoberta de PNs. Lee et al.125 utilizaram a metabolômica por NMR associada ao SMART 2.0 para o isolamento direcionado de lactonas sesquiterpênicas em Eupatorium fortunei, uma planta da família das Asteraceae amplamente distribuída no sudeste asiático e utilizada como medicamento tradicional para o tratamento de resfriados, calafrios e febre. Neste estudo, os pesquisadores realizaram aquisições de NMR 1D e 2D em 600 MHz de extratos dessa planta, e utilizaram os dados obtidos na ferramenta SMART 2.0 para desreplicação e identificação dos compostos presentes, tanto no extrato bruto, quanto nas frações obtidas. Por fim, foram isoladas dez β-lactonas sesquiterpênicas derivadas do germacradienol, entre as quais três compostos, 8β-[4',5'-dihidroxitigloiloxi]-costunolídeo (37), 1β-hidroxi-8β-[4'-hidroxitigloiloxi]-β-ciclocostunolídeo (38) e 2β-hidroxi-8β-[5'-hidroxitigloiloxi]-costunolídeo (39), apresentaram atividade contra células de câncer de próstata e de mama.126 Devido à alta complexidade dos extratos naturais, torna-se cada vez mais necessário o uso de ferramentas analíticas de alta resolução capazes de detectar e caracterizar compostos desconhecidos, aliadas às metodologias clássicas.90 Neste contexto, a mobilidade iônica (IM, do inglês ion mobility) destaca-se como uma tecnologia poderosa, permitindo a separação de íons em fase gasosa com base em propriedades físico-químicas específicas, como tamanho, forma tridimensional e razão massa-carga (m/z).127 A IM permite a diferenciação de moléculas isobáricas e isômeras estruturais que, devido às suas semelhanças em massa e estrutura, poderiam ser indistinguíveis por espectrometria de massas de alta resolução, como a Orbitrap, aumentando significativamente a capacidade de detectar novas entidades químicas em misturas complexas.127 Além da separação, a IM permite a determinação do diâmetro de colisão (collision cross section, CCS), que funciona como uma impressão digital adicional para a identificação de compostos.128-131 Diversos estudos têm demonstrado o valor da IM na descoberta de PNs. Por exemplo, Marshall et al.132 utilizaram a espectrometria de massas com mobilidade iônica para guiar o isolamento e identificação da estreptorubina B (40) em Streptomycescoelicolor M145. A IM foi crucial para diferenciar as estruturas isobáricas da estreptorubina B (40) e da butilcicloheptilprodigiosina, permitindo que a identidade da estreptorubina B (40) fosse confirmada de forma precisa.132
INTEGRAÇÃO DAS TÉCNICAS ÔMICAS, DESAFIOS E PESPECTIVAS FUTURAS NA DESCOBERTA DE PNS Atualmente a descoberta direcionada de compostos bioativos enfrenta desafios significativos.133 Tecnologias ômicas, como genômica e metabolômica, fornecem dados valiosos para a identificação de genes biossintéticos de interesse e a previsão de estruturas de compostos, por se tratarem de técnicas com grandes bancos de dados disponíveis.130 Já o uso isolado da proteômica e transcriptômica na busca por novos PNs tem se mostrado limitado devido à complexidade inerente às interações entre proteínas e genes, bem como às restrições técnicas relacionadas à identificação e quantificação precisas dessas biomoléculas.134 Além disso, o volume reduzido de dados disponíveis em bancos específicos para essas técnicas, em comparação com outras abordagens ômicas, impõe desafios adicionais à sua aplicação de forma independente. Essas limitações refletem não apenas as barreiras analíticas, mas também a necessidade de ampliar a integração entre diferentes camadas de dados biológicos para elucidar sistemas moleculares complexos. A integração de dados multidimensionais obtidos por diferentes plataformas ômicas (incluindo genômica, transcriptômica, proteômica e metabolômica) tem o potencial de oferecer uma visão mais abrangente dos mecanismos moleculares e pode auxiliar estudos biossintéticos de PNs.17 Esta abordagem integrada busca melhorar a eficiência na mineração de novas estruturas químicas e potenciais atividades biológicas.135 Contudo, a complexidade inerente à combinação de dados ômicos é considerável. As estratégias atuais baseadas na análise integrada de dados multiômicos para a descoberta de PNs ainda não são consideradas totalmente maduras, sendo uma oportunidade para futuros estudos.126 As ferramentas de bioinformática existentes, embora úteis para tarefas específicas como a mineração de genomas (por exemplo, antiSMASH), podem ter limitações na integração abrangente de diferentes dados ômicos e grande volume de dados.17 Nesse sentido, o estudo de novas vias biossintéticas de PNs demanda tempo devido à identificação incompleta de genes/enzimas funcionais, intermediários desconhecidos e vias biossintéticas pouco depositadas nos diferentes bancos de dados.136 Nesse contexto, a integração de técnicas ômicas torna-se indispensável para superar essas limitações e avançar na descoberta de PNs. Abordagens integrativas possibilitam a exploração de interações entre diferentes camadas biológicas, permitindo uma análise mais abrangente dos sistemas biológicos envolvidos. Há na literatura126,137,138 trabalhos que exploram a integração de metodologias e ferramentas avançadas, demonstrando sua aplicação prática. Além disso, ferramentas como NPLinker,139 MetaMiner,140 xMWAS,141 Kleisli142 e CORNET,143 destacam-se pela sua capacidade de integrar, manipular e analisar dados ômicos de maneira eficiente, aprimorando a compreensão dos mecanismos biológicos e facilitando a descoberta de novos produtos naturais. Estas ferramentas de integração ômica desempenham um papel crucial na análise e compreensão dos complexos dados biológicos gerados por diferentes plataformas experimentais.144 O MetaMiner140 é uma ferramenta computacional especializada que integra dados de espectrometria de massas (MS) com informações genômicas/metagenômicas, com o objetivo de identificar e priorizar novos produtos naturais peptídicos, especialmente os derivados de vias biossintéticas como peptídeos não ribossomais. Essa ferramenta tem sido amplamente usada em contextos ecológicos e biomédicos para explorar microbiomas e metagenomas, onde a descoberta de peptídeos bioativos pode ter implicações farmacológicas relevantes. Outra ferramenta relevante é o xMWAS0,141 que permite a integração de dados de diferentes plataformas ômicas, como transcriptômica e metabolômica, para estudar interações moleculares complexas e entender a fisiopatologia de doenças. O Kleisli,142 por sua vez, é um sistema de integração de dados em larga escala baseado em programação funcional, permitindo a manipulação e transformação de grandes volumes de dados. Já o CORNET143 é especializado em acessar dados transcriptômicos e de interação proteína-proteína de Arabidopsis thaliana, facilitando a análise de coexpressão e interações. A integração de dados metabolômicos e genômicos, conhecida como metabologenômica, possui um grande potencial para identificar BGCs, e correlaciona-los ao seus respectivos PNs. Essa abordagem permite uma exploração mais abrangente das vias metabólicas e seus reguladores, facilitando a descoberta de compostos bioativos e seus mecanismos moleculares.63 A plataforma de Pareamento de Dados Ômicos (Paired Omics Data Platform)145 é um exemplo de plataforma criada pela comunidade científica e tem como iniciativa integrar a metabologenômica na descoberta de PNs. A importância dessa plataforma de integração está na utilização de informações metabolômicas armazenadas em bancos de dados públicos como MassIVE ou MetaboLights146 para vincular a genomas públicos depositados em bancos de dados como Centro Nacional de Informação Biotecnológica (NCBI, do inglês National Library of Medicine)147 e/ou Instituto Conjunto do Genoma (JGI, do inglês Joint Genome Institute).148 Essa integração permite correlações em larga escala de alterações no metaboloma, facilitando novas descobertas e o desenvolvimento de algoritmos para prever estruturas químicas a partir de informações genômicas e metabolômicas. Um exemplo do sucesso da integração de dados de genômica e metabolômica foi a descoberta das liciumidas A (41), B (42), C (43) e D (44) e como sua biossíntese é acoplada a domínios BURPs. Esses domínios, que são conservados em várias plantas, possuem cerca de 230 aminoácidos e estão localizados nas regiões C-terminais de determinadas proteínas. Os domínios BURP estão relacionados à regulação de processos metabólicos e ao desenvolvimento de plantas, desempenhando um papel fundamental na biossíntese de compostos secundários, como as liciumidas (Figura 5).149 As liciumidas são peptídeos vegetais sintetizados ribossomicamente e modificados pós-tradução. Esse trabalho aponta a importância dos estudos de RiPPs em plantas, dado que a maioria dos metabólitos descobertos dessa classe provém de bactérias e fungos. Kersten e Weng149 identificaram genes precursores da liciumida A através de análises genômicas de nove genomas diferentes de plantas. Através das ferramentas de mineração genômica foi possível identificar domínios BURPs conservados, correspondentes à produção das liciumidas A, B e D nas plantas produtoras desses metabólitos. Esses genes foram então clonados de forma heteróloga. Essa estratégia ilustra o uso da mineração genômica associada a estratégias automatizáveis para caracterização de peptídeos ribossomais cíclicos ramificados pertencentes à classe das licuiminas.149 Outras ferramentas de destaque na integração de dados genômicos são NPLinker e MetaMiner. Essas plataformas integradoras foram desenvolvidas para correlacionar dados genômicos e metabolômicos, com ênfase na vinculação entre BGCs microbianos e metabólitos secundários. Essa ferramenta combina informações do genoma (predição de BGCs pelo antiSMASH/BiG-SCAPE) e do metaboloma (espectros de MS2 agrupados em famílias moleculares).
Devido aos avanços tecnológicos, a metabolômica por NMR vem sendo utilizada cada vez mais na busca por novos PNs. A integração entre a metabolômica por NMR e a mineração de genomas pode fornecer importantes informações para a identificação de características estruturais, assim como informações biossintéticas para direcionar a busca de novos PNs. Huang et al.123 utilizaram a metabolômica via NMR e a GM da Streptomyces sp. (CGMCC 14582) para a mineração de lenziamidas, peptídeos N-metilados. Neste estudo, a mineração de genoma da cepa apontou a presença de seis BGCs correspondentes à biossíntese de NRPS desconhecidos, entre eles, um responsável pela biossíntese de lenziamidas. As análises do BGC ainda apontaram a presença de genes modificadores (lenE e lenF), além de motivos e domínios que codificam ácido piperázico. A integração dessas técnicas ômicas, contribuiu na orientação para o isolamento de dois decapeptídeos cíclicos, lenziamidas D1 (45) e B1 (46) que apresentam um resíduo não proteinogênico de ácido piperázico (L-Piz). A metabologenômica também pode ser aplicada no isolamento de novas moléculas de interesse biológico em ensaios de bioatividade, conforme descrito no estudo de Ayon et al.150 Este estudo descreve um workflow integrando dados metabolômicos com seus respectivos agrupamentos biossintéticos em 110 fungos. Além disso, frações obtidas a partir dos extratos desses microrganismos foram avaliadas por LC-HRMS/MS e com relação ao potencial anticâncer. Através da bioquimiometria, foi realizada a integração das atividades biológicas dos extratos e frações bioativas com os dados de espectrometria de massas. Com isso, três novos análogos de temphonas foram isolados neste estudo: 19-acetylstephona B (47), G (48) e E (49). Esses compostos foram isolados de fungos do gênero Aspergillus (Aspergillus biplanus, A. diversus e A. conjunctus), cujos extratos demonstraram atividades antiproliferativas contra células humanas de melanoma (MDA-MB-435) e câncer de ovário (OVCAR3).150 A combinação de análises transcriptômicas e análises metabolômicas pode ser utilizada com sucesso na exploração de vias metabólicas em plantas medicinais, como no caso do estudo com as plantas Fagopyrum cymosum109 e Astragalus mongholicus.110 Além disso, Chen et al.151 utilizaram a integração transcriptômica- metabolômica para o estudo da biossíntese de sesquiterpenóides e poliacetilenos produzidos por Atractylodes lancea, uma erva nativa do Vietnã que é utilizada como medicamento fitoterápico para diversas doenças. A integração do perfil metabólico com os dados do transcriptoma de A. lancea permitiu a identificação de genes de expressão diferenciais associados à biossíntese de β-eudesmola (50), atractilona (51), e da atractilodina (52), se tornando o primeiro estudo a explorar essas vias metabólicas em A. lancea. Paulo et al.152 integraram dados genômicos e metabolômicos para a descoberta das megapolipeptinas A (53) e B (54), metabólitos híbridos com uma parte policetídica, peptídica não ribossomal e uma parte lipídica poli-insaturada. O BGC desses compostos, previamente silenciado em condições padrão de cultivo, foi identificado e expresso de maneira heteróloga em Burkholderia sp. FERM BP-3421. As megapolipeptinas representam uma classe de lipopeptídeos com uma estrutura que combina grupos hidrofílicos em suas extremidades e um centro hidrofóbico. Sua biossíntese foi associada a um BGC com 14 genes, que codificam enzimas responsáveis pela construção modular desses dois compostos. A integração de dados transcriptômicos e metabolômicos foi importante para Mai et al.153 na descoberta de um novo alcaloide indólico monoterpênico em Tabernaemontana litoralis. Neste estudo, foram identificados 3 compostos majoritários, sendo o 11-hidroxipseudovincadiformina (55) o alcaloide indólico monoterpênico majoritário nas folhas de T. litoralis. A pesquisa também realizou a expressão heteróloga de três enzimas chave para a produção desse PN; as monooxigenases de citocromo P450 (CYPs), atuando na oxidação estereoespecífica do esqueleto desses monoterpenos. Esses resultados enfatizam o potencial de T. litoralis como uma fonte rica para a exploração de produtos naturais bioativos. Neste sentido, a integração de dados metabolômicos e transcriptômicos possibilitou desvendar vias biossintéticas previamente inexploradas, incluindo a da 11-hidroxipseudovincadiformina.153 Com o desenvolvimento das novas tecnologias ômicas associadas a ferramentas computacionais, novos meios de acessar a diversidade de PNs vem sendo revelados. Paralelamente, as novas abordagens de inteligência artificial (IA) e aprendizado de máquinas estão impulsionando o design de novos medicamentos baseados em PNs, auxiliando a predição de atividades biológicas e ensaios in silico para alvos moleculares de interesse. Nestas ferramentas, dados de genômica, proteômica, transcriptômica, metabolômica, de bioatividade experimentais, entre outros, são integrados e interpretados por IAs.154-156 Manochkumar e Ramamoorthy154 apontam que, até o final de 2023, mais de quarenta ferramentas foram desenvolvidas para a mineração sistemática de PNs. As principais aplicações incluem a integração de ômicas para screening virtual de estruturas e ligantes, desenvolvimento de novas drogas a partir de PNs, predição de propriedades físico-químicas e bioatividade, desreplicação de dados de MS/MS e NMR, identificação de BGCs em metagenomas, entre outras aplicações. Com o sequenciamento do genoma de microrganismos se tornando cada vez mais acessível, a previsão de rotas biossintéticas envolvidas na produção de PNs vem aumentando exponencialmente. Os avanços das técnicas de engenharia genética e de bioprocessos, possibilitaram modificar geneticamente uma ampla gama de organismos, incluindo fontes produtoras de PNs. Com isso, faz-se possível a manipulação da produção de PNs através do conhecimento dos BGCs relacionados à sua via biossintética, o que, por sua vez, pode facilitar o seu isolamento, caracterização e geração de análogos bioativos.136,157 Recentemente, Bradley et al.158 utilizaram leveduras modificadas através de técnicas de biologia sintética para a biossíntese natural de um derivado de alcaloide indólico monoterpênico de plantas. Wilson et al.159 utilizaram sistemas de expressão heteróloga, fundamentado por um sequenciamento metagenômico e transcriptômico de esponjas para expressão de grupos de TSs do tipo I de várias esponjas, para descoberta de novos sesquiterpenóides nitrogenados. Por fim, cada vez mais, ferramentas de bioinformática baseadas em ML e IA vêm sendo desenvolvidas e desempenham um papel revolucionário na descoberta de PNs. Ferramentas baseadas em ML, como GECCO160 (gene cluster prediction with conditionalrandom fields) e SanntiS,161 superam as limitações das ferramentas baseadas em regras (como o antiSMASH) na descoberta de BGCs, sendo capazes de detectar mais BGCs ou, crucialmente, mais BGCs novos e não agrupados, e demonstraram melhor desempenho de precisão em conjuntos de dados comuns. Arquiteturas de redes neuronais têm sido usadas para genome mining e na elucidação de vias biossintéticas, através da construção de modelos baseados em algoritmos de ML. A análise abrangente de múltiplos dados ômicos de plantas medicinais é vista como um método eficaz para promover a precisão da previsão da função genética e das vias metabólica.162
CONCLUSÕES A incorporação das técnicas ômicas na pesquisa de PNs representa um grande avanço, permitindo uma compreensão mais profunda e integrada das vias biossintéticas, potenciais alvos terapêuticos, além de direcionar o isolamento de compostos inéditos bioativos. No entanto, a aplicação isolada dessas abordagens apresenta limitações, evidenciando a necessidade de estratégias multiômicas para uma análise mais abrangente e precisa. A integração de dados provenientes de genômica, transcriptômica, proteômica e metabolômica oferece uma visão holística das interações moleculares envolvidas na biossíntese de PNs. Essa abordagem integrada não apenas aumenta a confiabilidade na identificação de novos compostos bioativos, mas também impulsiona o desenvolvimento de estratégias de engenharia metabólica e de síntese de compostos bioativos, potencializando a inovação em áreas como farmacologia, química e biotecnologia. Contudo, a implementação eficaz de estratégias multiômicas enfrenta desafios significativos, tanto computacionais quanto analíticos. A complexidade e a heterogeneidade dos dados exigem o desenvolvimento de ferramentas bioinformáticas avançadas e algoritmos robustos para a integração e interpretação dos dados. Além disso, a padronização das metodologias ômicas e multiômicas é crucial para garantir a reprodutibilidade e comparabilidade dos resultados, facilitando a aplicação de aprendizado de máquina e inteligência artificial na análise dos dados. Futuras pesquisas devem focar no aprimoramento das técnicas de integração de dados ômicos, no desenvolvimento de protocolos padronizados e na validação experimental das descobertas. Além disso, é essencial a colaboração interdisciplinar entre pesquisadores de diferentes áreas para superar os desafios técnicos e conceituais, promovendo avanços significativos na descoberta e aplicação de PNs. Essa abordagem integrativa promete acelerar a inovação no campo da farmacologia e biotecnologia, ampliando as possibilidades terapêuticas derivadas de produtos naturais e contribuindo para o avanço da ciência e da saúde global.
DECLARAÇÃO DE DISPONIBILIDADE DE DADOS Todos os dados estão disponíveis no texto.
AGRADECIMENTOS Agradecemos ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), à Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES) e à Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP) (processos No. 2021/08947-3, 2022/02992-0, 2023/06874-4, 2022/02872-4 e 2019/17721-9) pelo apoio financeiro. REFERÊNCIAS 1. Ji, H.; Li, X.; Zhang, H.; EMBO Rep. 2009, 10, 194. [Crossref] 2. Yun, B. W.; Yan, Z.; Amir, R.; Hong, S.; Jin, Y. W.; Lee, E. K.; Loake, G. J.; Biotechnol. Genet. Eng. Rev. 2012, 28, 47. [Crossref] 3. Borges, W. D. S.; Berlinck, R. G. S.; Scotti, M. T.; Vieira, P. C.; Quim. Nova 2017, 40, 706. [Crossref] 4. Grigalunas, M.; Brakmann, S.; Waldmann, H.; J. Am. Chem. Soc. 2022, 144, 3314. [Crossref] 5. Abdel-Razek, A. S.; El-Naggar, M. E.; Allam, A.; Morsy, O. M.; Othman, S. I.; Processes 2020, 8, 470. [Crossref] 6. Pupo, M. T.; Gallo, M. B. C.; Vieira, P. C.; Quim. Nova 2007, 30, 1446. [Crossref] 7. Medina-Franco, J. L. Em Evidence-Based Validation of Herbal Medicine; Mukherjee, P. K., ed.; Elsevier: Amsterdam, 2015, p. 455. [Crossref] 8. Newman, D. J.; Cragg, G. M.; J. Nat. Prod. 2020, 83, 770. [Crossref] 9. Montanari, C. A.; Bolzani, V. D. S.; Quim. Nova 2001, 24, 105. [Crossref] 10. Nourbakhsh, F.; Lotfalizadeh, M.; Badpeyma, M.; Shakeri, A.; Soheili, V.; Phytother. Res. 2022, 36, 33. [Crossref] 11. Abraham, E. P.; Biogr. Mem. Fellows R. Soc. 1983, 29, 42. [Crossref] 12. Lalchhandama, K.; WikiJournal of Medicine 2021, 8, 1. [Link] acessado em julho 2025 13. Tu, Y.; Angew. Chem., Int. Ed. 2016, 55, 10210. [Crossref] 14. Chen, H.; Guo, M.; Dong, S.; Wu, X.; Zhang, G.; He, L.; Jiao, Y.; Chen, S.; Li, L.; Luo, H.; Plant Commun. 2023, 4, 100516. [Crossref] 15. Eiben, C. B.; de Rond, T.; Bloszies, C.; Gin, J.; Chiniquy, J.; Baidoo, E. E. K.; Petzold, C. J.; Hillson, N. J.; Fiehn, O.; Keasling, J. D.; ACS Synth. Biol. 2019, 8, 2238. [Crossref] 16. Aborode, A. T.; Awuah, W. A.; Mikhailova, T.; Abdul-Rahman, T.; Pavlock, S.; Kundu, M.; Yarlagadda, R.; Pustake, M.; Correia, I. F. D. S.; Mehmood, Q.; Shah, P.; Mehta, A.; Ahmad, S.; Asekun, A.; Nansubuga, E. P.; Amaka, S. O.; Shkodina, A. D.; Alexiou, A.; Curr. Top. Med. Chem. 2022, 22, 1751. [Crossref] 17. Palazzotto, E.; Weber, T.; Curr. Opin. Microbiol. 2018, 45, 109. [Crossref] 18. Gemperline, E.; Keller, C.; Li, L.; Anal. Chem. 2016, 88, 3422. [Crossref] 19. Caesar, L. K.; Montaser, R.; Keller, N. P.; Kelleher, N. L.; Nat. Prod. Rep. 2021, 38, 2041. [Crossref] 20. Tolani, P.; Gupta, S.; Yadav, K.; Aggarwal, S.; Yadav, A. K.; Adv. Protein Chem. Struct. Biol. 2021, 127, 127. [Crossref] 21. Dewey, F. E.; Pan, S.; Wheeler, M. T.; Quake, S. R.; Ashley, E. A.; Circulation 2012, 125, 931. [Crossref] 22. Dai, X.; Shen, L.; Front. Med. 2022, 9, 911861. [Crossref] 23. Roca, I.; Cojocaru, E.; Rusu, C.; Trandafir, L.; Săveanu, C.; Lupu, V.; Butnariu, L.; Ţarcă, V.; Moscalu, M.; Bernic, J.; Lupu, A.; Ţarcă, E.; Pediatr. Health, Med. Ther. 2024, 15, 1. [Crossref] 24. Huang, X.; Lowrie, D. B.; Fan, X. Y.; Hu, Z.; Biomed. Pharmacother. 2024, 171, 116087. [Crossref] 25. Castelli, F. A.; Rosati, G.; Moguet, C.; Fuentes, C.; Marrugo-Ramírez, J.; Lefebvre, T.; Volland, H.; Merkoçi, A.; Simon, S.; Fenaille, F.; Junot, C.; Anal. Bioanal. Chem. 2022, 414, 759. [Crossref] 26. Mishra, P.; Tripathi, A.; Dikshit, A.; Pandey, A. Em Natural Bioactive Products in Sustainable Agriculture; Singh, J.; Yadav, A. N., eds.; Springer: Singapore, 2020, cap. 6. [Crossref] 27. Navarro-Muñoz, J. C.; Selem-Mojica, N.; Mullowney, M. W.; Kautsar, S. A.; Tryon, J. H.; Parkinson, E. I.; De Los Santos, E. L. C.; Yeong, M.; Cruz-Morales, P.; Abubucker, S.; Roeters, A.; Lokhorst, W.; Fernandez-Guerra, A.; Cappelini, L. T. D.; Goering, A. W.; Thomson, R. J.; Metcalf, W. W.; Kelleher, N. L.; Barona-Gomez, F.; Medema, M. H.; Nat. Chem. Biol. 2020, 16, 60. [Crossref] 28. Lopes, N. P.; Vieira, P. C.; ACS Omega 2017, 2, 8794. [Crossref] 29. Ametaj, B. N. Em Periparturient Diseases of Dairy Cows; Ametaj, B. N., ed.; Springer International Publishing: Cham, 2017, cap. 2. [Crossref] 30. Rhoads, A.; Au, K. F.; Genomics, Proteomics Bioinf. 2015, 13, 278. [Crossref] 31. Steemers, F. J.; Gunderson, K. L.; Pharmacogenomics 2005, 6, 777. [Crossref] 32. Wang, Y.; Zhao, Y.; Bollas, A.; Wang, Y.; Au, K. F.; Nat. Biotechnol. 2021, 39, 1348. [Crossref] 33. Caesar, L. K.; Butun, F. A.; Robey, M. T.; Ayon, N. J.; Gupta, R.; Dainko, D.; Bok, J. W.; Nickles, G.; Stankey, R. J.; Johnson, D.; Mead, D.; Cank, K. B.; Earp, C. E.; Raja, H. A.; Oberlies, N. H.; Keller, N. P.; Kelleher, N. L.; Nat. Chem. Biol. 2023, 19, 846. [Crossref] 34. Ziemert, N.; Weber, T.; Medema, M. H. Em Comprehensive Natural Products III; Lui, H. W.; Begley, T. P., eds.; Elsevier: San Diego, 2020, p. 19. 35. Albarano, L.; Esposito, R.; Ruocco, N.; Costantini, M.; Mar. Drugs 2020, 18, 199. [Crossref] 36. Bologa, C. G.; Ursu, O.; Oprea, T. I.; Melançon, C. E.; Tegos, G. P.; Curr. Opin. Pharmacol. 2013, 13, 678. [Crossref] 37. Estrada, R.; Corredor, F. A.; Figueroa, D.; Salazar, W.; Quilcate, C.; Vásquez, H. V.; Maicelo, J. L.; Gonzales, J.; Arbizu, C. I.; Data 2022, 7, 155. [Crossref] 38. Van Santen, J. A.; Kautsar, S. A.; Medema, M. H.; Linington, R. G.; Nat. Prod. Rep. 2021, 38, 264. [Crossref] 39. Sayers, E. W.; Bolton, E. E.; Brister, J. R.; Canese, K.; Chan, J.; Comeau, D. C.; Connor, R.; Funk, K.; Kelly, C.; Kim, S.; Madej, T.; Marchler-Bauer, A.; Lanczycki, C.; Lathrop, S.; Lu, Z.; Thibaud-Nissen, F.; Murphy, T.; Phan, L.; Skripchenko, Y.; Tse, T.; Wang, J.; Williams, R.; Trawick, B. W.; Pruitt, K. D.; Sherry, S. T.; Nucleic Acids Res. 2022, 50, D20. [Crossref] 40. Camacho, C.; Boratyn, G. M.; Joukov, V.; Alvarez, R. V.; Madden, T. L.; BMC Bioinf. 2023, 24, 117. [Crossref] 41. Coudert, E.; Gehant, S.; de Castro, E.; Pozzato, M.; Baratin, D.; Neto, T.; Sigrist, C. J. A.; Redaschi, N.; Bridge, A.; The UniProt Consortium; Bioinformatics 2023, 39, btac793. [Crossref] 42. Hage, H.; Couillaud, J.; Salamov, A.; Loussouarn-Yvon, M.; Durbesson, F.; Ormeño, E.; Grisel, S.; Duquesne, K.; Vincentelli, R.; Grigoriev, I.; Iacazio, G.; Rosso, M. N.; Microb. Genomics 2023, 9, 1. [Crossref] 43. Yoon, B. J.; Curr. Genomics 2009, 10, 402. [Crossref] 44. Yamada, Y.; Kuzuyama, T.; Komatsu, M.; Shin-ya, K.; Omura, S.; Cane, D. E.; Ikeda, H.; Proc. Natl. Acad. Sci. U. S. A. 2015, 112, 857. [Crossref] 45. Blin, K.; Shaw, S.; Kloosterman, A. M.; Charlop-Powers, Z.; van Wezel, G. P.; Medema, M. H.; Weber, T.; Nucleic Acids Res. 2021, 49, W29. [Crossref] 46. Lee, S.; Van Santen, J. A.; Farzaneh, N.; Liu, D. Y.; Pye, C. R.; Baumeister, T. U. H.; Wong, W. R.; Linington, R. G.; ACS Cent. Sci. 2022, 8, 223. [Crossref] 47. Skinnider, M. A.; Johnston, C. W.; Gunabalasingam, M.; Merwin, N. J.; Kieliszek, A. M.; MacLellan, R. J.; Li, H.; Ranieri, M. R. M.; Webster, A. L. H.; Cao, M. P. T.; Pfeifle, A.; Spencer, N.; To, Q. H.; Wallace, D. P.; Dejong, C. A.; Magarvey, N. A.; Nat. Commun. 2020, 11, 6058. [Crossref] 48. Kautsar, S. A.; van der Hooft, J. J. J.; de Ridder, D.; Medema, M. H.; GigaScience 2021, 10, giaa154. [Crossref] 49. Zdouc, M. M.; Blin, K.; Louwen, N. L. L.; Navarro, J.; Loureiro, C.; Bader, C. D.; Bailey, C. B.; Barra, L.; Booth, T. J.; Bozhüyük, K. A. J.; Cediel-Becerra, J. D. D.; Charlop-Powers, Z.; Chevrette, M. G.; Chooi, Y. H.; D'Agostino, P. M.; de Rond, T.; Del Pup, E.; Duncan, K. R.; Gu, W.; Hanif, N.; Helfrich, E. J. N.; Jenner, M.; Katsuyama, Y.; Korenskaia, A.; Krug, D.; Libis, V.; Lund, G. A.; Mantri, S.; Morgan, K. D.; Owen, C.; Phan, C. S.; Philmus, B.; Reitz, Z. L.; Robinson, S. L.; Singh, K. S.; Teufel, R.; Tong, Y.; Tugizimana, F.; Ulanova, D.; Winter, J. M.; Aguilar, C.; Akiyama, D. Y.; Al-Salihi, S. A. A.; Alanjary, M.; Alberti, F.; Aleti, G.; Alharthi, S. A.; Rojo, M. Y. A.; Arishi, A. A.; Augustijn, H. E.; Avalon, N. E.; Avelar-Rivas, J. A.; Axt, K. K.; Barbieri, H. B.; Barbosa, J. C. J.; Segato, L. G. B.; Barrett, S. E.; Baunach, M.; Beemelmanns, C.; Beqaj, D.; Berger, T.; Bernaldo-Agüero, J.; Bettenbühl, S. M.; Bielinski, V. A.; Biermann, F.; Borges, R. M.; Borriss, R.; Breitenbach, M.; Bretscher, K. M.; Brigham, M. W.; Buedenbender, L.; Bulcock, B. W.; Cano-Prieto, C.; Capela, J.; Carrion, V. J.; Carter, R. S.; Castelo-Branco, R.; Castro-Falcón, G.; Chagas, F. O.; Charria-Girón, E.; Chaudhri, A. A.; Chaudhry, V.; Choi, H.; Choi, Y.; Choupannejad, R.; Chromy, J.; Donahey, M. S. C.; Collemare, J.; Connolly, J. A.; Creamer, K. E.; Crüsemann, M.; Cruz, A. A.; Cumsille, A.; Dallery, J. F.; Damas-Ramos, L. C.; Damiani, T.; de Kruijff, M.; Martín, B. D.; Sala, G. D.; Dillen, J.; Doering, D. T.; Dommaraju, S. R.; Durusu, S.; Egbert, S.; Ellerhorst, M.; Faussurier, B.; Fetter, A.; Feuermann, M.; Fewer, D. P.; Foldi, J.; Frediansyah, A.; Garza, E. A.; Gavriilidou, A.; Gentile, A.; Gerke, J.; Gerstmans, H.; Gomez-Escribano, J. P.; González-Salazar, L. A.; Grayson, N. E.; Greco, C.; Gomez, J. E. G.; Guerra, S.; Flores, S. G.; Gurevich, A.; Gutiérrez-García, K.; Hart, L.; Haslinger, K.; He, B.; Hebra, T.; Hemmann, J. L.; Hindra, H.; Höing, L.; Holland, D. C.; Holme, J. E.; Horch, T.; Hrab, P.; Hu, J.; Huynh, T. H.; Hwang, J. Y.; Iacovelli, R.; Iftime, D.; Iorio, M.; Jayachandran, S.; Jeong, E.; Jing, J.; Jung, J. J.; Kakumu, Y.; Kalkreuter, E.; Kang, K. B.; Kang, S.; Kim, W.; Kim, G. J.; Kim, H.; Kim, H. U.; Klapper, M.; Koetsier, R. A.; Kollten, C.; Kovács, A. T.; Kriukova, Y.; Kubach, N.; Kunjapur, A. M.; Kushnareva, A. K.; Kust, A.; Lamber, J.; Larralde, M.; Larsen, N. J.; Launay, A. P.; Le, N. T. H.; Lebeer, S.; Lee, B. T.; Lee, K.; Lev, K. L.; Li, S. M.; Li, Y. X.; Licona-Cassani, C.; Lien, A.; Liu, J.; Lopez, J. A. V.; Machushynets, N. V.; Macias, M. I.; Mahmud, T.; Maleckis, M.; Martinez-Martinez, A. M.; Mast, Y.; Maximo, M. F.; McBride, C. M.; McLellan, R. M.; Bhatt, K. M.; Melkonian, C.; Merrild, A.; Metsä-Ketelä, M.; Mitchell, D. A.; Müller, A. V.; Nguyen, G. S.; Nguyen, H. T.; Niedermeyer, T. H. J.; O'Hare, J. H.; Ossowicki, A.; Ostash, B. O.; Otani, H.; Padva, L.; Paliyal, S.; Pan, X.; Panghal, M.; Parade, D. S.; Park, J.; Parra, J.; Rubio, M. P.; Pham, H. T.; Pidot, S. J.; Piel, J.; Pourmohsenin, B.; Rakhmanov, M.; Ramesh, S.; Rasmussen, M. H.; Rego, A.; Reher, R.; Rice, A. J.; Rigolet, A.; Romero-Otero, A.; Rosas-Becerra, L. R.; Rosiles, P. Y.; Rutz, A.; Ryu, B.; Sahadeo, L. A.; Saldanha, M.; Salvi, L.; Sánchez-Carvajal, E.; Santos-Medellin, C.; Sbaraini, N.; Schoellhorn, S. M.; Schumm, C.; Sehnal, L.; Selem, N.; Shah, A. D.; Shishido, T. K.; Sieber, S.; Silviani, V.; Singh, G.; Singh, H.; Sokolova, N.; Sonnenschein, E. C.; Sosio, M.; Sowa, S. T.; Steffen, K.; Stegmann, E.; Streiff, A. B.; Strüder, A.; Surup, F.; Svenningsen, T.; Sweeney, D.; Szenei, J.; Tagirdzhanov, A.; Tan, B.; Tarnowski, M. J.; Terlouw, B. R.; Rey, T.; Thome, N. U.; Torres Ortega, L. R.; Tørring, T.; Trindade, M.; Truman, A. W.; Tvilum, M.; Udwary, D. W.; Ulbricht, C.; Vader, L.; van Wezel, G. P.; Walmsley, M.; Warnasinghe, R.; Weddeling, H. G.; Weir, A. N. M.; Williams, K.; Williams, S. E.; Witte, T. E.; Rocca, S. M. W.; Yamada, K.; Yang, D.; Yang, D.; Yu, J.; Zhou, Z.; Ziemert, N.; Zimmer, L.; Zimmermann, A.; Zimmermann, C.; van der Hooft, J. J. J.; Linington, R. G.; Weber, T.; Medema, M. H.; Nucleic Acids Res. 2025, 53, D678. [Crossref] 50. Huang, R.; Wang, Y.; Liu, D.; Wang, S.; Lv, H.; Yan, Z.; Microbiol. Spectrum 2023, 11, e01501. [Crossref] 51. Alam, K.; Islam, M. M.; Gong, K.; Abbasi, M. N.; Li, R.; Zhang, Y.; Li, A.; Comput. Biol. Med. 2022, 140, 105046. [Crossref] 52. Bach, E.; Passaglia, L. M. P.; Jiao, J.; Gross, H.; Crit. Rev. Microbiol. 2022, 48, 121. [Crossref] 53. Helfrich, E. J. N.; Ueoka, R.; Dolev, A.; Rust, M.; Meoded, R. A.; Bhushan, A.; Califano, G.; Costa, R.; Gugger, M.; Steinbeck, C.; Moreno, P.; Piel, J.; Nat. Chem. Biol. 2019, 15, 813. [Crossref] 54. Ziemert, N.; Alanjary, M.; Weber, T.; Nat. Prod. Rep. 2016, 33, 988. [Crossref] 55. Nickles, G. R.; Oestereicher, B.; Keller, N. P.; Drott, M. T.; Nucleic Acids Res. 2023, 51, 7220. [Crossref] 56. Chen, H.; Bai, X.; Sun, T.; Wang, X.; Zhang, Y.; Bian, X.; Zhou, H.; Molecules 2023, 28, 6937. [Crossref] 57. Tang, J.; Matsuda, Y.; Nat. Commun. 2024, 15, 4312. [Crossref] 58. Batista, A. N. L.; Santos-Pinto, J. R. A. D.; Batista, J. M.; Souza-Moreira, T. M.; Santoni, M. M.; Zanelli, C. F.; Kato, M. J.; López, S. N.; Palma, M. S.; Furlan, M.; J. Nat. Prod. 2017, 80, 1275. [Crossref] 59. Hoover, C. A.; Slattery, M.; Marsh, A. G.; Mar. Biotechnol. 2007, 9, 411. [Crossref] 60. Boik, J.; Kirakosyan, A.; Kaufman, P. B.; Seymour, E. M.; Spelman, K. Em Recent Advances in Plant Biotechnology; Kirakosyan, A.; Kaufman, P. B., eds.; Springer: Boston, 2009, cap. 10. [Crossref] 61. Schmitz, K.; Haggarty, S. J.; McPherson, O. M.; Clardy, J.; Koehler, A. N.; J. Am. Chem. Soc. 2007, 129, 11346. [Crossref] 62. Amos, G. C. A.; Awakawa, T.; Tuttle, R. N.; Letzel, A. C.; Kim, M. C.; Kudo, Y.; Fenical, W.; Moore, B. S.; Jensen, P. R.; Proc. Natl. Acad. Sci.U. S. A. 2017, 114. [Crossref] 63. Adua, E.; J. Hum. Hypertens. 2022, 37, 253. [Crossref] 64. Lam, P. Y.; Wang, L.; Lo, C.; Zhu, F. Y.; Int. J. Mol. Sci. 2022, 23, 7355. [Crossref] 65. Fukushima, A.; Nakamura, M.; Suzuki, H.; Yamazaki, M.; Knoch, E.; Mori, T.; Umemoto, N.; Morita, M.; Hirai, G.; Sodeoka, M.; Saito, K.; Front. Plant Sci. 2016, 7, 1883. [Crossref] 66. Haas, B. J.; Papanicolaou, A.; Yassour, M.; Grabherr, M.; Blood, P. D.; Bowden, J.; Couger, M. B.; Eccles, D.; Li, B.; Lieber, M.; MacManes, M. D.; Ott, M.; Orvis, J.; Pochet, N.; Strozzi, F.; Weeks, N.; Westerman, R.; William, T.; Dewey, C. N.; Henschel, R.; LeDuc, R. D.; Friedman, N.; Regev, A.; Nat. Protoc. 2013, 8, 1494. [Crossref] 67. Xie, Y.; Wu, G.; Tang, J.; Luo, R.; Patterson, J.; Liu, S.; Huang, W.; He, G.; Gu, S.; Li, S.; Zhou, X.; Lam, T. W.; Li, Y.; Xu, X.; Wong, G. K. S.; Wang, J.; Bioinformatics 2014, 30, 1660. [Crossref] 68. Verma, M.; Ghangal, R.; Sharma, R.; Sinha, A. K.; Jain, M.; PLoS One 2014, 9, e103583. [Crossref] 69. Guo J.; Ma, Y. Em The Salvia miltiorrhiza Genome; Lu, S., ed.; Springer International Publishing: Cham, 2019, p. 129. [Crossref] 70. Darma, R.; Shang, Z.; Bracegirdle, J.; Moggach, S.; McDonald, M. C.; Piggott, A. M.; Solomon, P. S.; Chooi, Y. H.; ACS Chem. Biol. 2025, 20, 421. [Crossref] 71. Muroi, M.; Osada, H.; TheJournal of Antibiotics 2021, 74, 639. [Crossref] 72. Chavda, V.; Dange, K.; Joglekar, M. Em Bioinformatics Tools for Pharmaceutical Drug Product Development; Chavda, V.; Anand, K.; Apostolopoulos, V., eds.; Wiley: Weinheim, 2023, p. 77. 73. Bumpus, S. B.; Evans, B. S.; Thomas, P. M.; Ntai, I.; Kelleher, N. L.; Nat. Biotechnol. 2009, 27, 951. [Crossref] 74. Bielow, C.; Mastrobuoni, G.; Kempa, S.; J. Proteome Res. 2016, 15, 777. [Crossref] 75. Ludwig, C.; Gillet, L.; Rosenberger, G.; Amon, S.; Collins, B. C.; Aebersold, R.; Mol. Syst. Biol. 2018, 14, e8126. [Crossref] 76. UniProt, https://www.uniprot.org/, acessado em julho 2025. 77. Bielecka, M.; Stafiniak, M.; Pencakowski, B.; Ślusarczyk, S.; Jastrzębski, J. P.; Paukszto, Ł.; Łaczmański, Ł.; Gharibi, S.; Matkowski, A.; Sci. Rep. 2024, 14, 3046. [Crossref] 78. Kyoto Encyclopedia of Genes and Genomes (KEGG), https://www.genome.jp/kegg/, acessado em julho 2025. 79. Liu, S.; Li, J.; Wu, Y.; Ren, Y.; Liu, Q.; Wang, Q.; Zhou, X.; Cai, M.; Zhang, Y.; Genes Genomics 2017, 39, 317. [Crossref] 80. Silva, E.; Dantas, R.; Barbosa, J. C.; Berlinck, R. G. S.; Fill, T.; Mol. Omics 2024, 20, 154. [Crossref] 81. Hong, J.; Yang, L.; Zhang, D.; Shi, J.; Int. J. Mol. Sci. 2016, 17, 767. [Crossref] 82. Medeiros, L. S.; de Araújo Junior, M. B.; Peres, E. G.; da Silva, J. C. I.; Bassicheto, M. C.; Di Gioia, G.; Veiga, T. A. M.; Koolen, H. H. F. Em Microbial Natural Products Chemistry: A Metabolomics Approach; Fill, T. P., ed.; Springer International Publishing: Cham, 2023, cap. 8. [Crossref] 83. Theodoridis, G.; Gika, H.; Franceschi, P.; Caputi, L.; Arapitsas, P.; Scholz, M.; Masuero, D.; Wehrens, R.; Vrhovsek, U.; Mattivi, F.; Metabolomics 2012, 8, 175. [Crossref] 84. Wolfender, J. L.; Nuzillard, J. M.; Van Der Hooft, J. J. J.; Renault, J. H.; Bertrand, S.; Anal. Chem. 2019, 91, 704. [Crossref] 85. Bauermeister, A.; Mannochio-Russo, H.; Costa-Lotufo, L. V.; Jarmusch, A. K.; Dorrestein, P. C.; Nat. Rev. Microbiol. 2022, 20, 143. [Crossref] 86. Cajka, T.; Fiehn, O.; Anal. Chem. 2016, 88, 524. [Crossref] 87. Greño, M.; Plaza, M.; Marina, M. L.; Puyana, M. C.; Food Chem. 2023, 402, 134209. [Crossref] 88. Maimone, N. M.; K. Pereira, A.; Gimenes, L.; Fernandes, H. P.; Fill, T. P.; Borges, R. M.; Lira, S. P.; Fernandes, J. B.; Bauermeister, A.; Rev. Virtual Quim. 2024, 16, 248. [Crossref] 89. Alseekh, S.; Aharoni, A.; Brotman, Y.; Contrepois, K.; D'Auria, J.; Ewald, J.; C. Ewald, J.; Fraser, P. D.; Giavalisco, P.; Hall, R. D.; Heinemann, M.; Link, H.; Luo, J.; Neumann, S.; Nielsen, J.; de Souza, L. P.; Saito, K.; Sauer, U.; Schroeder, F. C.; Schuster, S.; Siuzdak, G.; Skirycz, A.; Sumner, L. W.; Snyder, M. P.; Tang, H.; Tohge, T.; Wang, Y.; Wen, W.; Wu, S.; Xu, G.; Zamboni, N.; Fernie, A. R.; Nat. Methods 2021, 18, 747. [Crossref] 90. Alvarez-Rivera, G.; Ballesteros-Vivas, D.; Parada-Alfonso, F.; Ibañez, E.; Cifuentes, A.; TrAC, Trends Anal. Chem. 2019, 112, 87. [Crossref] 91. Bouslimani, A.; Sanchez, L. M.; Garg, N.; Dorrestein, P. C.; Nat. Prod. Rep. 2014, 31, 718. [Crossref] 92. Vu, H.; Pham, N. B.; Quinn, R. J.; SLAS Discovery 2008, 13, 265. [Crossref] 93. Wang, H.; de Carvalho, L. P. S.; Curr. Opin. Chem. Biol. 2023, 74, 102287. [Crossref] 94. Belinato, J.; Bazioli, J.; Sussulini, A.; Augusto, F.; Fill, T. P.; Quim. Nova 2019, 42, 546. [Crossref] 95. Covington, B. C.; McLean, J. A.; Bachmann, B. O.; Nat. Prod. Rep. 2017, 34, 6. [Crossref] 96. Salem, M. A.; de Souza, L. P.; Serag, A.; Fernie, A. R.; Farag, M. A.; Ezzat, S. M.; Alseekh, S.; Metabolites 2020, 10, 37. [Crossref] 97. Nielsen, K. F.; Larsen, T. O.; Front. Microbiol. 2015, 6. [Crossref] 98. Smith, C. A.; Maille, G. O.; Want, E. J.; Qin, C.; Trauger, S. A.; Brandon, T. R.; Custodio, D. E.; Abagyan, R.; Siuzdak, G.; Ther. Drug Monit. 2005, 27, 747. [Crossref] 99. METLIN, https://metlin.scripps.edu/auth-login.html, acessado em julho 2025. 100. LeClair, M. M.; Maw, Z. A.; Grunwald, A. L.; Kelly, J. R.; Haltli, B. A.; Kerr, R. G.; Cartmell, C.; Molecules 2022, 27, 22 [Crossref]; GNPS, https://gnps.ucsd.edu/ProteoSAFe/static/gnps-splash.jsp?redirect=auth, acessado em julho 2025. 101. Wang, M.; Carver, J. J.; Phelan, V. V.; Sanchez, L. M.; Garg, N.; Peng, Y.; Nguyen, D. D.; Watrous, J.; Kapono, C. A.; Luzzatto-Knaan, T.; Porto, C.; Bouslimani, A.; Melnik, A. V.; Meehan, M. J.; Liu, W. T.; Crüsemann, M.; Boudreau, P. D.; Esquenazi, E.; Sandoval-Calderón, M.; Kersten, R. D.; Pace, L. A.; Quinn, R. A.; Duncan, K. R.; Hsu, C. C.; Floros, D. J.; Gavilan, R. G.; Kleigrewe, K.; Northen, T.; Dutton, R. J.; Parrot, D.; Carlson, E. E.; Aigle, B.; Michelsen, C. F.; Jelsbak, L.; Sohlenkamp, C.; Pevzner, P.; Edlund, A.; McLean, J.; Piel, J.; Murphy, B. T.; Gerwick, L.; Liaw, C. C.; Yang, Y. L.; Humpf, H. U.; Maansson, M.; Keyzers, R. A.; Sims, A. C.; Johnson, A. R.; Sidebottom, A. M.; Sedio, B. E.; Klitgaard, A.; Larson, C. B.; Boya, P. C. A.; Torres-Mendoza, D.; Gonzalez, D. J.; Silva, D. B.; Marques, L. M.; Demarque, D. P.; Pociute, E.; O'Neill, E. C.; Briand, E.; Helfrich, E. J. N.; Granatosky, E. A.; Glukhov, E.; Ryffel, F.; Houson, H.; Mohimani, H.; Kharbush, J. J.; Zeng, Y.; Vorholt, J. A.; Kurita, K. L.; Charusanti, P.; McPhail, K. L.; Nielsen, K. F.; Vuong, L.; Elfeki, M.; Traxler, M. F.; Engene, N.; Koyama, N.; Vining, O. B.; Baric, R.; Silva, R. R.; Mascuch, S. J.; Tomasi, S.; Jenkins, S.; Macherla, V.; Hoffman, T.; Agarwal, V.; Williams, P. G.; Dai, J.; Neupane, R.; Gurr, J.; Rodríguez, A. M. C.; Lamsa, A.; Zhang, C.; Dorrestein, K.; Duggan, B. M.; Almaliti, J.; Allard, P. M.; Phapale, P.; Nothias, L. F.; Alexandrov, T.; Litaudon, M.; Wolfender, J. L.; Kyle, J. E.; Metz, T. O.; Peryea, T.; Nguyen, D. T.; VanLeer, D.; Shinn, P.; Jadhav, A.; Müller, R.; Waters, K. M.; Shi, W.; Liu, X.; Zhang, L.; Knight, R.; Jensen, P. R.; Palsson, B. Ø.; Pogliano, K.; Linington, R. G.; Gutiérrez, M.; Lopes, N. P.; Gerwick, W. H.; Moore, B. S.; Dorrestein, P. C.; Bandeira, N.; Nat. Biotechnol. 2016, 34, 828. [Crossref] 102. Zuffa, S.; Schmid, R.; Bauermeister, A.; Gomes, P. W.; Caraballo-Rodriguez, A. M.; El Abiead, Y.; Aron, A. T.; Gentry, E. C.; Zemlin, J.; Meehan, M. J.; Avalon, N. E.; Cichewicz, R. H.; Buzun, E.; Terrazas, M. C.; Hsu, C. Y.; Oles, R.; Ayala, A. V.; Zhao, J.; Chu, H.; Kuijpers, M. C. M.; Jackrel, S. L.; Tugizimana, F.; Nephali, L. P.; Dubery, I. A.; Madala, N. E.; Moreira, E. A.; Costa-Lotufo, L. V.; Lopes, N. P.; Rezende-Teixeira, P.; Jimenez, P. C.; Rimal, B.; Patterson, A. D.; Traxler, M. F.; Pessotti, R. D. C.; Alvarado-Villalobos, D.; Tamayo-Castillo, G.; Chaverri, P.; Escudero-Leyva, E.; Quiros-Guerrero, L. M.; Bory, A. J.; Joubert, J.; Rutz, A.; Wolfender, J. L.; Allard, P. M.; Sichert, A.; Pontrelli, S.; Pullman, B. S.; Bandeira, N.; Gerwick, W. H.; Gindro, K.; Massana-Codina, J.; Wagner, B. C.; Forchhammer, K.; Petras, D.; Aiosa, N.; Garg, N.; Liebeke, M.; Bourceau, P.; Kang, K. B.; Gadhavi, H.; de Carvalho, L. P. S.; dos Santos, M. S.; Pérez-Lorente, A. I.; Molina-Santiago, C.; Romero, D.; Franke, R.; Brönstrup, M.; De León, A. V. P.; Pope, P. B.; La Rosa, S. L.; La Barbera, G.; Roager, H. M.; Laursen, M. F.; Hammerle, F.; Siewert, B.; Peintner, U.; Licona-Cassani, C.; Rodriguez-Orduña, L.; Rampler, E.; Hildebrand, F.; Koellensperger, G.; Schoeny, H.; Hohenwallner, K.; Panzenboeck, L.; Gregor, R.; O'Neill, E. C.; Roxborough, E. T.; Odoi, J.; Bale, N. J.; Ding, S.; Damsté, J. S. S.; Guan, X. L.; Cui, J. J.; Ju, K. S.; Silva, D. B.; Silva, F. M. R.; da Silva, G. F.; Koolen, H. H. F.; Grundmann, C.; Clement, J. A.; Mohimani, H.; Broders, K.; McPhail, K. L.; Ober-Singleton, S. E.; Rath, C. M.; McDonald, D.; Knight, R.; Wang, M.; Dorrestein, P. C.; Nat. Microbiol. 2024, 9, 336. [Crossref] 103. Spicer, R. A.; Salek, R.; Steinbeck, C.; Sci. Data 2017, 4, 170138. [Crossref] 104. Costa, M. S.; Clark, C. M.; Ómarsdóttir, S.; Sanchez, L. M.; Murphy, B. T.; J. Nat. Prod. 2019, 82, 2167. [Crossref] 105. Tuck, M.; Grélard, F.; Blanc, L.; Desbenoit, N.; Front. Chem. 2022, 10, 904688. [Crossref] 106. Kuo, T. H.; Huang, H. C.; Hsu, C. C.; Anal. Chim. Acta 2019, 1080, 95. [Crossref] 107. Heinonen, M.; Shen, H.; Zamboni, N.; Rousu, J.; Bioinformatics 2012, 28, 2333. [Crossref] 108. Hoffmann, M. A.; Nothias, L. F.; Ludwig, M.; Fleischauer, M.; Gentry, E. C.; Witting, M.; Dorrestein, P. C.; Dührkop, K.; Böcker, S.; Nat. Biotechnol. 2022, 40, 411. [Crossref] 109. Ludwig, M.; Dührkop, K.; Böcker, S.; Bioinformatics 2018, 34, i333. [Crossref] 110. Lai, J.; Huang, L.; Bao, Y.; Wang, L.; Lyu, Q.; Kuang, H.; Wang, K.; Sang, X.; Yang, Q.; Shan, Q.; Cao, G.; Analyst 2022, 147, 4739. [Crossref] 111. Huang, L.; Shan, Q.; Lyu, Q.; Zhang, S.; Wang, L.; Cao, G.; Anal. Chem. 2023, 95, 9940. [Crossref] 112. Aghdam, S. A.; Brown, A. M. V.; Environ. Microbiome 2021, 16, 6. [Crossref] 113. Pang, Z.; Zhou, G.; Ewald, J.; Chang, L.; Hacariz, O.; Basu, N.; Xia, J.; Nat. Protoc. 2022, 17, 1735. [Crossref] 114. Tautenhahn, R.; Patti, G. J.; Rinehart, D.; Siuzdak, G.; Anal. Chem. 2012, 84, 5035. [Crossref] 115. Tsugawa, H.; Cajka, T.; Kind, T.; Ma, Y.; Higgins, B.; Ikeda, K.; Kanazawa, M.; VanderGheynst, J.; Fiehn, O.; Arita, M.; Nat. Methods 2015, 12, 523. [Crossref] 116. Peng, X. Y.; Wu, J. T.; Shao, C. L.; Li, Z. Y.; Chen, M.; Wang, C. Y.; Mar. Life Sci. Technol. 2021, 3, 363. [Crossref] 117. Adra, C.; Tran, T. D.; Foster, K.; Tomlin, R.; Kurtböke, D. İ.; Antibiotics 2023, 12, 1373. [Crossref] 118. Aziz, M.; Ahmad, S.; Khurshid, U.; Pervaiz, I.; Lodhi, A. H.; Jan, N.; Khurshid, S.; Arshad, M. A.; Ibrahim, M. M.; Mersal, G. A. M.; Alenazi, F. S.; Alamri, A. A. S.; Butt, J.; Saleem, H.; El-Bahy, Z. M.; Molecules 2022, 27, 779. [Crossref] 119. D'Auria, M.; Emanuele, L.; Lorenz, R.; Mecca, M.; Racioppi, R.; Romano, V. A.; Viggiani, L.; Nat. Prod. Res. 2023, 38, 2511. [Crossref] 120. Lei, H.; Yue, J.; Yin, X. Y.; Fan, W.; Tan, S. H.; Qin, L.; Zhao, Y. N.; Bai, J. H.; Phytochem. Anal. 2023, 34, 317. [Crossref] 121. Sachito, D.; de Oliveira, L.; J. Braz. Chem. Soc. 2022, 33, 734. [Crossref] 122. Barbosa, J. C. J.: Exploração do Terpenoma da Streptomyces sp. CBMAI 2043 e Streptomyces cyaneus A21; Dissertação de Mestrado, Universidade Estadual de Campinas, Campinas, Brasil. [Link] acessado em julho 2025 123. Huang, H.; Yue, L.; Deng, F.; Wang, X.; Wang, N.; Chen, H.; Li, H.; J. Nat. Prod. 2023, 86, 2122. [Crossref] 124. Reher, R.; Kim, H. W.; Zhang, C.; Mao, H. H.; Wang, M.; Nothias, L. F.; Caraballo-Rodriguez, A. M.; Glukhov, E.; Teke, B.; Leao, T.; Alexander, K. L.; Duggan, B. M.; Van Everbroeck, E. L.; Dorrestein, P. C.; Cottrell, G. W.; Gerwick, W. H.; J. Am. Chem. Soc. 2020, 142, 4114. [Crossref] 125. Lee, J.; Park, J.; Kim, J.; Jeong, B.; Choi, S. Y.; Jang, H. S.; Yang, H.; ACS Omega 2020, 5, 23989. [Crossref] 126. Liu, Q. R.; Zhang, X. J.; Zheng, L.; Meng, L. J.; Liu, G. Q.; Yang, T.; Lu, Z. M.; Chai, L. J.; Wang, S. T.; Shi, J. S.; Shen, C. H.; Xu, Z. H.; Food Res. Int. 2023, 167, 112594. [Crossref] 127. Carnevale Neto, F.; Clark, T. N.; Lopes, N. P.; Linington, R. G.; J. Nat. Prod. 2022, 85, 519. [Crossref] 128. Otvos, R. A.; Still, K. B. M.; Somsen, G. W.; Smit, A. B.; Kool, J.; SLAS Discovery 2019, 24, 362. [Crossref] 129. Wang, Y.; Zhu, G.; Wang, M.; Wu, J.; Fu, D.; Xie, Q.; Shi, Q.; Xu, C.; Han, Y.; Chem. Eng. Sci. 2023, 273, 118677. [Crossref] 130. Gaudêncio, S. P.; Bayram, E.; Bilela, L. L.; Cueto, M.; Díaz-Marrero, A. R.; Haznedaroglu, B. Z.; Jimenez, C.; Mandalakis, M.; Pereira, F.; Reyes, F.; Tasdemir, D.; Mar. Drugs 2023, 21, 308. [Crossref] 131. Luo, M.; Yin, Y.; Zhou, Z.; Zhang, H.; Chen, X.; Wang, H.; Zhu, Z. J.; Nat. Commun. 2023, 14, 1813. [Crossref] 132. Marshall, A. P.; Johnson, A. R.; Vega, M. M.; Thomson, R. J.; Carlson, E. E.; J. Nat. Prod. 2020, 83, 159. [Crossref] 133. Belhadji, K. A.; Bouzouina, M.; Addou, A.; Phytotherapie 2022, 20, 288. [Crossref] 134. Jensen, P. R.; Chavarria, K. L.; Fenical, W.; Moore, B. S.; Ziemert, N.; J. Ind. Microbiol. Biotechnol. 2014, 41, 203. [Crossref] 135. Wei, L.; Niraula, D.; Gates, E. D. H.; Fu, J.; Luo, Y.; Nyflot, M. J.; Bowen, S. R.; El Naqa, I. M.; Cui, S.; Br. J. Radiol. 2023, 96, 20230211. [Crossref] 136. Valiante, V.; ChemBioChem 2023, 24, e202300008. [Crossref] 137. Brunetti, A. E.; Carnevale Neto, F.; Vera, M. C.; Taboada, C.; Pavarini, D. P.; Bauermeister, A.; Lopes, N. P.; Chem. Soc. Rev. 2018, 47, 1574. [Crossref] 138. Merwin, N. J.; Mousa, W. K.; Dejong, C. A.; Skinnider, M. A.; Cannon, M. J.; Li, H.; Dial, K.; Gunabalasingam, M.; Johnston, C.; Magarvey, N. A.; Proc. Natl. Acad. Sci.U. S. A. 2020, 117, 371. [Crossref] 139. Eldjárn, G. H.; Ramsay, A.; Van Der Hooft, J. J. J.; Duncan, K. R.; Soldatou, S.; Rousu, J.; Daly, R.; Wandy, J.; Rogers, S.; PLoS Comput. Biol. 2021, 17, e1008920. [Crossref] 140. Cao, L.; Gurevich, A.; Alexander, K. L.; Naman, C. B.; Leão, T.; Glukhov, E.; Luzzatto-Knaan, T.; Vargas, F.; Quinn, R.; Bouslimani, A.; Nothias, L. F.; Singh, N. K.; Sanders, J. G.; Benitez, R. A. S.; Thompson, L. R.; Hamid, M. N.; Morton, J. T.; Mikheenko, A.; Shlemov, A.; Korobeynikov, A.; Friedberg, I.; Knight, R.; Venkateswaran, K.; Gerwick, W. H.; Gerwick, L.; Dorrestein, P. C.; Pevzner, P. A.; Mohimani, H.; Cell Syst. 2019, 9, 600. [Crossref] 141. Uppal, K.; Ma, C.; Go, Y. M.; Jones, D. P.; Bioinformatics 2018, 34, 701. [Crossref] 142. Chung, S. Y.; Wong, L.; Trends Biotechnol. 1999, 17, 351. [Link] acessado em julho 2025 143. De Bodt, S.; Hollunder, J.; Nelissen, H.; Meulemeester, N.; Inzé, D.; New Phytol. 2012, 195, 707. [Crossref] 144. Chen, Y.; Li, E. M.; Xu, L. Y.; Metabolites 2022, 12, 357. [Crossref] 145. Paired Omics Data Platform, https://pairedomicsdata.bioinformatics.nl/, acessado em julho 2025. 146. MetaboLights, https://www.ebi.ac.uk/metabolights/, acessado em julho 2025. 147. Geer, L. Y.; Marchler-Bauer, A.; Geer, R. C.; Han, L.; He, J.; He, S.; Liu, C.; Shi, W.; Bryant, S. H.; Nucleic Acids Res. 2010, 38, D492. [Crossref] 148. Nordberg, H.; Cantor, M.; Dusheyko, S.; Hua, S.; Poliakov, A.; Shabalov, I.; Smirnova, T.; Grigoriev, I. V.; Dubchak, I.; Nucleic Acids Res. 2014, 42, D26. [Crossref] 149. Kersten, R. D.; Weng, J. K.; Proc. Natl. Acad. Sci. U. S. A. 2018, 115, E10961. [Crossref] 150. Ayon, N. J.; Earp, C. E.; Gupta, R.; Butun, F. A.; Clements, A. E.; Lee, A. G.; Dainko, D.; Robey, M. T.; Khin, M.; Mardiana, L.; Longcake, A.; Rangel-Grimaldo, M.; Hall, M. J.; Probert, M. R.; Burdette, J. E.; Keller, N. P.; Raja, H. A.; Oberlies, N. H.; Kelleher, N. L.; Caesar, L. K.; Metabolomics 2024, 20, 90. [Crossref] 151. Chen, L.; Zhang, S.; Wang, Y.; Sun, H.; Wang, S.; Wang, D.; Duan, Y.; Niu, J.; Wang, Z.; Int. J. Biol. Macromol. 2023, 253, 127044. [Crossref] 152. Paulo, B. S.; Recchia, M. J. J.; Lee, S.; Fergusson, C. H.; Romanowski, S. B.; Hernandez, A.; Krull, N.; Liu, D. Y.; Cavanagh, H.; Bos, A.; Gray, C. A.; Murphy, B. T.; Linington, R. G.; Eustaquio, A. S.; Chem. Sci. 2024, 15, 16567. [Crossref] 153. Mai, Z.; Kim, K.; Richardson, M. B.; Deschênes, D. A. R.; Garza ‐ Garcia, J. J. O.; Shahsavarani, M.; Perley, J. O.; Njoku, D. I.; Deslongchamps, G.; De Luca, V.; Qu, Y.; Plant J. 2024, 120, 2770. [Crossref] 154. Manochkumar, J.; Ramamoorthy, S.; Curr. Sci. 2024, 126, 19. [Crossref] 155. Saldívar-González, F. I.; Aldas-Bulos, V. D.; Medina-Franco, J. L.; Plisson, F.; Chem. Sci. 2022, 13, 1526. [Crossref] 156. Mullowney, M. W.; Duncan, K. R.; Elsayed, S. S.; Garg, N.; Van Der Hooft, J. J. J.; Martin, N. I.; Meijer, D.; Terlouw, B. R.; Biermann, F.; Blin, K.; Durairaj, J.; González, M. G.; Helfrich, E. J. N.; Huber, F.; Leopold-Messer, S.; Rajan, K.; De Rond, T.; Van Santen, J. A.; Sorokina, M.; Balunas, M. J.; Beniddir, M. A.; Van Bergeijk, D. A.; Carroll, L. M.; Clark, C. M.; Clevert, D. A.; Dejong, C. A.; Du, C.; Ferrinho, S.; Grisoni, F.; Hofstetter, A.; Jespers, W.; Kalinina, O. V.; Kautsar, S. A.; Kim, H.; Leao, T. F.; Masschelein, J.; Rees, E. R.; Reher, R.; Reker, D.; Schwaller, P.; Segler, M.; Skinnider, M. A.; Walker, A. S.; Willighagen, E. L.; Zdrazil, B.; Ziemert, N.; Goss, R. J. M.; Guyomard, P.; Volkamer, A.; Gerwick, W. H.; Kim, H. U.; Müller, R.; Van Wezel, G. P.; Van Westen, G. J. P.; Hirsch, A. K. H.; Linington, R. G.; Robinson, S. L.; Medema, M. H.; Nat. Rev. Drug Discovery 2023, 22, 895. [Crossref] 157. Sword, T. T.; Abbas, G. S. K.; Bailey, C. B.; Front. Nat. Prod. 2024, 3, 1353362. [Crossref] 158. Bradley, S. A.; Lehka, B. J.; Hansson, F. G.; Adhikari, K. B.; Rago, D.; Rubaszka, P.; Haidar, A. K.; Chen, L.; Hansen, L. G.; Gudich, O.; Giannakou, K.; Lengger, B.; Gill, R. T.; Nakamura, Y.; Dugé de Bernonville, T.; Koudounas, K.; Romero-Suarez, D.; Ding, L.; Qiao, Y.; Frimurer, T. M.; Petersen, A. A.; Besseau, S.; Kumar, S.; Gautron, N.; Melin, C.; Marc, J.; Jeanneau, R.; O'Connor, S. E.; Courdavault, V.; Keasling, J. D.; Zhang, J.; Jensen, M. K.; Nat. Chem. Biol. 2023, 19, 1551. [Crossref] 159. Wilson, K.; de Rond, T.; Burkhardt, I.; Steele, T. S.; Schäfer, R. J. B.; Podell, S.; Allen, E. E.; Moore, B. S.; Proc. Natl. Acad. Sci.U. S. A. 2023, 120, e2220934120. [Crossref] 160. Carroll, L. M.; Larralde, M.; Fleck, J. S.; Ponnudurai, R.; Milanese, A.; Cappio, E.; Zeller, G.; BioRxiv, 2021. [Crossref] 161. Sanchez, S.; Rogers, J. D.; Rogers, A. B.; Nassar, M.; McEntyre, J.; Welch, M.; Hollfelder, F.; Finn, R. D.; BioRxiv, 2023. [Crossref] 162. Mukherjee, M.; Blair, R. H.; Wang, Z. Q.; Metab. Eng. 2022, 74, 139. [Crossref]
Editor Convidado responsável pelo artigo: Lucas S. Abreu |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access