Revisão
|
|
| A importância da determinação analítica de intermediários reativos e de seus produtos de reações com biomacromoléculas: uma mini revisão Importance of analytical determination of reactive intermediates and their reaction products with biomacromolecules: a mini review |
|
Eline S. Gonçalves; Juliana M. Bastos da Silva; Thelma Pavesi; Josino C. Moreira
Fundação Oswaldo Cruz, Avenida Brasil, 4365, Manguinhos, 21040-360 Rio de Janeiro - RJ, Brasil Recebido em 25/06/2013 *e-mail: elinesg@gmail.com Metabolic reactive intermediates can react with biomolecules such as DNA and proteins to produce adducts. Recently, research has shown that such adducts can act as precursors of some chronic diseases (cancer, Parkinson's, immunologic system diseases, etc.), and their determination is important because they are biomarkers of undesirable health effects. These compounds are produced at very low concentrations, but the development and dissemination of sensitive new analytical tools, especially those based on chromatography coupled to other analytical instruments, make such determinations possible. This mini review is focused on the formation of reactive intermediates, their reaction with biomolecules, and the importance of their determination. INTRODUÇAO Algumas reaçoes idiossincráticas e várias doenças têm sido relacionadas a produtos do metabolismo de xenobióticos pelo organismo. Os xenobióticos sao substâncias químicas alheias ao sistema biológico com as quais o homem tem contato, tais como medicamentos, produtos industriais, alimentos, pesticidas, cosméticos, poluentes, etc. De fato, o homem tem sido exposto a um número continuamente crescente destas substâncias como resultado do desenvolvimento científico, tecnológico e industrial, seja a partir dos alimentos, do ambiente ou mesmo de procedimentos terapêuticos.1 Por serem substâncias estranhas ao organismo e para prevenir qualquer tipo de dano que eventualmente podem causar, o organismo precisa transformá-las em formas atóxicas e/ou eliminá-las, e para isto, possuem uma série de sistemas enzimáticos capazes de metabolizá-las, por meio de reaçoes bioquímicas. Este mecanismo é particularmente importante quando os xenobióticos apresentam características lipofílicas, já que tendem a se acumular nos tecidos gordurosos e sao mais dificilmente excretados pelo organismo. Ou seja, ter-se-ia um processo de bioacumulaçao destas substâncias e os consequentes efeitos que pode trazer aos organismos.1 Em geral, o metabolismo tende a produzir substâncias mais hidrossolúveis e menos reativas que as moléculas originais, facilitando sua eliminaçao e diminuindo o risco de agravos à saúde. Entretanto, nem sempre isto é verdade, como será discutido a seguir.
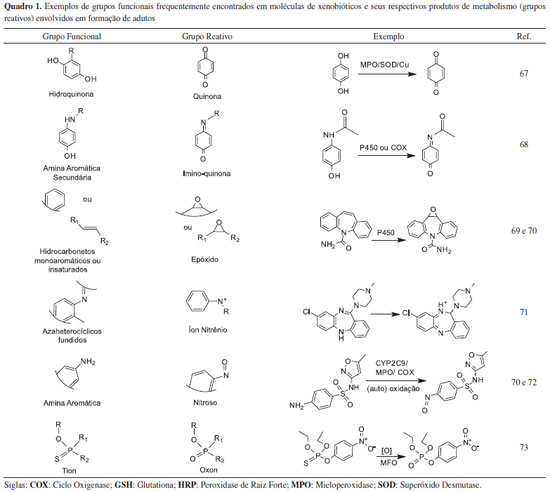
METABOLISMO DE XENOBIOTICOS E A FORMAÇAO DE INTERMEDIARIOS REATIVOS Os processos metabólicos podem ser considerados como se ocorressem em duas fases: fases I e II (eventualmente considera-se uma fase adicional, a fase III que corresponderia a exclusao do xenobiótico nao metabolizado para fora das células diminuindo sua concentraçao intracelular).2 Na fase I, sob a açao de cerca de 10 famílias enzimáticas oxidantes, redutoras (açao sobre grupos azo e nitro) ou hidrolíticas (hidrólise de ésteres e amidas), grupos funcionais como -OH, -NH2, -SH ou -COOH sao expostos ou introduzidos nas moléculas xenobióticas, facilitando a excreçao, mas geralmente aumentando a reatividade dos produtos resultantes. Embora a funçao principal do metabolismo seja a de detoxificaçao, ou seja, de eliminaçao dos agentes tóxicos contaminantes, em algumas situaçoes os xenobióticos podem ser bioativados, principalmente por enzimas da fase I. Nestas circunstâncias sao formadas substâncias eletrofílicas capazes de sofrerem processos de reduçao que resultam na formaçao de radicais livres, além da reaçao com uma série de nucleófilos endógenos, dentre os quais proteínas, DNA e RNA, formando adutos. Inclusive, as próprias enzimas atuantes na fase I do metabolismo podem ser inativadas por reaçoes de conjugaçao deste tipo.3 O Quadro 1 apresenta alguns exemplos de grupos funcionais comumente presentes nos xenobióticos, bem como os grupos reativos, resultantes de seus metabolismos enzimáticos.
Eletrófilos e espécies oxigenadas reativas sao também normalmente formados pelo metabolismo celular e, assim como os metabólitos reativos, podem danificar as células e, desta forma, suas concentraçoes devem ser rigidamente controladas para se evitar efeitos indesejados.4 Vários exemplos de bioativaçao sao bem conhecidos, como aquele responsável pela toxicidade de pesticidas organofosforados e carbamatos a partir do qual ocorre a formaçao de oxons-análogos resultantes da desulfuraçao oxidativa de suas moléculas e que sao capazes de reagirem com as colinesterases; ou ainda, a formaçao de epóxidos durante o metabolismo de hidrocarbonetos policíclicos aromáticos capazes de reagirem com DNA ou biomoléculas (vide Quadro 1). Na fase II (fase de conjugaçao) outros sistemas enzimáticos catalisam a conjugaçao desses metabólitos reativos com moléculas mais polares inativando-os e facilitando suas excreçoes por meio da bile ou da urina. Entretanto, produtos bioativados podem ser também resultantes desta fase. Em geral, três tipos de reaçoes na fase II podem ser capazes de originarem produtos tóxicos: a produçao de um conjugado reativo, a ativaçao dos conjugados enzima-metabólito e a liberaçao do metabólito reativo original.5-9 As principais reaçoes que acontecem nesta fase sao a sulfataçao (grupos hidroxila e amina ligados a aromáticos e alcoóis), a metilaçao (grupos hidroxila ligados a aromáticos, aminas primárias e secundárias, tiol), a glicuronizaçao (grupos hidroxila, carboxila, carbonila, amina primária e secundária, tiol), a acetilaçao (aminas aromáticas e alifáticas, hidrazidas e sulfonamidas) e a conjugaçao com glutationa (epóxidos e haletos orgânicos) ou aminoácidos, as quais ocorrem com a participaçao das enzimas sulfotransferases (SULT), metiltransferases (MT), N-acetiltransferases (NAT), glutationa-S-transferase (GST), uridina-5-difosfo α-D-glucurônico transferases (UDP-transferases), respectivamente.1 As duas fases dos processos metabólicos devem atuar de maneira sincronizada. Um excelente artigo de revisao destes processos foi elaborado por Liska e colaboradores (2006).10 Assim, quando a induçao da atividade enzimática de uma das fases ocorre sem que haja co-induçao da fase subsequente, os produtos intermediários do metabolismo podem se acumular no organismo e/ou reagirem com biomoléculas como o DNA, o RNA ou proteínas podendo causar impactos sobre o organismo. Estas reaçoes ocorrem por mecanismos de substituiçao ou de adiçao, e envolvem a doaçao de um par de elétrons pelo nucleófilo e a consequente formaçao de ligaçao covalente.11,12 O conceito de ácidos e bases duros e macios de Pearson tem sido bastante utilizado para a previsao qualitativa destes tipos de reaçoes.11,12 Embora estes conceitos sejam de importância na previsao reacional, a reatividade de um grupo é consequência nao apenas de suas características de dureza/maciez, mas também é funçao de fatores estéricos e eletrônicos dependentes da estrutura terciária das proteínas (nucleófilos).11,13 Os intermediários reativos reagem com sítios nucleofílicos das proteínas como os grupos amina terminal, tiol (cisteína), imidazol (histidina), amino e guanidino, grupos de lisina e arginina, carboxila de ácido aspártico e glutâmico e anel aromático da tirosina.11 No caso do DNA, por exemplo, os principais sítios de alquilaçao sao os átomos de N e O (N3 da adenina e N7 e O da guanina) sendo que o N1 e N7 da adenina, os N3 da citosina e da guanina e O da timina podem ser igualmente utilizados.14 Ao todo existem 18 possibilidades de ataque à molécula do DNA.15 Um exemplo de um destes processos, representando esquematicamente a ativaçao metabólica da Aflatoxina B1 pelo sistema CYP 450 e a subsequente reaçao de um de seus metabólitos reativos (AFB1 8,9-epóxido) com o sítio N7 da guanina da molécula de DNA, pode ser visto na Figura 1.
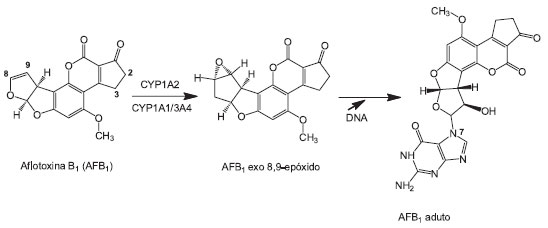 Figura 1. Representaçao esquemática da ativaçao metabólica da Aflatoxina B1 pelo sistema CYP 450 e da reaçao de seu metabólito reativo (AFB1 8,9-epóxido) com o sítio N7 da guanina da molécula de DNA (adaptada da ref. 16)
Estas reaçoes envolvem também co-fatores adquiridos através da dieta e sao influenciadas por fatores ambientais.2,17 Ou seja, a combinaçao de todos estes fatores faz com que exista uma grande variaçao inter-individual nas concentraçoes de adutos após a exposiçao a um dado xenobiótico. Realmente, a literatura científica tem relatado várias associaçoes epidemiológicas entre a eficiência dos sistemas enzimáticos de detoxificaçao e eliminaçao de vários xenobióticos e de seus produtos de metabolismo no organismo humano e o desenvolvimento de algumas doenças. Dentre essas doenças podemos citar alguns tipos de câncer, a doença de Parkinson, algumas disfunçoes do sistema imunológico, agranulocitose, etc.2,18 A fase I do metabolismo tem papel preponderante na formaçao de metabólitos intermediários reativos, embora, como dito anteriormente, esta atividade nao seja exclusiva desta fase. Dentre as enzimas que participam da fase I, os citocromos P450 constituem a superfamília mais importante. Esta superfamília está envolvida na oxidaçao de mais de cerca de 70% dos xenobióticos.19 Os CYP 450, que sao proteínas contendo grupos heme-tiolato, têm em comum as seguintes propriedades químicas: i- habilidade de se ligar e ativar átomos de oxigênio; ii- capacidade de se ligar a peróxido de hidrogênio ou peróxidos com a participaçao da ligaçao heme-apoproteína e usar um dos oxigênios em monooxigenaçoes.20 A reaçao geral da catálise pelo CYP 450 é representada na Figura 2, embora exceçoes sejam conhecidas.21 Estudos mais detalhados destes mecanismos sao encontrados na literatura.21-26
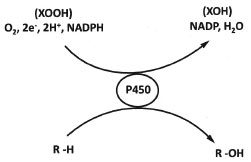 Figura 2. Representaçao da reaçao geral do metabolismo com a participaçao das enzimas do CYP 450 (adaptada da ref. 20)
O estudo do genoma humano revelou a presença de 107 genes P450 dos quais 59 sao ativos e 58 pseudogenes, o que demonstra o amplo polimorfismo desta superfamília.27,28 Polimorfos com atividades afins sao agrupados em famílias. Dentro dos CYP 450 as famílias CYP 1-3 estao relacionadas ao metabolismo de drogas e outros xenobióticos, enquanto as famílias 4, 11, 17, 19 e 21 estao envolvidas no metabolismo de substâncias endógenas como ácidos graxos, esteróis, eicosanóides, ácidos biliares e vitaminas lipossolúveis.28 Excelentes artigos de revisao sobre o polimorfismo da família CYP 450 estao disponíveis.19,26-28 O polimorfismo do sistema citocromo P450 têm sido considerado como um importante fator para as diferenças interindividuais observadas tanto na eliminaçao quanto nas respostas humanas e nos riscos de efeitos adversos em diferentes indivíduos.26,29 O metabolismo exibe significativa variabilidade individual e é afetado por aspectos genéticos (polimorfismos), ambientais, idade e gênero, estilo de vida, dieta e estado de saúde (presença de alguma doença).30 Estes fatores podem afetar a toxidade de um xenobiótico alterando o processo metabólico com um todo, sua taxa de eliminaçao ou taxa de ativaçao.1 Consequentemente, a formaçao dos intermediários reativos bem como de suas reaçoes com biomoléculas, considerada etapa essencial para efeitos adversos, sao igualmente influenciadas por estas variáveis, além da natureza do eletrófilo, sua disponibilidade e o impedimento estérico eventualmente causado pela estrutura terciária das proteínas.31 Por outro lado, como as enzimas sao codificadas por genes, variaçoes genéticas nestes genes codificadores produzem alteraçoes na expressao, atividade e estabilidade das enzimas por eles codificadas. Dentre estas alteraçoes destacam-se a especificidade pelos substratos e a cinética enzimática. Ou seja, podem existir diferenças individuais nas atividades de uma determinada enzima dentro de um mesmo grupo populacional, como resultado de terem sido expressas por diferentes alelos. A estas diferentes constituiçoes enzimáticas dá-se o nome de polimorfismo. Sendo assim, considerando-se as possíveis variaçoes individuais no metabolismo, alguns indivíduos podem apresentar maior susceptibilidade a sofrerem efeitos indesejados quando expostos a uma substância, sob as mesmas condiçoes, que outros. Por exemplo, alguns estudos reportam a relaçao entre o câncer de próstata e o polimorfismo do citocromo 4501A1, importante enzima de detoxificaçao da fase I, responsável pela atividade aril-hidroxilase e envolvida no metabolismo de um grande número de substâncias carcinogênicas. Neste caso, dois polimorfos têm sido associados ao câncer: o 3801T>C e o 2455A>G. Entretanto, uma meta-análise dos diversos dados reportados para este estudo encontrou uma correlaçao positiva (risco aumentado) somente entre os portadores do polimorfo 2455A>G e nenhuma correlaçao entre aqueles que possuíam o polimorfo 3801T>C, o que demonstra a influência do polimorfismo gênico sobre os efeitos tóxicos de xenobióticos.32
DETERMINAÇAO DE METABOLITOS INTERMEDIARIOS REATIVOS COMO BIOINDICADORES O interesse em se conhecer as relaçoes existentes entre a exposiçao à xenobióticos, a contaminaçao humana, seus efeitos sobre a saúde e, particularmente, o porquê de certos organismos serem mais susceptíveis que outros a estes efeitos é bastante antigo, e para isto dispomos de uma série de biomarcadores ou bioindicadores utilizados para a avaliaçao de riscos.33,34 As principais categorias destes biomarcadores e suas importâncias biológicas relativas sao apresentadas na Figura 3.
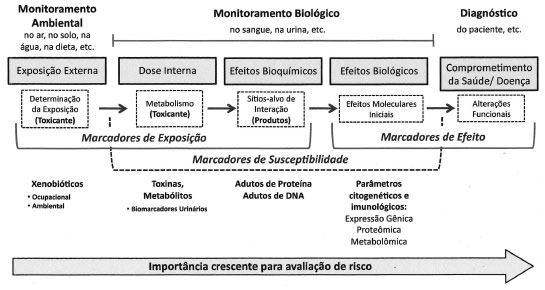 Figura 3. Representaçao esquemática dos tipos de biomarcadores utilizados no monitoramento biológico (adaptada das refs. 33 e 34)
Até pouco tempo, as avaliaçoes de risco à saúde se baseavam em medidas da concentraçao do agente tóxico em amostras ambientais coletadas nos locais onde havia exposiçao humana (indicadores externos) e na determinaçao das concentraçoes em fluidos biológicos, principalmente em sangue e urina (indicadores de dose interna). Na urina pode ser determinada tanto a substância nao metabolizada quanto os produtos metabolizados. Como por exemplo, a determinaçao de fenol no caso de exposiçao a benzeno. Estas determinaçoes, embora sejam importantes, nao mostram com exatidao qual a dose biológica realmente efetiva, ou seja, aquela capaz de atingir os sítios de toxicidade e, consequentemente, trazer riscos à saúde. Considerando-se que para exercer seus efeitos tóxicos, uma determinada substância deve ter acesso às estruturas biológicas sensíveis, pode-se depreender da Figura 3 que existe uma hierarquia entre estes indicadores e, à medida que estes ficam hierarquicamente mais importantes, suas determinaçoes tornam-se mais complexas devido ao aumento da complexidade das amostras e às baixas concentraçoes relativas presentes concentraçoes da ordem de pico ou fentogramas. O desenvolvimento de novas ferramentas analíticas com capacidade de determinarem concentraçoes compatíveis com aquelas observadas no organismo, associado ao advento das tecnologias resultantes do desenvolvimento da biologia molecular como, por exemplo, a genômica, a proteômica, a transcriptômica e a metabolômica (denominadas tecnologias "omicas") possibilitaram hoje a realizaçao de análises de material biológico de grande complexidade para a detecçao de compostos-alvo nos níveis de concentraçoes adequados e exatidao aceitável.35 Sendo assim, tornou-se possível a determinaçao de uma série de metabólitos intermediários reativos, geralmente realizada a partir de seus produtos de reaçao com biomoléculas, o que tem facilitado o entendimento dos processos toxicológicos, possibilitando, inclusive, um maior entendimento dos fatores determinantes da suscetibilidade individual. Neste contexto, adutos de DNA e de proteínas têm sido utilizados como biomarcadores importantes para estes estudos. Inúmeros métodos têm sido propostos para a determinaçao quantitativa de adutos.36-43 Sob o ponto de vista analítico, adutos de albumina e de hemoglobina sao os mais investigados, pois quimicamente sao mais estáveis, já que nao sofrem a açao de mecanismos de reparo característicos das moléculas de DNA e acessíveis em quantidades superiores aos adutos de DNA. Ademais, tem sido observada uma relaçao de proporcionalidade entre os adutos de hemoglobina e de DNA. A Figura 4 apresenta as concentraçoes e as estabilidades relativas de metabólitos urinários, adutos de hemoglobina e DNA.
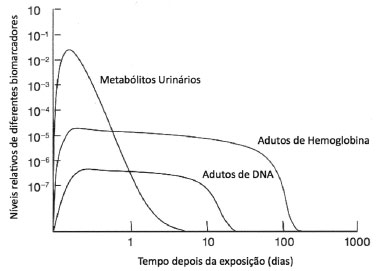 Figura 4. Variaçao temporal das concentraçoes relativas de diversos metabólitos e adutos após uma exposiçao única (adaptada da ref. 17)
Dentre as metodologias analíticas atualmente disponíveis para a determinaçao quantitativa de adutos três grupos de técnicas se sobressaem: a pós marcaçao com P32, os imunoensaios e as cromatografias gasosa e líquida (CG e CL), particularmente aquelas associadas à Espectrometria de Massa (EM). A técnica de pós marcaçao com P32 requer pequena quantidade de DNA (1 - 10 µg) e tem excelente sensibilidade (1 aduto/1010 nucleotídeos) e versatilidade, mas tem limitaçoes quanto a identificaçao dos adutos e pode fornecer resultados subestimados. Os imunoensaios (ELISA e RIA) tem boa sensibilidade (1 aduto/108 nucleotídeos), sao baratos e facilmente aplicados, mas exigem quantidade de DNA entre 1 - 200 µg e anticorpos produzidos contra adutos específicos. Estas técnicas ainda apresentam problemas de reaçoes-cruzadas com outros adutos. Já as técnicas cromatográficas apresentam excelente sensibilidade (1 aduto por 109 - 1011 nucleotídeos), requerem amostras de 10 a 100 µg de DNA, exigem equipamentos e pessoal especializado e muitas vezes requerem padroes analíticos, mas tem a vantagem de possibilitar a identificaçao do aduto, de acordo com o método de detecçao utilizado. Uma discussao mais aprofundada destas metodologias pode ser encontrada na literatura.34,36-38,42 Dentre as metodologias cromatográficas, aquelas que se baseiam no uso de sistemas de detecçao por espectrometria de massa-massa (EM/EM) têm sido recomendadas para a determinaçao de adutos, notadamente aquelas em que esse sistema esteja acoplado à cromatografia líquida (CL-EM/EM), devido à elevada especificidade, versatilidade e sensibilidade que tem sido obtidas com equipamentos cada vez mais modernos.39,42-44 De fato, a CL-EM/EM é uma ferramenta que somente há poucos anos começou a ser empregada por um número representativo de laboratórios bioanalíticos, de modo que estudos relacionados à determinaçao de adutos e suas correlaçoes com a suscetibilidade individual e doenças humanas ainda sao relativamente poucos e devem ser incentivados.13,34,36-39,45-65 Adutos de DNA estao presentes em amostras teciduais em concentraçoes tao baixas quanto <100 fmol/pgDNA ou <3 adutos/105 nucleotídeos, enquanto os de proteínas sao mais abundantes situando-se na faixa de pmol/ml de plasma ou de fmol/mg de proteína.66 No caso de adutos de DNA, a maioria dos métodos consegue determinar 1 aduto em 109 nucleotídeos. Entretanto, alguns métodos baseados em detecçao por espectrometria de massa associado à cromatografia líquida conseguem medir 1 aduto para cada 1012 nucleotídeos ou ainda 500 fg (aproximadamente 1 fmol).36,39 Atualmente, é possível determinar adutos de 4-aminobifenila-DNA na bexiga de pacientes cancerosos por CL-EM/EM com limite de detecçao de 1 aduto por 109 bases nitrogenadas.45 Nos exemplos reportados a seguir é possível observar a versatilidade e a potencialidade desta técnica. Os adutos de DNA N-(deoxiguanosin-8-il), 2-amino-1-metil-6-fenilimidazol[4,5-b]piridina, 2-amino-9H-pirido[2,3-b]indol, 2-amino-3,8-dimetilmidazo[4,5-f]quinoxalina e 4-aminobifenila, presentes na saliva de fumantes e consumidores de produtos cárneos, foram determinados por CL-EM/EM.61 O método proposto foi considerado eficiente para a determinaçao destas moléculas em concentraçoes de 1-9 adutos a cada 108 bases nitrogenadas. Além disso, a saliva mostrou-se uma matriz útil para este tipo de análise, principalmente, por apresentar uma coleta pouco invasiva. Os adutos formados entre o DNA e o ácido 8-metoxi-6-nitrofenantro-[3,4-d]-1,3-dioxolo-5-carboxílico, substância potencialmente carcinogênica da família dos nitrofenantrenos e encontrada em preparaçoes a base de Aristolochia sp., foram determinados em tecidos renais em residentes de Taiwan, por CL-EM/EM. Em pacientes humanos foram encontrados valores que se situaram entre 9 e 338 adutos a cada 108 de bases nitrogenadas.59 Intermediários reativos de monômeros de vinila (N2,3-Etenoguanina) e DNA foram analisados por CL-EM/EM como base para o entendimento de seu potencial mutagênico e os resultados sugerem que estes adutos contribuem para a carcinogênese desta substância.63 A formaçao de adutos de DNA por meio de sua reaçao com acetaldeído em células humanas é reportado.65 De acordo com os autores, há formaçao inequívoca de aduto, o que pode facilitar o entendimento dos processos de carcinogênese por exposiçao à esta substância. Substâncias capazes de formarem eteno-adutos com DNA foram determinadas em urina de trabalhadores nao expostos para a determinaçao dos níveis basais destes adutos.65
CONCLUSOES O entendimento do papel dos adutos na bioquímica dos sistemas biológicos é inegavelmente complexo, mas esta complexidade deve ser encarada como um incentivo ao seu estudo. Isto perpassa a necessidade de se desenvolver metodologias eficientes e para isto, novas ferramentas analíticas já se encontram disponíveis, dentre as quais sobressaem os sistemas EM/EM. Embora o número de estudos que utilizam a CL-EM/EM esteja crescendo nos últimos anos, ainda há grande necessidade de pesquisas e nota-se que resultados discrepantes têm sido encontrados em estudos relacionando adutos como precursores de efeitos biológicos precoces ou mesmo de doenças tais como os diversos tipos de câncer, arteriosclerose, Alzheimer, diabetes, etc. Entretanto, qualquer que seja o seu papel, o conhecimento do mesmo faz-se necessário para o entendimento dos processos bioquímicos vitais, o que torna esta área altamente atual e atraente. De fato, este conhecimento é necessário e essencial para o entendimento de doenças relacionadas ao seu desenvolvimento, assim como suas prevençoes.
REFERENCIAS 1. Marrs, T. C.; Rimbell, J. A. Em General and Applyed Toxicology; Ballantyne, B.; Marrs, T. C.; Syversen, T., eds.; Wiley: New York, 2009, ed. 3, vol. 1, p. 89-126. 2. Lyska, D.-A. J.; Altern. Med. Rev. 1998, 3, 187. 3. Fontana, E.; Dansette, P. M.; Poli, S. M.; Curr. Drug Metab. 2005, 6, 413. 4. Zhang, Q.; Jingbo, P.; Woods, C. G.; Andersen, M. E.; Toxicol. Appl. Pharmacol. 2009, 237, 345. 5. Glatt, H.; Chem. Biol. Interact. 2000, 129, 141. 6. Huber, P. C.; Almeida, W. P.; Fatima, A.; Quim. Nova 2008, 31, 1170. 7. Jancova, P., Anzenbacher, P., Anzenbacherova, E.; Biomed. Pap.Med. Fac. Univ. Palacky Olomouc Czech Repub. 2010, 154, 103. 8. van Bladeren, P. J.; Chem. Biol. Interact. 2000, 129, 61. 9. Hinson, J. A.; Forket, P. G.; Can. J. Physiol. Pharmacol. 1995, 73, 1407. 10. Liska, D.; Lyon, M.; Jones, D. S.; Explore 2006, 2, 122. 11. LoPachin, R. M.; DiCaprio, A. P.; Toxicol. Sci. 2005, 86, 214. 12. Ho, T.-S.; Ho, H. C.; Hamilton, L. D.; Chem. Biol. Interact. 1978, 23, 65. 13. Rubino, F. M.; Pitton, M.; Di-Fabio, D.; Colombi, A.; Mass Spectrom. Rev. 2009, 28, 725. 14. Tarun, M.; Rusling, J. F.; Crit. Rev. Eukaryot. Gene Expr. 2005, 15, 295. 15. Dipple, A.; Carcinogenesis 1995, 16, 437. 16. Zhou, S.-F.; Wang, B.; Yang, L.-P.; Liu, J.-P.; Drug Metabolism Reviews 2010, 42, 268. 17. Henderson, R. F.; Bechtold, W. E.; Bond. J. A.; Sun, J. D.; Crit. Rev. Toxicol. 1989, 20, 65. 18. Kaddurah-Daouk, R.; Kristal B.S.; Weinshilboum, R. M.; Annu. Rev. Pharmacol. Toxicol. 2008, 48, 653. 19. Zhou, S.-F.; Liu, J.-P.; Chowbay, B.; Drug Metabolism Reviews 2009, 41, 89. 20. Anzenbacher, P.; Anzenbacherová, E.; Cell. Mol. Life Sci. 2001, 58, 737. 21. Guengerich, F. P.; Chem. Res. Toxicol. 2001, 14, 611. 22. Masubuchi, Y.; Horie, T.; Crit. Rev. Toxicol. 2007, 37, 389. 23. Masic, L. P.; Curr. Drug Metab. 2011, 12, 35. 24. Hollenberg, P. F.; Kent, U. M.; Bumpus, N. N.; Chem. Res. Toxicol. 2008, 21, 189. 25. Cooper, H. L.; Groves, J. T.; Arch. Biochem. Biophys. 2011, 507, 111. 26. Ingelman-Sundberg, M.; Naunyn-Schmiedeberg's Arch. Pharmacol. 2004, 369, 89. 27. Singh, D.; Kashyap, A.; Pandey, R. V.; Saini, K.S.; Drug Discovery Today 2011, 16, 93. 28. Guengerich, F. P.; Chem. Res. Toxicol. 2008, 21, 70. 29. Olden, K.; White, S.; Med. Clin. North Am. 2005, 89, 721. 30. Zhang, Z. Y.; Wong, Y. N.; Curr. Drug Metab. 2005, 6, 241. 31. Kalgutkar, A. S.; Gardner, I.; Obach, R. S.; Shaffer, C. L.; Callegari, E.; Henne, K. R.; Mutlib, A. E.; Dalvie, D. K.; Lee, J. S.; Nakai, Y.; O'Donnell, J. P.; Boer, J.; Harriman, S. P.; Curr. Drug Metab. 2005, 6, 161. 32. Li, H.; Xiao, D.; Hu, L.; He, T.; Mol. Biol. Rep. 2012, 39, 10273. 33. Gyorffy, E.; Anna, L.; Kovacs, K.; Rudnai, P.; Schoket, B.; Mutagenesis 2008, 23, 1. 34. Farmer, P. B.; Singh, R.; Mutat. Res., Rev. Mutat. Res. 2008, 659, 68. 35. Aardema, M.J.; MacGregor, J.T.; Mutat. Res. 2002, 499, 13. 36. Poirier, M. C.; Santella, R. M.; Weston, A.; Carcinogenesis 2000, 21, 353. 37. Himmelstein, M. W.; Boogaard, P. J.; Cadet, J.; Farmer, P. B.; Kim, J. H.; Martin, E. A.; Persaud, R.; Shuker, D. E.; Crit. Rev. Toxicol. 2009, 39, 679. 38. Tarun, M.; Rusling, J. F.; Crit. Rev. Eukaryot. Gene Expr. 2005, 15, 295. 39. Koivisto, P.; Peltonen, K.; Anal. Bioanal. Chem. 2010, 398, 2563. 40. Netto, A. D. P.; Moreira, J. C.; Arbilla, G.; Ferreira, L. F. V.; Oliveira, A.S.; Barek, J.; Quim. Nova 2000, 23, 765; Jarabek, A. M.; Pottenger, L. H.; Andrews, L. S.; Casciano, D.; Embry, M. R.; Kim, J. H.; Julian Preston, R.; Vijayaraj Reddy, M.; Schoeny, R.; Shuker, D.; Skare , J.; Swenberg, J.; Williams, G. M.; Zeiger, E.; Crit. Rev. Toxicol. 2009, 39, 659. 41. Schmeiser, H. H.; Stiborova, M.; Arlt, V. M.; Methods Mol. Biol. 2013, 1044, 389.; Klaene, J.J.; Sharma, V.K.; Glick, J.; Vouros, P.; Cancer Letters 2013, 334, 10. 42. Watson, W. P.; Mutti, A.; Biomarkers 2004, 9, 211. 43. Theodoridis, G.; Gika, H. G.; Wilson, I. D.; Mass Spectrom. Rev. 2011, 30, 884. 44. Cadet, J.; Douki, T.; Frelon, S.; Sauvaigo, S.; Pouget, J. P.; Ravanat, J. L.; Free Radical Biol. Med. 2002, 33, 441. 45. Zayas, B.; Stillwell, S. W.; Wishnok, J. S.; Trudel, L. J.; Skipper, P.; Yu, M. C.; Tannenbaum, S. R.; Wogan, G. N.; Carcinogenesis 2007, 28, 342. 46. Sorensen, M.; Autrup, H.; Moller, P.; Hertel, O.; Jensen, S. S.; Vinzents, P.; Knudsen, L. E.; Loft, S.; Mutat. Res., Rev. Mutat. Res. 2003, 544, 255. 47. Metz, T. O.; Zhang, Q.; Page, J. S.; Shen, Y.; Callister, S. J.; Jacobs, J. M.; Smith, R. D.; Biomark. Med. 2007, 1, 159. 48. Liebler, D. C.; Chem. Res. Toxicol. 2006, 19, 610. 49. Bonassi, S.; Neri, M.; Puntoni, R.; Mutat. Res., Fundam. Mol. Mech. Mutagen. 2001, 480, 349. 50. McHale, C. M.; Zhang, L.; Smith, M. T.; Carcinogenesis 2012, 33, 240. 51. Blair, I. A.; Biomed. Chromatogr. 2010, 24, 29. 52. Angerer, J.; Ewers, U.; Wilhelm, M.; Int. J. Hyg. Environ. Health 2007, 210, 201. 53. Yoon, S.; Han, S. S.; Rana, S. V.; J. Environ. Biol. 2008, 29, 1. 54. Zhu, M.; Ma, L.; Zhang, H.; Humphreys, W. G.; Anal. Chem. 2007, 79, 8333. 55. Baillie, T. A.; Pearson, P. G.; Rashed, M. S.; Howald, W. N.; J. Pharm. Biomed. Anal. 1989, 7, 1351. 56. Hoos, J. S.; Damsten, M. C.; de Vlieger, J. S.; Commandeur, J. N.; Vermeulen, N. P.; Niessen, W. M.; Lingeman, H.; Irth, H.; J. Chromatogr. B 2007, 859, 147. 57. Ma, S.; Subramanian, R.; J. Mass Spectrom. 2006, 41, 1121. 58. Tang, W.; Abbott, F. S.; Chem. Res. Toxicol. 1996, 9, 517. 59. Person, M. D.; Monks, T. J.; Lau, S. S.; Chem. Res. Toxicol. 2003, 16, 598. 60. Fisher, A. A.; Labenski, M. T.; Malladi, S.; Gokhale, V.; Bowen, M. E.; Milleron, R. S.; Bratton, S. B.; Monks, T. J.; Lau, S. S.; Biochemistry 2007, 46, 11090. 61. Bessette, E. E.; Spivack, S. D.; Goodenough, A. K.; Wang, T.; Pinto, S.; Kadlubar, F. F.; Turesky, R. J.; Chem. Res. Toxicol. 2010, 23, 1234. 62. Yun, B. H.; Rosenquist, T. A.; Sidorenko, V.; Iden C. R.; Chen, C. H.; Pu, Y. S.; Bonala, R.; Johnson, F.; Dickman, K. G.; Grollman, A. P.; Turesky, R. J.; Chem. Res. Toxicol. 2012, 25, 1119. 63. Mutlu, E.; Collins, L. B.; Stout, M. D.; Upton, P. B.; Daye, L. R.; Winsett, D.; Hatch, G.; Evansky, P.; Swenberg, J. A.; Chem. Res. Toxicol. 2010, 23, 1485. 64. Garcia, C. C.; Angeli, J. P.; Freitas, F. P.; Gomes, O. F.; de Oliveira, T. F.; Loureiro, A. P.; Di Mascio, P.; Medeiro, M. H; J. Am. Chem. Soc. 2011, 133, 9140. 65. Gonzalez-Reche, L. M.; Koch, H. M.; Weiss, T.; Müller, J.; Drexler, H.; Angerer, J.; Toxicol. Lett. 2002, 134, 71. 66. Peng, T.; Li, L. Q.; Peng, M. H.; Liu, Z. M.; Liu, T. W.; Yan, L. N.; Shen, H. M.; Wang, Q.; Wang, K. B.; Liang, R. X.; Wei, Z. L.; Ong, C. N.; Santella, R. M.; Cancer Sci. 2007, 98, 140. 67. Smith, M. T.; Environ. Health Perspect. 1996, 104, 1219. 68. Bessems, J. G.; Vermeulen, N. P.; Crit. Rev. Toxicol. 2001, 31, 55. 69. Bu, H. Z.; Zhao, P.; Dalvie, D. K; Pool, W. R.; Rapid Commun. Mass Spectrom. 2007, 21, 3317. 70. Park, B. K; Pirmohamed, M.; Tingle, M. D.; Madden, S.; Kitteringham, N. R.; Toxicol. In Vitro 1994, 8, 613. 71. Gardner, I.; Leeder, J. S.; Chin, T.; Zahid, N.; Uetrecht, J. P.; Mol. Pharmacol. 1998, 53, 999. 72. Sanderson, J.P.; Naisbitt, D.J.; Farrell, J.; Ashby, C.A.; Tucker, M. J.; Rieder, M. J.; Pirmohamed, M.; Clarke, S.E.; Park, K.; J. Immunol. 2007, 178, 5533. 73. Fukuto, T. R.; Environ. Health Perspect. 1990, 87, 245. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access