Artigo
|
|
| Avaliação da eficiência de degradação de hidrocarbonetos aromáticos por bactérias provenientes de estação de tratamento de efluente de refinaria de petróleo Evaluation of the efficiency of deterioration of aromatic hydrocarbons by bacteria from wastewater treatment plant of oil refinery |
|
Fernanda R. PinhatiI,*; Eduardo M. Del AguilaII; Ana Paula R. TôrresIII; Maíra P. de SousaIII; Vânia Maria J. SantiagoIII; Joab T. SilvaII,+; Vânia Margaret F. PaschoalinII
IDepartamento de Química e Ambiental, Universidade do Estado do Rio de Janeiro, Faculdade de Tecnologia, Avenida Presidente Dutra, km 298, Resende - RJ, Brasil Recebido em 06/11/2013 *e-mail: fernandaromanholi@yahoo.com.br Three bacterial strains were isolated from the activated sludge system of petroleum refinery wastewater, identified by partial sequencing of 16S rDNA, and classified as Acinetobacter genomospecies 3, Bacillus pumilus, and Bacillus flexus. The degradation efficiency of aromatic hydrocarbons was evaluated by gas chromatography with a flame ionization detector. In a mineral medium containing anthracene and phenanthrene and the consortium of microorganisms, the removal efficiency was 96% and 99%, respectively, after 30 days. The good rate of hydrocarbon degradation proves the operational efficiency of the microbial consortium in treating effluents containing these compounds. INTRODUÇAO A água é reconhecida como um dos bens naturais mais importantes do planeta. Devido à crescente degradaçao dos corpos d'água, as preocupaçoes com o seu uso e destino têm mobilizado pessoas de várias áreas do conhecimento visando sua preservaçao.1 A água, muitas vezes, considerada como recurso natural renovável e infinito, é um recurso ambiental limitador do desenvolvimento sustentável. Os grandes problemas de escassez sao relacionados, muitas vezes, a utilizaçao em quantidades superiores ao volume disponível, seja na utilizaçao por indústrias, agricultura ou até mesmo devido ao aumento populacional.2 Houve avanços em relaçao à conservaçao da qualidade das águas superficiais e subterrâneas. O interesse dos setores público e privado por assuntos ambientais resultou na criaçao de várias organizaçoes governamentais e nao governamentais que hoje debatem, estabelecem normas e discutem práticas de minimizaçao e remediaçao de substâncias químicas potencialmente poluentes.3 O interesse da comunidade acadêmica e a criaçao de órgaos de proteçao ambiental, como a Environmental Protection Agency (EPA), nos Estados Unidos, e o Conselho Nacional do Meio Ambiente (CONAMA), no Brasil, promoveram um crescimento de pesquisas envolvendo monitoramento de contaminantes orgânicos em diversas áreas ambientais.4 Muitos produtos orgânicos sao descartados diariamente nos corpos hídricos, provenientes de esgotos domésticos e da atividade industrial (como, siderúrgicas, indústrias farmacêuticas e automobilísticas e refinarias de petróleo). As atividades industriais sao as que mais contribuem para a contaminaçao ambiental, uma vez que empregam grandes volumes de água e, consequentemente, geram grandes quantidades de efluentes líquidos contendo espécies tóxicas ou de degradaçao lenta e/ou difícil. Estes efluentes sao descartados, principalmente, em rios e mananciais de uso múltiplo, que tem como principal e mais nobre uso o consumo humano.5 O petróleo representa a principal fonte de combustível da humanidade e as operaçoes de exploraçao, transporte, refino e distribuiçao representam potenciais fontes de poluiçao ambiental.6 Sendo um dos setores industriais para o qual o enquadramento às normas ambientais se torna mais urgente. A indústria do petróleo tem nos seus sistemas produtivos vários processos nos quais as correntes de efluentes hídricos contêm altas quantidades de compostos tóxicos, que provocam danos ao meio ambiente. Assim, é necessário buscar formas de diminuir a presença destas substâncias nos efluentes da indústria de petróleo ou desenvolver processos que permitam a remoçao destas moléculas nos efluentes. A contaminaçao da água por hidrocarbonetos policíclicos aromáticos (HPAs) e os métodos para sua eliminaçao têm sido estudados em todo mundo pelo fato destas substâncias serem contaminantes onipresentes, originados, principalmente, nos processos de refino de petróleo.7 Para a remoçao de hidrocarbonetos aromáticos de águas e efluentes têm sido usados processos físico-químicos, como auto e fotoxidaçao, ozonizaçao, adsorçao e processos biológicos.8 Entretanto, a remediaçao natural é a forma mais barata de atenuaçao e vem sendo desenvolvida como tecnologia de recuperaçao de áreas impactadas. Esta técnica baseia-se em mecanismos envolvendo processos físicos, químicos e biológicos sem a intervençao humana, de tal maneira a reduzir a concentraçao dos contaminantes.9 Entre as técnicas de remediaçao natural, a biorremediaçao vem sendo bastante usada na recuperaçao de ambientes impactados por hidrocarbonetos de petróleo por utilizar o potencial metabólico de microrganismos e diminuir o caráter tóxico dos compostos contaminantes.10 A maioria dos microrganismos nao possui a capacidade de degradar hidrocarbonetos ou hidrocarbonetos aromáticos como fonte de carbono e energia, justificando a necessidade de se isolar e selecionar microrganismos degradadores de HPAs.11 O isolamento de linhagens puras de bactérias e fungos capazes de metabolizar hidrocarbonetos policíclicos aromáticos vem sendo realizado desde a década de 1950, mas somente nos últimos anos tem sido dada atençao ao estudo de consórcios microbianos como agentes degradadores de HPAs, que têm se mostrado mais efetivo na degradaçao destes compostos do que as linhagens puras. A maior capacidade de degradaçao dos consórcios microbianos aumenta a possibilidade de elaboraçao de processos de biorremediaçao mais efetivos, com a eliminaçao total dos contaminantes do ambiente. A alta dependência humana em relaçao à água e sua relativa disponibilidade fazem com que seja necessário evitar tanto o seu gasto excessivo quanto a contaminaçao dos corpos hídricos. Assim, é necessário desenvolver ferramentas e tecnologias que possibilitem uma melhor compreensao dos mecanismos de degradaçao dos principais compostos tóxicos e a reduçao da contaminaçao da água, minimizando os impactos ambientais, com uso racional e reutilizaçao de água residual, sempre que possível. Neste estudo, três cepas de bactérias isoladas de lodo biológico coletado a partir de uma estaçao de tratamento de efluentes de uma refinaria de petróleo foram identificadas. Após o sequenciamento parcial do gene rDNA 16S, avaliou-se a eficiência de degradaçao de antraceno, fenantreno e naftaleno como uma fonte única de carbono por meio de cromatografia gasosa com detector de ionizaçao de chama (CG-DIC).
PARTE EXPERIMENTAL Isolamento de bactérias O lodo biológico da refinaria de petróleo foi coletado do fundo do decantador. Uma amostra de 10 g deste lodo biológico foi centrifugada a 10.000 g por 15 minutos. O sobrenadante foi coletado e esterilizado por autoclavaçao a 121 ºC por 1,5 atm por 15 minutos para ser usado como meio de crescimento. Foram reservados 10 g de sedimento para serem usados como inóculo. O sobrenadante esterilizado (100 mL) foi enriquecido com naftaleno, fenantreno ou antraceno na concentraçao final de 250 mg L-1. Os frascos foram inoculados com o sedimento e incubados a 37 ºC com agitaçao de 250 rpm por 4 semanas. As culturas foram, entao, plaqueadas em meio mineral (MM) contendo (g L-1) 0,04 CaCl2 2.H2O; 0,1 KH2PO4; 0,8 NaCl; 1,0 NH4Cl; 0,2 MgSO4 7.H2O; 0,1 KCl, e (mg L-1) 0,1 CoCl2 6.H2O; 0,425 MnCl2 4.H2O; 0,05 ZnCl2; 0,015 CuSO4 5.H2O; 0,01 NiCl2 6.H2O; 0,01 Na2MoO4 2.H2O; 0,01 Na2SeO4 2.H2O, pH 7,0 ajustado com HCl e enriquecido com naftaleno, fenantreno ou antraceno na concentraçao (250 mg L-1), para isolamento de colônias. Foram isoladas três colônias, uma de cada meio seletivo contendo antraceno, fenantreno ou naftaleno, selecionadas apenas por diferenças morfológicas. Amplificaçao do gene 16S rDNA e análise filogenética Os microrganismos isolados foram cultivados em 10 mL de meio LB líquido (triptona 1%, NaCl 1% e extrato de levedura 0,5%) a 37 ºC, 250 rpm por 18h, como descrito em Sambrook et al.12 A extraçao do DNA realizada foi como descrita por Leao et al.13 O DNA total dos microrganismos isolados do lodo biológico foram amplificados por PCR utilizando o kit MicroSeq® 16S rDNA Full Gene (Applied Biosystem). A reaçao foi realizada em termociclador Veriti 60-well Thermal Cycler (Applied Biosystem), com volume final de 30 µL, contendo 10 ng de DNA e 15 µL da mistura de primers do kit MicroSeq® 16S rDNA Full Gene (Applied Biosystem). A reaçao foi realizada sob as seguintes condiçoes: 95 ºC por 10 min, 25 ciclos de amplificaçao a 95 ºC/30 s, 60 ºC/30 s e 72 ºC/45 s e um ciclo de extensao final de 10 min a 72 ºC. Os produtos amplificados foram analisados em gel de agarose 1,2% em tampao TAE 1X (40 mM Tris; 1mM EDTA, 20 mM ácido acético) a 100 V por 1 h. Após eletroforese, o gel foi corado em soluçao de Gelred (Biotium) por 30 min e as bandas foram visualizadas em fotodocumentador MiniLumi (DNS Bio Imaging Systems). Os fragmentos amplificados foram purificados e utilizados na reaçao de sequenciamento. A reaçao de sequenciamento foi conduzida no mesmo termociclador utilizando 7 µL do produto de PCR purificado e 13 µL do BigDye Terminator v1. 1 Cycle Sequencing (Applied Biosystems). A mistura foi submetida a 25 ciclos de temperatura a 96 ºC/10 s, 50 ºC/5 s e 60 ºC/4 min. Finalizada a reaçao, o produto foi purificado utilizando o Kit BigDye X Terminator (Applied Biosystem), conforme as recomendaçoes do fabricante. Os produtos fluorescentes foram separados em capilar de 50 cm (3130/3100-AvantTM Genetic Analyzer Capillary Array, 50 cm) preenchido com polímero POP 7 (POP-7TM Polymer for 3130/3130xl Genetic Analyzers). Os dados foram coletados pelo software Data Collection v1. 0.1 (Applied Biosystems) e as sequências foward e reverse obtidas foram alinhadas para reconstruçao da sequência completa da regiao V3 do gene 16S rDNA de cada bactéria utilizando o programa Sequence Analysis v.5.2 (Applied Biosystem). As sequências obtidas foram comparadas com uma biblioteca de sequências microbianas pelo software MicroSEQ System® (Applied Biosystem) que forneceu como resultado uma lista de espécies geneticamente relacionadas e uma árvore filogenética apresentando as relaçoes entre as espécies. Remoçao de HPAs do meio mineral (MM) A remoçao de HPAs pelas cepas isoladas, individualmente ou em consórcio, foi medida por CG-DIC. Porçoes de 50,0 mL de MM contendo 250 mg L-1 de cada HPA foram inoculados com 3 x 107 células mL-1 e incubou-se a 30 ºC por 30 dias em agitador rotativo (150 rpm). A perda por volatilizaçao dos HPAs também foi avaliada por CG-DIC. Frascos contendo apenas 250 mg L-1 de cada HPA em MM (50 mL) foram incubados nas mesmas condiçoes experimentais citadas acima. Nos tempos 0, 15 e 30 dias foram recolhidas amostras de 10 mL dos frascos testes e controle por centrifugaçao a 5.000 g durante 10 min e filtrada através de filtros de membrana (poros de 0,22 µm - Millipore). Os filtrados obtidos foram entao concentrados em corrente de gás hélio (grau analítico), em temperatura ambiente, até o volume de aproximadamente 2,0 mL. Os HPAs foram extraídos destas amostras com igual volume de diclorometano (grau HPLC). Após vigorosa agitaçao em vórtex, a fase orgânica foi recolhida e a fase aquosa foi extraída uma segunda vez com diclorometano. As fases orgânicas foram reunidas e armazenadas em frasco de vidro âmbar com tampa de teflon a -80 ºC, até utilizaçao. Todos os experimentos foram realizados em duplicata. Análise CG-DIC Análise de HPA foi realizada em cromatógrafo gasoso Shimadzu GC-2010 (Shimadzu Corporation, Japao), com detector de ionizaçao de chama, equipado com coluna capilar HP-5MS (30 m x 0,25 mm x 0,25 µm), sendo usado hélio como gás de arraste (fluxo de 40 mL min-1). Após a injeçao da amostra (1 µL), a coluna foi aquecida durante 3 min a 160 ºC, em seguida aumentada para 160 ºC e 300 ºC com uma rampa de 6 ºC min-1, e mantida durante 10 min. As temperaturas da porta de injeçao e detector de ionizaçao de chama foram de 300 ºC e 325 ºC, respectivamente. A soluçao estoque padrao certificado (Ultra Scientific) contendo HPAs (acenafteno, acenaftileno, antraceno, benzo [a] antraceno, benzo [a] pireno, benzo [b] fluoranteno, benzo [g, h, i] perileno, benzo [k] fluoranteno, criseno, dibenzo [a, h] antraceno, fluoranteno, fluoreno, indeno [1,2,3 -cd] pireno, naftaleno, fenantreno e pireno) na concentraçao individual de 2.000 µg mL-1 foi diluída em hexano, para se alcançar as concentraçoes de 50, 100, 150, 200, 250 e 300 µg mL-1 e injetadas no cromatógrafo a gás. As curvas de calibraçao com coeficientes de determinaçao (r2) de 0,9979, 0,9985 e 0,9988 foram obtidas para antraceno, fenantreno e naftaleno, respectivamente.
RESULTADOS E DISCUSSAO Identificaçao das bactérias isoladas Três microrganismos foram isolados a partir de lodo biológico coletado na estaçao de tratamento de efluentes de refinaria de petróleo por meio da técnica de enriquecimento usando antraceno, fenantreno ou naftaleno como única fonte de carbono e energia. Após a extraçao e amplificaçao parcial do gene 16S rDNA, as sequências foram reunidas e editadas usando o programa MicroSeq v.1.0 para a identificaçao dos isolados. Os resultados mostram que a sequência de 16S rDNA do isolado em antraceno apresentou alta homologia com a espécie Acinetobacter genomospecies 3 ATCC 17922 (99,97%), enquanto os isolados em fenantreno e naftaleno apresentaram 99,99% e 100% de similaridade com Bacillus pumilus ATCC 7061 e Bacillus flexus ATCC 49095, respectivamente. Com base nas sequências parciais do 16S rDNA foi construída a árvore filogenética (Figura 1).
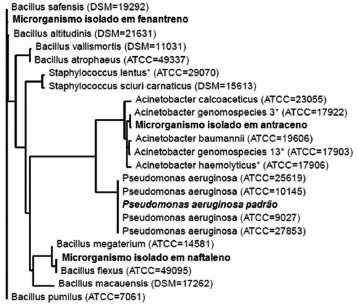 Figura 1. Arvore filogenética (UPGMA) mostrando as relaçoes entre as sequências dos genes 16S rDNA dos isolados de lodo biológico e das estirpes. Os fragmentos de DNA foram analisados em sequenciador automático ABI-3130 (Applied Biosystem), o alinhamento entre as sequências foi realizado pelo programa Sequence Analysis e alinhadas as bibliotecas microbianas do MicroSeq System (Applied Biosystem)
O dendrograma mostra as relaçoes entre as cepas isoladas em HPAs e as espécies mais proximamente relacionadas por comparaçao com a sequência da regiao hiper-variável V3 do 16S rDNA. Estas bactérias já foram isoladas e identificadas por outros pesquisadores em outras regioes impactadas com algum tipo de hidrocarboneto. Bactérias do gênero Acinetobacter parecem ser ubíquas em ecossistemas de lodo biológico14,15 e várias espécies de Acinetobacter que sao capazes de degradar ftalato de dibutila, foram isoladas a partir de lodo biológico de duas estaçoes de tratamento de esgoto no Japao.16 Estas determinam um importante papel no aumento da remoçao biológica de fósforo no tratamento de águas residuais,17 biorremediaçao de solo contaminado com n-alcanos,18 ambientes contaminados com combustíveis derivados de petróleo19,20 e sao descritas como capazes de utilizar HPA como única fonte de carbono.21-24 O gênero Bacillus compreende bactérias gram-positivas, em forma de bastonete, e formadoras de esporos sendo comumente encontradas no solo, em águas residuais e sedimentos de mangue.25,26 B. pumilus, juntamente com B. cereus e B. subtilis, constituem o principal grupo de comunidades bacterianas marinhas.27-29 B. flexus e B. pumilus sao bactérias halotolerantes,30 têm sido isoladas de lodo biológico utilizado para a produçao de hidrogênio,31 como bactérias endofíticas em raízes de banana32 e foram caracterizadas como microrganismos metabolizadores de pireno,33,34 tal como Bacillus subtilis.35 Assim, conforme mostrado pelos trabalhos, devido ao comprovado potencial de degradaçao de HPAs de tais bactérias, estas sao possíveis candidatas para o aprimoramento de sistemas de tratamento de efluentes de refinaria de petróleo. Eficiência de remoçao dos HPAs A perda dos HPAs do meio por volatilizaçao foi avaliada por CG-DIC. Os resultados mostraram uma diminuiçao na concentraçao de antraceno, fenantreno e naftaleno de 8,2%, 8,4% e 87,1% em 15 dias e 9,2%, 9,1% e 88,6%, respectivamente, em 30 dias. A estrutura química do naftaleno é a grande responsável por estes resultados. A pressao de vapor do naftaleno é 4,92 x 10-2 mmHg, enquanto que a do antraceno e fenantreno sao 2,00 x 10-4 e 6,80 x 10-4 mmHg, respectivamente. Visto que, a pressao de vapor diminui com o aumento da massa molecular do HPA, antraceno e fentantreno sao menos voláteis que o naftaleno.36 Devido à alta volatilidade do naftaleno, a avaliaçao do consumo deste composto ao longo do crescimento bacteriano tornou-se difícil de ser determinada com exatidao. Resultados similares foram observados por Valdman37 que estudou a cinética de biodegradaçao do naftaleno por Pseudomonas flourescens HK44 em meio mineral. Os resultados de biodegradaçao após 100 min de teste revelaram uma perda por volatilizaçao do naftaleno de 16,3%, em teste controle. Enquanto Loibner,38 ao investigar a taxa de biodegradaçao de HPAs em solo, com e sem bactérias, concluiu que a reduçao do naftaleno ocorreu por volatilizaçao, uma vez que as amostras de solo com microrganismos nativos e exógenos foram iguais aos obtidos para as amostras com solo esterilizado. Entretanto, o antraceno e fenantreno, que apresentam a mesma massa molecular e pressoes de vapor muito similares, apresentaram uma pequena taxa de volatilizaçao, e por isso puderam ter seu consumo estimado. No meio contendo um único tipo de HPA e um microrganismo, Acinetobacter gemonospecies 3 apresentou eficiência de degradaçao de antraceno de 9,9% e 38,7% em 15 e 30 dias, respectivamente. A eficiência de degradaçao por este mesmo microrganismo foi maior para o fenantreno, 35,2% e 53% em 15 e 30 dias, respectivamente. A menor taxa de biodegradaçao de antraceno foi obtida pelo isolado B. pumilus, que degradou apenas 3,7% e 9,4%, em 15 e 30 dias, respectivamente. Este isolado apresentou em média eficiência de remoçao de fenantreno de 36,7% ao longo de todo o experimento. Entretanto, o isolado B. flexus foi o que apresentou maior eficiência de biodegradaçao de antraceno em 30 dias, 69,5%. A biodegradaçao de fenantreno para este isolado foi de 34,3% em 30 dias (Figura 2).
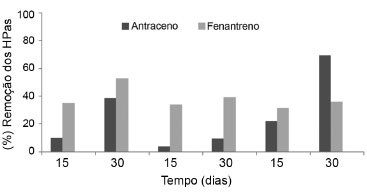 Figura 2. Biodegradaçao dos HPAs por cada isolado em meio mineral contendo apenas um tipo de HPA. A. genomospecies 3, B. pumilus e B. flexus foram cultivados em meio mineral contendo 250 mg L-1 de antraceno ou fenantreno. HPA residual foi determinado após 15 e 30 dias por CG-DIC. Percentagem de remoçao foi calculada considerando a quantidade do composto perdido por evaporaçao. Os HPAs foram estimados utilizando uma curva de calibraçao construída para cada HPA com coeficiente de determinaçao (r2) de 0,9979 e 0,9985 para o antraceno e fenantreno, respectivamente
Os resultados obtidos estao de acordo com aqueles apresentados por outro trabalho que também avaliou a eficiência de remoçao de HPAs por microrganismos isolados de diferentes ambientes contaminados com antraceno e fenantreno no Egito.39 As taxas de degradaçao de antraceno por Escherichia coli (EF105548), bactéria do solo (EF105549), Alcaligenes sp. (EF105546) e Thiobacter subterraneus (EF105547) foram 29%, 31%, 27% e 32%, enquanto a degradaçao do fenantreno foi de 43%, 48%, 34% e 40% , respectivamente, durante um período de 22 dias. Quando a mistura de HPA está na presença de apenas uma das bactérias, Acinetobacter gemonospecies 3 apresentou diminuiçao na taxa de biodegradaçao de antraceno e fenantreno (7,5% e 14% de degradaçao de antraceno, e 5% e 7% de degradaçao de fenantreno, em 15 e 30 dias, respectivamente) em relaçao ao teste anterior (Figura 3). Entretanto, observou-se um aumento na taxa de biodegradaçao de antraceno por B. pumilus (44,4% e 47,2% em 15 e 30 dias, respectivamente). A taxa de biodegradaçao de fenantreno para este isolado foi um pouco maior em relaçao ao experimento anterior (44%) em 15 dias, porém, a taxa de degradaçao diminui com 30 dias (26,7%). O isolado B. flexus apresentou uma reduçao na taxa de biodegradaçao de antraceno, sendo neste experimento de 39,6% em 30 dias e praticamente o mesmo valor de biodegradaçao de fenantreno em relaçao ao experimento anterior (33% em 30 dias) (Figura 3). Os melhores resultados de degradaçao de HPAs por B. pumilus obtidos no presente trabalho sao corroborados pelos resultados de Das et al.35 Estes investigaram e compararam a eficiência de utilizaçao de pireno pelas seguintes bactérias: B. subtilis DM-04, Pseudomonas aeruginosa mucoide (M) e nao-mucoide (NM). Estas foram isoladas a partir de uma amostra de solo contaminado com petróleo do nordeste da India. Tais microrganismos sao capazes de utilizar hidrocarbonetos como única fonte de carbono. O B. subtilis DM-04 exibiu maior utilizaçao e assimilaçao celular de pireno em comparaçao com P. aeruginosa M e NM, na presença de 100 mg L-1 de pireno por 96 h, com eficiências de remoçao de 48% e 32%, respectivamente. A eficiência de degradaçao dos HPAs nestes dois experimentos (um único microrganismo e um HPA em um único microrganismo em uma mistura dos três HPAs) foi muito semelhante. Esta similaridade na eficiência indica que estes microrganismos poderiam remover tais compostos policíclicos aromáticos mesmo em misturas complexas. Uma das hipóteses que pode explicar essa eficiência de remoçao de diferentes HPAs por uma mesma bactéria é a capacidade de utilizarem vias metabólicas comuns. As enzimas envolvidas na conversao do naftaleno a salicilato, por exemplo, podem degradar fenantreno e antraceno a 1-hidroxi-2-naftoato e 2-hidroxi-3-naftoato, respectivamente.40 Considerando que uma via metabólica pode estar envolvida na metabolizaçao de diferentes HPAs, Radwan e Sorkhoh41 isolaram duas cepas de Bacillus capazes de degradar 80-90% de petróleo bruto (5 g L-1) no prazo de cinco dias. A alta eficiência de degradaçao do consórcio de microrganismos no meio contendo os três tipos de HPAs em estudo, pode ser observada na Figura 4. Aproximadamente 55% do antraceno foram degradados em apenas 15 dias e 80%, em 30 dias. A melhor taxa de biodegradaçao foi de fenantreno, que em apenas 15 dias teve uma remoçao de 96,8% e uma eficiência de degradaçao de 99,6% em 30 dias. Embora muitas bactérias sejam capazes de metabolizar diferentes poluentes orgânicos, uma única bactéria pode nao possuir a capacidade enzimática para degradar todos, ou mesmo a maioria dos compostos orgânicos em um ambiente poluído. Comunidades microbianas mistas apresentam maior potencial de degradaçao, porque a informaçao genética de mais de um organismo pode ser necessária para degradar o complexo de misturas de compostos orgânicos presentes em áreas contaminadas. O potencial genético e determinados fatores ambientais, como temperatura, pH e fontes disponíveis de fósforo e nitrogênio parecem determinar a taxa e a extensao da degradaçao dos compostos poluentes, tal como foi observado por Samanta et al.42 em estudo no qual espécies foram capazes de utilizar até 100 diferentes compostos orgânicos como fonte de carbono. Para as Pseudomonas, por exemplo, o imenso potencial de degradaçao nao depende exclusivamente das enzimas catabólicas, mas também da sua capacidade de regulaçao metabólica. Em geral, os microrganismos preferem utilizar as fontes de carbono mais simples em relaçao aos compostos complexos quando ambos estao presentes no ambiente. Portanto, a presença de fontes de carbono simples pode reduzir significativamente a eficiência de um microrganismo em relaçao a compostos complexos. Se um microrganismo é impedido de utilizar fontes de carbono simples, torna-se totalmente dependente de compostos complexos, aumentando a especificidade e eficiência de degradaçao dos compostos mais complexos.42 Alguns trabalhos destacam esta alta eficiência de degradaçao de compostos complexos, em pequenos intervalos de tempo, por alguns microrganismos, quando estes sao submetidos a altas concentraçoes de HPAs complexos e sao desprovidos de fontes de carbono simples.43-45 A variaçao dos resultados obtidos por diferentes estudos pode ser interpretada com base na variaçao de "background" genético das linhagens bacterianas utilizadas, tempo de adaptaçao e condiçoes de incubaçao. Comparando-se os três experimentos realizados, observa-se que no experimento em que cada isolado foi inoculado em apenas um tipo de HPA, o isolado B. flexus foi o que apresentou melhor resultado de biodegradaçao de antraceno em 30 dias e A. genomospecies 3, a melhor eficiência para fenantreno em 30 dias (Figura 2). No segundo experimento, o isolado B. pumilus mostrou-se mais eficiente na degradaçao de antraceno e fenantreno, com médias muito próximas de degradaçao dos dois HPAs (Figura 3). Entretanto, a maior eficiência de biodegradaçao deve-se à presença do consórcio microbiano, que em apenas 15 dias degradou mais de 50% de cada HPA presente no meio mineral (Figura 4). Tal resultado foi similarmente demonstrado por Venkateswaran e Harayama,46 que ao utilizarem o enriquecimento de culturas, isolaram uma populaçao bacteriana capaz de degradar petróleo bruto. Verificaram que 28-51% da fraçao saturada e 0-18% da fraçao aromática presente foram biodegradadas por uma cultura mista (Acinetobacter sp., Aeromonas sp., Bacillus sp., Escherichia coli, Flavobacterium sp., Klebsiella cepacia, Micrococcus luteus, Moraxella phenylpiruvica, Nocardia sp., Ochrobacterium anthorpi, Pseudomonas aeruginosa, Pseudomonas sp., Proteus mirabilis, Vibrio sp., Rhodococcus sp., Streptomyces sp., Vibrio fisheri e Xanthomonas maltophilia). Contudo, quando foi utilizada cultura pura, nenhuma delas apresentou degradaçao superior à do consórcio.
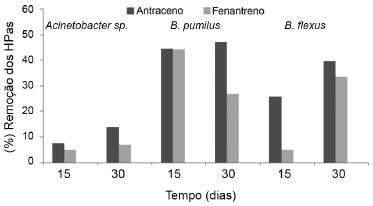 Figura 3. Biodegradaçao dos HPAs por cada isolado em meio mineral contendo os três HPAs. A. genomospecies 3, B. pumilus e B.flexus foram cultivados em meio mineral contendo 250 mg L-1 de antraceno ou fenantreno. HPA residual foi determinado após 15 e 30 dias por CG-DIC. Percentagem de remoçao foi calculada considerando a quantidade do composto perdido por evaporaçao. Os HPAs foram estimados utilizando uma curva de calibraçao construída para cada HPA com coeficiente de determinaçao (r2) de 0,9979 e 0,9985 para o antraceno e fenantreno, respectivamente
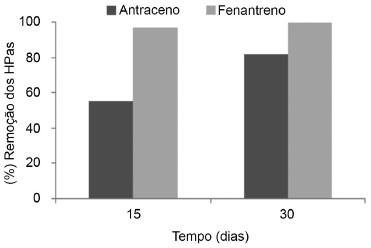 Figura 4. Biodegradaçao dos HPAs pelo consórcio microbiano em meio mineral contendo a mistura dos três HPAs. A. genomospecies 3, B. pumilus e B.flexus foram cultivados em meio mineral contendo 250 mg L-1 de antraceno ou fenantreno. HPA residual foi determinado após 15 e 30 dias por CG-DIC. Percentagem de remoçao foi calculada considerando a quantidade do composto perdido por evaporaçao. Os HPAs foram estimados utilizando uma curva de calibraçao construída para cada HPA com coeficiente de determinaçao (r2) de 0,9979 e 0,9985 para o antraceno e fenantreno, respectivamente
Uma melhor eficiência do consórcio microbiano para degradaçao de HPAs também foi relatada por Jacques et al.47 Um consórcio formado por seis bactérias isoladas de solo "landfarming" (Mycobacterium fortuitum, Bacillus cereus, Microbacterium sp., Gordonia polysioprenivorans, Microbacteriaceae bacterium e Naphthalene-utilizing bacterium, e um fungo identificado como Fusarium oxysporum) foi capaz de degradar 49% do antraceno e, 22 % de fenantreno e pireno em meio mineral (250 mg L-1 de cada HPA), no prazo de 48 dias. Entretanto, o consórcio isolado neste presente trabalho mostrou-se mais eficiente na remoçao de antraceno e fenantreno em um menor período de dias (Figura 4). A eficiência de degradaçao de HPAs pelo consórcio microbiano também tem sido atribuída à produçao e liberaçao para o meio de biossurfactantes produzido por alguns microrganismos. Estes compostos aumentam a superfície hidrofóbica de substratos insolúveis em água, aumentando sua biodisponibilidade, facilitando assim o crescimento de bactérias e maior taxa de biorremediaçao. Diversos trabalhos destacam que os biossurfactantes produzidos por diferentes espécies de Acinetobacter,48 A. calcoaceticus BU0349 e B. subtilis50 sao mais biodegradáveis e menos tóxicos em comparaçao com surfactantes químicos.51,52 Inclusive sao mais eficazes no aumento da solubilidade do fenantreno e benzo(a)pireno em comparaçao com surfactantes sintéticos, o que permitiu uma remoçao de aproximadamente 43% dos HPAs no meio.53 Uma vez que os resultados de eficiência de remoçao de HPAs obtidos sao consistentes com os trabalhos citados, tais espécies, sendo potenciais produtoras de biossurfactante, podem estar sob a açao destes compostos, o que resultaria na contribuiçao de remoçao destes. Para melhor compreensao da eficiência de remoçao de HPAs é de grande importância avaliar a presença e os efeitos de possível biossurfactante que esses organismos possam estar produzindo. O metabolismo bacteriano dos HPAs tem sido estudado há algum tempo, porém, a identificaçao dos genes e das enzimas envolvidas na rota de metabolizaçao necessita de maiores estudos. Alguns trabalhos destacam que estes genes podem estar localizados em plasmídeos.54-56 Pesquisadores identificaram espécies dos gêneros Pseudomonas,56 Bacillus57 e Acinetobacters,58 cujos plasmídeos transportam a informaçao genética essencial para o início da degradaçao dos HPAs. A presença destes genes associados com estes plasmídeos é ainda, em grande parte, desconhecida para a maioria das bactérias capazes de degradar HPA.59 A confirmaçao da presença do plasmídeo em uma bactéria potencialmente capaz de degradar HPAs torna-se uma ferramenta biológica de grande importância. Segundo Leahy e Colwell,55 os plasmídeos tem um importante papel na adaptaçao genética de microrganismos, visto que estes representam uma forma de DNA altamente móvel que pode ser transferida via conjugaçao ou transformaçao. Considerando que as bactérias isoladas tenham a informaçao genética em seus plasmídeos, e que os mesmos podem ser transferidos para outras bactérias, a inoculaçao destas bactérias selecionadas com uma cultura incapaz de degradar os HPAs, pode ser o passo inicial para potencializar o sistema biológico da estaçao no tratamento de efluentes contaminados. Neste contexto, é interessante investigar a presença de plasmídeos nestes isolados, a fim de pesquisar a presença dos genes catalisadores e a eficiência de açao destes em bactérias receptoras dos plasmídeos.
CONCLUSAO Três cepas de bactérias isoladas e identificadas mostraram potencial para degradaçao de HPAs. As maiores taxas médias de degradaçao de HPAs foram obtidos por B. flexus, que removeu 45% do antraceno após 15 dias e 47% do fenantreno após 30 dias de incubaçao. Taxas de degradaçao máximas foram obtidas quando um consórcio microbiano foi utilizado. Aproximadamente, 56% e 80% de biodegradaçao de antraceno em 15 e 30 dias, respectivamente e 96% e 99,6% de biodegradaçao de fenantreno em 15 e 30 dias, respectivamente. Visto que age sinergicamente, o consórcio de microrganismos apresenta larga capacidade enzimática capaz de degradar os poluentes. Associadas a esta capacidade, verificam-se a versatilidade e adaptabilidade de bactérias que contem plasmídeos catabólicos. A compreensao das características deste gene catabólico plasmidial é um assunto de investigaçao em curso.
AGRADECIMENTOS Os autores agradecem à Petrobras pelo apoio técnico e financeiro. LAABBM (Laboratório Avançado de Análises em Bioquímica e Biologia Molecular) foi apoiado por doaçoes da FAPERJ (Fundaçao de Amparo à Pesquisa do Estado do Rio de Janeiro) e CAPES (Coordenaçao de Aperfeiçoamento de Pessoal de Nível Superior). Em memória de J. T. Silva.
REFERENCIAS 1. Beeckman, G. B.; International Journal of Water Resources Development 1998, 14, 353. 2. Nascimento, J. F.; Dissertaçao de mestrado, Universidade Federal Fluminense, Rio de Janeiro, Brasil, 2004. 3. Hambright, K. D.; Parparov, A.; Berman, T.; Aquatic Conservation: Marine and Freshwater Ecosystems 2000, 10, 393. 4. Tirburtius, E. R. L.; Zamora, P. P.; Leal, E. S.; Quim. Nova 2004, 27, 441. 5. Azevedo, D. A.; Gerchon, E.; Reis, E. O.; J. Braz. Chem. Soc. 2004, 15, 165. 6. Geraldes-Kataoka, A. P. A.; Tese de doutorado, Universidade Estadual Paulista, Sao Paulo, Brasil, 2001. 7. Sabaté, J.; Bayona, J. M.; Solanas, A. M.; Chemosphere 2001, 44, 119. 8. Fan, S.; Zhao, L.; Lin, J.; Talanta 2007, 72, 1618. 9. EPA (U.S. Environmental Protection Agency). Guidance for Assessing Chemical Contaminant Data for Use in Fish Advisories. vol. 2: Risk Assessment and Fish Consumption Limits, Second edition. EPA 823-B-97-009, Office of Water, U.S. EPA, Washington, D.C., 1997. 10. Chaillan, F.; Le Flèche, A.; Bury, E.; Phantavong, Y.; Grimont, P.; Saliot, A.; Oudot, J.; Res. Microbiol. 2004, 155, 587. 11. Gaylarde, C. C.; Bellinaso, M. L.; Manfio, G. P.; Biotecnologia Ciência & Desenvolvimento 2005, 34, 36. 12. Sambroock, J.; Fritsch, E. F.; Maniatins, T.; Molecular cloning: a laboratory manual, 3th ed., Cold Spring Harbor Laboratory Press: Huntington, 2001. 13. Leao, S.; Martins, A.; Mejia, G. I.; Palomino, J. C.; Robledo, J.; Telles, M. A. S.; Portaeles, F.; Practical handbook for the phenotypic and genotipic identification of mycobacteria, 2004. 14. Knight, G. C., Seviour, E. M., Seviour, R. J.; Water Res. 1995, 29, 2085. 15. Carr, E.; Eason, H.; Feng, S.; Hoogenraad, A.; Croome, R.; Soddell, J.; Lindrea, K.; Seviour, R.; Appl. Microbiol. 2001, 90, 309. 16. Ogawa, G.; Ishida, M.; Urano, N.; Appl. Environ. Microbiol. 2009, 55, 261. 17. Wagner, M.; Erhart, R.; Manz, W.; Amann, R.; Lemmer, H.; Wedi, D.; Schleifer, K.; Appl. Environ. Microbiol. 1994, 60, 792. 18. Rosenberg, E.; Zuckerberg, A.; Rubinovitz, C.; Gutnick, D. L.; Appl. Environ. Microbiol. 1979, 37, 402. 19. Ishige, T.; Tani, A.; Sakai, Y.; Kato, N.; Appl. Environ. Microbiol. 2000, 66, 3481. 20. Gallego, J. L. R.; Loredo, J.; Llamas, J. F.; Vazquez, F.; Sanchez, J.; Biodegradation 2001, 12, 325. 21. Caposio, P.; Pessione, E.; Giuffrida, G.; Conti, A.; Landolfo, S.; Giunta, C.; Gribaudo, G; Res. Microbiol. 2002, 153, 69. 22. Gao, J.; Ellis, L. B. M.; Wackett, L. P.; Nucleic Acids Res. 2006, 38, 488. 23. Thangaraj, K.; Kapley, A.; Purohit, H. J.; Bioresour. Technol. 2008, 99, 2488. 24. Zhao, Z. Y.; Wong, J. W. C.; Environ. Technol. 2009, 30, 291. 25. Chang, B. V.; Chang, I. T.; Yuan, S. V.; J. Environ. Sci. Health, Part A: Environ. Sci. Eng. 2008, 43, 233. 26. Naggar, A. Y. E.; Kamel, M. M.; Aladly, A. A.; Ismail, N. A.; J. Am. Sci. 2010, 6, 661. 27. Ruger, H. J.; Mar. Ecol.: Prog. Ser. 1989, 57, 45. 28. Ortigosa, M.; Garay, E.; Pujalte, M. J.; Microbiologia 1997, 13, 453. 29. Ivanova, E. P.; Vysotskii, M. V.; Svetashev, V. I.; Nedashkovskaya, O. I.; Gorshkova, N. M.; Mikhailov, V. V.; Yumoto, N.; Shigeri, Y.; Taguchi, T., Yoshikawa, S.; Int. Microbiol. 1999, 2, 267. 30. Suresh, K.; Prabagaran, S. R.; Sengupta, S.; Shivaji; S.; Int. J. Syst. Evol. Microbiol. 2004, 54, 1369. 31. Lee, J. H.; Lee, D. G.; Park, J. I.; Kim, J. Y.; Korean J. Chem. Eng. 2010, 27, 187. 32. Souza, S. A.; Dissertaçao de mestrado, Universidade Estadual de Montes Claros, Minas Gerais, Brasil, 2011. 33. Khanna, P.; Tese de doutorado, Thapar University of India, India, 2011. 34. Khanna, P.; Goyal, D.; Khanna, S.; Polycyclic Aromat. Compd. 2011, 31, 1. 35. Das, K.; Mukherjee, A. K.; J. Appl. Microbiol. 2007, 102, 195. 36. Baird, C.; Cann, M.; Química Ambiental, 2ª ed., Editora Bookman: Porto Alegre, 2002, cap. 12. 37. Valdman, E.; Tese de doutorado, Universidade Federal do Rio de Janeiro, Rio de Janeiro, Brasil, 2004. 38. Loibner, A. P.; J. Appl. Microbiol. 2003, 2, 15. 39. Abd-Elsalam, H. E.; Hafez, E. E.; Hussain, A. A.; Ali, A. G.; El-Hanafy, A. A.; American-Eurasian J. Agric. Environ. Sci. 2009, 5, 31. 40. Menn, F. M.; Applegate, B. M.; Sayler, G. S.; Appl. Environ. Microbiol. 1993, 59, 6. 41. Radwan, S. S.; Sorkhoh, N. A.; Adv. Appl. Microbiol. 1993, 39, 29. 42. Samanta, S. K.; Bhushan, B.; Jain, R. K.; Appl. Microbiol. Biotechnol. 2001, 55, 627. 43. Liu, Y.; Zhang, J.; Zhang, Z.; Biodegradation 2004, 15, 205. 44. Zhang, H.; Kallimanis, A.; Koukkou, A. I.; Drainas, C.; Appl. Microbiol. Biotechnol. 2004, 65, 124. 45. Coral, G.; Karagoz, K.; Ann. Microbiol. (Heidelberg, Ger.) 2005, 55, 255. 46. Venkateswaran, K.; Harayama, S.; Can. J. Microbiol. 1995, 41, 767. 47. Jacques, R. J. S.; Santos, E. C.; Bento, F. M.; Peralba, M. C. R.; Selbach, P. A.; Sá, E. L. S.; Camargo, F. A. O.; Int. Biodeterior. Biodegrad. 2005, 56, 143. 48. Barkay, T.; Navon-Venezia, S.; Ron, E. Z.; Rosenberg, E.; Appl. Environ. Microbiol. 1999, 65, 2697. 49. Wong, J. W. C.; Zhao, Z.; Zheng, G.; Proceedings of the Annual International Conference on Soils, Sediments, Water and Energy 2010, 15, 36. 50. Rosenberg, E.; Ron, E. Z.; Curr. Opin. Biotechnol. 1997, 8, 313. 51. Zhao, Z. Y.; Wong, J. W. C.; Environ. Technol. 2009, 30, 291. 52. Romine, M. F.; Stillwell, L. C.; Wong, K. K.; Thurston, S. J.; Sisk, E. C.; Sensen, C.; Gaasterland, T.; Fredrickson, J. K.; Saffer, J. D.; J. Bacteriol. 1990, 181, 5. 53. Greated, A.; Lambertsen, L.; Williams, P. A.; Thomas, C. M.; Environ. Microbiol. 2002, 4, 12. 54. Maeda, K.; Nojiri, H.; Shintani, M.; Yoshida, T.; Habe, H.; Omori, T.; J. Mol. Biol. 2003, 326, 1. 55. Leahy, J. G.; Colwell, R. R.; Microbiol. Rev. 1990, 5, 303. 56. Dunn, N. W.; Gunsalus, I. C.; J. Bacteriol. 1973, 114, 974. 57. Pannucci, J.; Okinaka, R. T.; Sabin, R.; Kuske, C. R.; J. Bacteriol. 2002, 184, 1. 58. The Biology of Acinetobacter; Towner, K. J.; Bergogne-Bérézin, E.; Fewson, C. A, eds.; F.E.M.S. Symposium No. 57, Paris, 1991. 59. Frantz, B.; Chakrabarty, A. M.; The biology of Pseudomonas 1996, 10, 295. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access





