Artigo
|
|
| Seleção polifásica de microrganismos produtores de polihidroxialcanoatos Poliphasic screening of polyhydroxyalkanoate-producing microorganisms |
|
Amanda L. S. Silva; Elane C. L. dos Santos; Italo A. P. dos Santos; Ana M. Q. López*
Departamento de Química, Instituto de Química e Biotecnologia, Universidade Federal de Alagoas, 57072-900 Maceió - AL, Brasil Recebido em 11/11/2015 *e-mail: amql@qui.ufal.br Polyhydroxyalkanoates (PHAs) are fully biodegradable biopolyesters produced by many prokaryotes and accumulated as insoluble cytoplasmatic inclusions. The detection of these intracellular granule is usually provided using lipofilic azodyes, which are not specific. Another way to screen PHA-producing bacteria is through culture-independent molecular techniques such Polymerase Chain Reaction (PCR), and the search for new PHA-producer strains is essential to reduce the cost at industrial level. The application of these both methods is desirable. In the present study, 24 bacteria isolated from soil of the Atlantic forest in Maceió (AL, Brazil) and from agri-industrial sludge (Coruripe-AL, Brazil) were studied regarding to their capacity of growing in mineral salt medium, as indicative of PHA synthesis. All strains were submitted to biochemical characterization, whilst PCR has proved that isolates BMA-05, BMA-10, BMA-13 and BDL-07 has the gene phaC, which encodes a PHA synthase, the key for PHA synthesis. The amplification and sequencing of their 16S r-DNA region was able to identify these bacteria respectively as Pseudomonas fluorescens, Enterobacter aerogenes, Klebsiella oxytoca and Bacillus pumilus. The same minimal medium supplement with peptone demonstrated to induce growing of those strains. INTRODUÇAO Para ser considerado um plástico degradável, a molécula deve apresentar cadeia com ou sem ramificaçao ou com grupos funcionais éster, amida ou acetal.1 Comumente, tais características nao sao atendidas pelos polímeros sintéticos, e sua degradaçao no ambiente dá-se muito lentamente. Por outro lado, os plásticos denominados biodegradáveis possuem os requisitos apresentados para tal e por isso sao rapidamente decompostos por muitas espécies bacterianas e microalgas. Conforme descrito por Khanna e Srivastava,2 distribuem-se em três categorias: a) a dos polímeros sintéticos suscetíveis a degradaçao e/ou ataque enzimático de microrganismos, mas que nao atendem a todas as características necessárias para exploraçao comercial; b) a dos bioplásticos baseados no polissacarídeo amido, que sao parcialmente degradáveis, pois os microrganismos degradam o amido mas nao o polímero sintético que faz parte da composiçao desses produtos; c) a dos polihidroxialcanoatos (PHAs) produzidos por diversos microrganismos e armazenados intracelularmente como inclusoes amorfas, mas que sao totalmente degradáveis por organismos vivos. Nesse sentido, desde a década de 90 do século passado, o Brasil vem dedicando um considerável esforço para o desenvolvimento de processos visando a geraçao em larga escala e utilizaçao de polihidroxialcanoatos (PHAs) em produtos.3 Cerca de 150 monômeros diferentes já foram identificados como constituintes de PHAs produzidos por bactérias a partir de diversas fontes de carbono.4 Vale ressaltar que, na maioria das vezes, esse polímero biodegradável é produzido em condiçoes nao balanceadas pela limitaçao de algum nutriente (como fósforo e nitrogênio) e rico na fonte de carbono.5 Cupriavidus necator (também conhecida como Wautersia eutropha, Ralstonia eutropha e Alcaligenes eutrophus) é a espécie bacteriana mais estudada na produçao desses biopolímeros,6 mas a busca por outros isolados em amostras ambientais diversas que favoreçam o mesmo tem se intensificado. Do ponto de vista econômico, embora os PHAs apresentem vantagens ambientais sobre os plásticos de origem petroquímica, seu alto custo de produçao, em especial com utilizaçao de fontes de carbono puras, e também de metodologias de extraçao, ainda limita a aplicaçao comercial.4 Por isso, a busca de substratos ricos em carbono, de baixo custo, e que agreguem a remoçao de resíduos poluentes do ambiente, tem aumentado. Diversas pesquisas destacam o uso de melaço e caldo-de-cana. Outra opçao é o soro de leite, um substrato abundante em laticínios de produçao de queijo e caseína,5 geralmente descartado de modo a poluir o ambiente ou utilizado como suplemento alimentar ou bebida láctea de baixo valor no mercado. Este trabalho visou a seleçao de bactérias com o gene habilitador da produçao de polihidroxialcanoatos entre isolados obtidos em amostras de solo (horizonte A de regiao selvagem de Mata Atlântica) e de lodo de esgoto da Estaçao de Tratamento de Efluentes (ETE) de indústria sucroalcooleira, coletados em Coruripe, litoral Sul de Alagoas (Brasil).
PARTE EXPERIMENTAL Coleta de amostras de solo e lodo e isolamento de bactérias A coleta de solo de Mata Atlântica (Coruripe-AL) foi realizada num local onde nao há interferência antrópica (10°17'07'S, 36°21'07"W). A camada superior do horizonte "O" foi removida com auxílio de ferramentas de jardinagem e amostras do solo do horizonte "A" foram coletadas, depositadas no interior de tubos tipo Falcon previamente esterilizados e transferidas para caixa de isopor com gelo (≅ 4 °C) para o transporte ao laboratório de Bioquímica do Parasitismo e Microbiologia Ambiental (LBPMA) da Universidade Federal de Alagoas (UFAL), onde foram mantidas sob refrigeraçao (≅ 6 °C) até seu processamento. As amostras de lodo de lagoa de sedimentaçao da Estaçao de Tratamento de Efluentes (ETE) da "Usina Coruripe Açúcar e Alcool S. A.", localizada no mesmo município, também foram coletadas, acondicionadas em tubos tipo Falcon, preservadas e transportadas para o LBPMA conforme o descrito para amostras do solo. A partir daí, inoculou-se individualmente 4 g de cada amostra em meio mínimo mineral (40 mL) com a seguinte composiçao e posterior ajuste do pH para 7,0 (g L-1): (NH4)2SO4, 0,2; KH2PO4.7H2O, 13,3; MgSO4, 1,3; ácido cítrico, 1,7; soluçao de elementos traço esterilizada por filtraçao, 10 mL [FeSO4.7H2O, 10; ZnSO4.7H2O 2,25; CuSO4.5H2O, 1; MnSO4.5H2O, 0,5; CaCl2.2H2O, 2; Na2B4O7.10H2O, 0,23; (NH4)6Mo7O24, 0,1; 35 % HCl, 10 mL] e glicose, 20.7 Quando foi necessário utilizar esse meio sólido, acrescentaram-se 20 g L-1 de ágar. Após 72 h de incubaçao sob agitaçao orbital (120 rpm, 30 ± 1 °C), as culturas foram submetidas a diluiçoes seriadas e alíquotas dessas diluiçoes reinoculadas em meio sólido Agar Nutriente com 0,03 g L-1 do antifúngico fluconazol (Medley®).8 Características morfobioquímicas das bactérias isoladas Nos testes para a identificaçao presuntiva dos isolados bacterianos, foram realizadas investigaçao da motilidade, coloraçoes (Gram e verde malaquita) e testes para expressoes da catalase e oxidase.9 O sistema de galerias API® 20E foi utilizado de acordo as recomendaçoes do fabricante para avaliar a presença das seguintes enzimas/reaçoes: β-galactosidase, arginina dihidrolase, lisina descarboxilase, ornitina descarboxilase, utilizaçao de citrato, produçao de H2S, urease, expressao da triptofanase, produçao de indol, produçao de acetoína, gelatinase, fermentaçao/oxidaçao de glucose, manitol, inositol, sorbitol, ramnose, sacarose, melibiose, amigdalina, arabinose, e produçao de citocromo-oxidase. Os microrganismos fermentadores da glicose foram cultivados em meio Vermelho de Metila-Voges Proskauer para determinar a via pela qual esse processo ocorre.8 Seleçao de bactérias produtoras de PHAs Turbidez das culturas Cada bactéria isolada foi reinoculada em meios líquidos contendo a mesma composiçao daquele descrito para isolamento, diversificando-se, entretanto, a fonte de carbono original (glicose), a qual foi substituída por óleo de soja, glicerol e soro de leite (2%, em todos os casos). Em seguida, as culturas foram incubadas nas mesmas condiçoes anteriormente descritas e a turbidez, usada como um reflexo da produçao de PHAs, foi registrada atribuindo-se a legenda: + (pouco turvo); ++ (turbidez mediana); +++ (muito turvo), por inspeçao visual após 72 h de cultivo (30 ± 1°C).7 Coloraçao das colônias Duas metodologias de coloraçao das colônias microbianas produtoras de polihidroxialcanoatos foram utilizadas com o meio mineral sólido descrito para o isolamento das bactérias. Na primeira, uma soluçao do corante preto de Sudao (0,3 g em 100 mL de etanol 70%) foi aplicada diretamente a colônias obtidas após 72 h de cultivo (30 ± 1 °C). Após 20 min, efetuou-se a lavagem das colônias com etanol 100% e as colônias que retiveram o corante e permaneceram escurecidas representaram resultado positivo. Na segunda estratégia de seleçao, um volume de 0,5 µL de soluçao do corante azul do Nilo (1% em DMSO) foi adicionado a um litro de meio mínimo antes da esterilizaçao. Após utilizaçao desse meio para inoculaçao e a incubaçao conforme condiçoes descritas na primeira metodologia, as colônias com brilho alaranjado após excitaçao luminosa de luz ultravioleta (UV-longo, 360 nm) foram considerados positivas para a produçao de bioplástico. Detecçao do gene codante da phaC sintase Após 24 h de incubaçao dos isolados bacterianos, sob agitaçao orbital (120 rpm, 30 ± 1 °C), em caldo nutriente, efetuou-se a extraçao de seu material genético por meio de kit comercial (Norgen Biotek).8 O par de oligonucleotídeos iniciadores (primers) G-D (GTGCCGCCSYRSATCAACAAGT) e G-2R (GTAGTTCCASAYCAGGTCGTT), desenhado por Romo et al.,10 foi sintetizado pela empresa GenOne. A amplificaçao por PCR seguiu o seguinte protocolo de reaçao (volume final de 25 µL): 100 µg de DNA, 10 ρmol de cada oligonucleotídeo iniciador, 0,8 mmol L-1 de dNTPs, 2,5 mmol L-1 de MgCl2, 2,5 ml de tampao 10X (Biosystems), 5 µg de BSA, DMSO e 1 U de Taq DNA Polimerase (Biosystems).11 O DNA amplificado foi visualizado com auxílio de transiluminador (UV emitido diretamente no gel de agarose corado com brometo de etídio). Identificaçao molecular (16S rDNA) das bactérias produtoras de PHA O material genético das bactérias previamente selecionadas como produtoras de PHAs foi novamente extraído, amplificado e visualizado conforme descrito no item anterior, exceto pelo fato de que nas reaçoes de PCR utilizou-se o par 356F/1064R como oligonucleotídeos iniciadores da amplificaçao do gene 16S r-DNA e nao houve acréscimo de DMSO.12 O amplicon de cada um dos isolados foi purificado por meio de kit comercial (Norgen Biotek) e, depois, sequenciado em ambas as direçoes pela Plataforma de Sequenciamento de DNA da Universidade Federal de Pernambuco. As sequências foram editadas, convertidas para o formato FASTA e comparadas com aquelas registradas para tal gene identificador nos bancos do NCBI (National Center for Biotechnological Information) e do RDP (Ribosomal Database Project). Tais sequências também foram depositadas no GenBank (NCBI). O dendograma foi criado no programa CLUSTALW.13 Cinética de crescimento das bactérias produtoras de PHA A fim de determinar qual a composiçao do meio de cultura mais favorável ao crescimento dos microrganismos produtores de PHAs obtidos neste trabalho, a composiçao do meio descrito anteriormente foi modificado quanto à variaçao da concentraçao de glicose e quanto à presença de peptona. As culturas receberam as seguintes codificaçoes: Meio 01 (meio mínimo convencional, com 20 g L-1 de glicose), Meio 02 (5 g L-1 de peptona), Meio 03 (2 g L-1 de glicose e 5 g L-1 de peptona), Meio 04 (controle; ausente de glicose e peptona), Meio 05 (20 g L-1 de glicose e 5 g L-1 de peptona). Culturas submersas de cada isolado em meio caldo nutriente com 24 h de incubaçao foram utilizadas para inocular os diferentes meios de cultura líquidos, distribuídos em volume de 10 mL em tubos tipo Falcon (50 mL), com alíquotas que no final rendessem 106 céls mL-1. Triplicatas das culturas foram incubadas (65 rpm, 30 ± 1 °C) por um período de 96 h e a cada intervalo de 24 h foram colhidas amostras para determinaçao de pH, biomassa seca, concentraçao de proteínas e glicídeos redutores totais.14 Todas as análises foram realizadas em triplicatas. Os resultados de cada parâmetro nos diferentes tempos foram comparados por meio de análise de variância (ANOVA) e suas médias, através do teste de Tukey, sempre ao nível de 5% de significância.
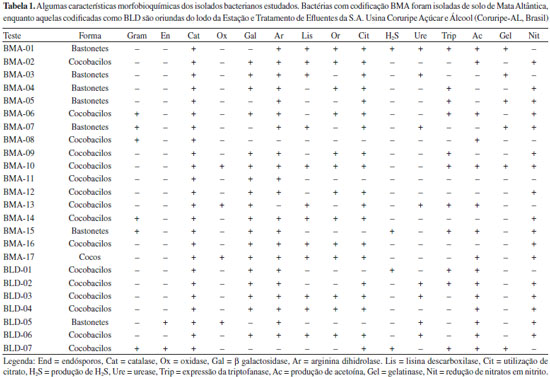
RESULTADOS E DISCUSSAO Perfil bioquímico dos microrganismos isolados Foram isoladas 24 bactérias, sendo 17 nas amostras de solo (horizonte A) da Mata Atlântica e sete nas de lodo de ETE de agroindústria de Coruripe-AL. O pequeno número de isolados pode ser explicado pela composiçao do meio de cultura que foi utilizado para o isolamento, isto é, limitado em nitrogênio [0,2 g L-1 de (NH4)2SO4]. As linhagens produtoras de PHAs sao classificadas em dois grupos principais, baseado nas condiçoes de cultivo que estimulam a produçao do polímero: há aquelas que necessitam da limitaçao de um elemento essencial como N, P, Mg, K, O ou S, e aquelas que produzem o polímero mesmo quando há abundância de todos os nutrientes. Porém, mesmo nesse último caso, as bactérias sintetizam maior quantidade de PHA quando o meio é limitante e, por isso, o meio mínimo limitado em nitrogênio é escolhido pelos pesquisadores quando buscam selecionar microrganismos autóctones produtores do polímero.15 A quase totalidade das bactérias utiliza compostos inorgânicos de nitrogênio, em especial sais de amônio e ocasionalmente nitratos (raramente nitritos), mas alguns isolados exigem fontes orgânicas, representadas por um número variável de aminoácidos. Assim, a ausência de fontes orgânicas impediu o crescimento de colônias representativas dos microbiomas presentes nas amostras, restringindo-se ao aparecimento somente daquelas de interesse, isto é, potencialmente produtoras de PHAs. Apesar de gêneros formadores de endósporos serem comumente encontrados no solo (como Bacillus, Clostridium, Sporosarcina e Streptomyces), nenhum dos 17 microrganismos isolados do solo de Mata Atlântica apresentou tal estrutura no interior celular após serem corados com verde malaquita. Por outro lado, das sete bactérias isoladas do lodo da ETE, duas (BLD05 e BLD07) sao Gram positivas formadora de endósporos. A catalase, enzima importante para as bactérias no combate ao excesso de radicais livres na célula, foi detectada em todos os microrganismos isolados. As características morfológicas e bioquímicas dos isolados obtidos neste trabalho estao expostas na Tabela 1.
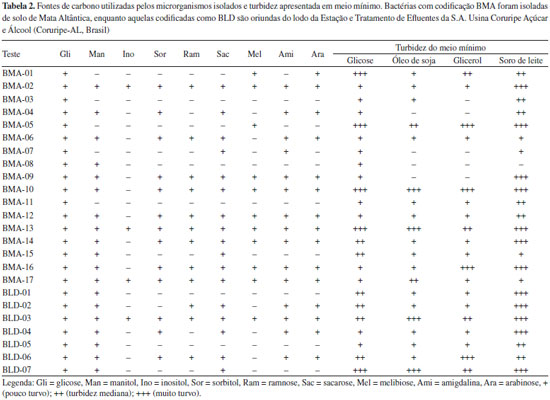
Seleçao dos produtores de polihidroxialcanoatos Fontes de carbono e turbidez em meio mínimo A procura por bactérias produtoras de PHAs deve levar em consideraçao o substrato disponibilizado a elas, visto que o mesmo determinará o tipo de biopolímero sintetizado e os custos do processo. Fontes mais puras podem aumentar em até 80% os gastos de produçao, mas em geral aumentam a produtividade do PHA.16,17 Por outro lado, pesquisas com C. necator cultivada em óleo de canola revelam alto rendimento de síntese de PHA (92%).16 Essa bactéria também é capaz de acumular maior quantidade de PHA em meio contendo metanol - uma das fontes de carbono mais baratas que existem - do que glicose.18 O butirato é outro substrato bastante promissor para ser utilizado na produçao de PHAs, já que a partir dele o isolado bacteriano pode conseguir elevado rendimento do biopolímero.19 A verificaçao de quais fontes de carbono sao utilizadas por cada microrganismo serve para direcionar a escolha do substrato para a produçao de PHAs em grandes volumes, isto é, cultura submersa em biorreatores.20 Todos os microrganismos obtidos no presente trabalho utilizam a glicose como fonte de carbono e a maioria utiliza também sacarose e manitol (Tabela 2). Para verificar a utilizaçao de outras fontes de carbono como óleo e soro de leite, os microrganismos foram cultivados em meio mínimo e a turbidez averiguada após 72 h. A turbidez do meio de cultura geralmente está relacionada com a concentraçao de células do microrganismo, mas no caso de cultivo em meio para síntese de PHAs, esta é relacionada com a quantidade intracelular do polímero, isto é, quanto mais turvo o meio, devido a opacidade da cultura presente, provavelmente houve mais síntese do bioplástico. Verificou-se, dessa forma, que a maioria dos isolados utilizam outros substratos além da glicose para crescer e provavelmente produzir PHAs. As bactérias BMA-05,10 e 13 e BLD-07 apresentaram maior turbidez em todos os meios testados e foram selecionadas para os testes de coloraçao de suas colônias com os corantes preto de Sudao e azul do Nilo, e para o rastreamento do gene phaC.
Coloraçao das colônias Os quatro microrganismos selecionados retiveram o preto de Sudao em suas colônias, mesmo após lavagem com álcool. Entretanto, apenas o isolado codificado como BMA-10 produziu fluorescência sob luz UV, embora com intensidade inferior à da bactéria de referência, Cupriavidus necator ATCC 17699. Alguns corantes sao muito utilizados para seleçao de microrganismos produtores de PHAs devido a sua capacidade de penetrar na membrana e corar grânulos desses polímeros.21 O preto de Sudao é um corante ligeiramente solúvel em solvente orgânico e insolúvel em água, capaz de conferir coloraçao negra azulada às estruturas hidrofóbicas visíveis em microscópio óptico. Quando em contato com as colônias, o corante é retido pelos microrganismos produtores de PHA, tornando-as azuladas.22 O uso do preto de Sudao antes da descoberta de outros corantes foi consideravelmente grande, porém, ele nao é específico para PHA, uma vez que é capaz de agir em todos materiais lipídicos presente na célula. Contudo, seu uso na identificaçao de microrganismos produtores é a etapa inicial de muitas pesquisas.23 O azul do Nilo, por sua vez, é um corante básico de oxazina, solúvel em água e álcool etílico. Em virtude da oxidaçao espontânea do azul do Nilo em soluçao aquosa, resultando na produçao de vermelho do Nilo, suspeita-se que este seja o responsável pela coloraçao e fluorescência dos grânulos de PHA nas células bacterianas. Esse corante possui propriedades de maior sensibilidade e seletividade para a identificaçao de grânulos intracelulares de PHA, pois as membranas celulares e outros componentes celulares lipídicos nao absorvem quantidades suficientes de corante para emitir fluorescência.24 Apesar da maior especificidade do azul do Nilo aos PHAs, Mesquita et al.25 compararam as imagens de células bacterianas coradas com preto de Sudao (obtidas em microscópio de campo pulsado) com as imagens obtidas de esfregaços corados com azul do Nilo (obtidas em microscópio de fluorescência) e constataram que nao somente a detecçao de microrganismos com a habilidade de acumular o polímero por meio de qualquer um desses corantes é igualmente eficiente, como também é confiável para estimar a concentraçao dos polihidroxialcanoatos presentes nas células. Detecçao do gene codante da phaC sintase As pesquisas mais recentes sobre PHAs abordam o estudo das enzimas que estao diretamente envolvidas na síntese de polihidroxialcanoatos. Existem quatro classes de PHA sintases, das quais destacam-se as PHA sintases da classe I, capazes de polimerizar unidades de hidroxiacil-CoA com cadeia curta (3-5 átomos de carbono) e as de classe II, específicas para monômeros de cadeia média (6-14 átomos de carbono).26 Independentemente de qual classe a PHA sintase de um determinado microrganismo pertença, todas elas possuem a subunidade PhaC, e, por isso, essa é a regiao do DNA escolhida para ser detectada pela técnica de PCR, quando se espera detectar molecularmente possíveis produtores do polímero em questao. Os quatro microrganismos avaliados apresentaram o amplicon esperado de 492 pb, sendo que os primers utilizados na reaçao de PCR hibridizam internamente o gene codante da PhaC sintase (classes I e II) de maneira universal.10 López et al.27 foram os primeiros pesquisadores a utilizar a técnica de PCR para identificar organismos que apresentassem o gene em questao, mas na ocasiao os primers foram baseados apenas no genoma de Cupriavidus necator. Com a popularizaçao dessa técnica molecular aliada à crescente disponibilidade de microrganismos sequenciados, diversos autores começaram a desenhar iniciadores com degeneraçao das suas bases em diferentes proporçoes. Sheu et al.8 foram alguns desses pesquisadores: eles elaboraram, com sucesso, um protocolo para detectar isolados bacterianos produtores de PHAs obtidos de amostras ambientais, por meio de colony PCR. As sequências altamente degeneradas propostas pelo grupo sao utilizados para detectar genes phaC das classes I e II. Até o presente momento, nao há primers universais para detectar genes phaC das classes III e/ou IV,28 embora existam aqueles que sao específicos para bactérias redutoras de sulfato e haloarchaea que codificam para a PhaC sintase de classe III.29 Shamala et al.30 publicaram um trabalho divulgando primers nao degenerados que delimitam o gene phaC específico da PhaC sintase IV, presente em Bacillus spp., porém os autores nao conseguiram amplificaçao desse gene em B. anthracis e B. thuringensis. O referido trabalho levou Solaiman e Ashby31 a sugerirem que a sintases de classe IV sao divididas em dois subgrupos: subgrupo B. megaterium (incluindo B. sphaerica, B. circulans e B. brevis) e o subgrupo B. cereus (incluindo B. anthracis e B. thuringensis). Sendo assim, isso significa que apesar da detecçao do gene nos isolados BMA-05, 19, 13 e BLD-07, nao é possível afirmar qual classe de phaC está presente em cada uma dessas bactérias. Identificaçao molecular (16S rDNA) das bactérias produtores de PHA Os primers utilizados nas reaçoes de PCR deste trabalho para amplificar a regiao 16S rDNA resultaram em fragmentos gênicos com cerca de 700 pb em todos os quatro isolados testados. A comparaçao entre as bases nucleotídicas obtidas e os bancos de dados permitiram identificar que os microrganismos produtores de PHAs pertencem ao domínio Bacteria, sendo um representante do filo Firmicutes (BLD-07) e os outros do filo Gammaproteobacteria. A análise do alinhamento múltiplo das sequências nucleotídicas mostraram que a regioes 16S rDNA sequenciadas dos isolados BMA-05, 10, 13 e BLD-07 possuem 99% de identidade com Pseudomonas fluorescens P10-1, Enterobacter aerogenes KCTC 2190, Klebsiella oxytoca KCTC 1686 e Bacillus pumilus SAFR-032, respectivamente (Figura 1). Os números de acesso no GenBank (NCBI) das sequências depositadas sao: KU555925 (Pseudomonas fluorescens BMA05), KU555926 (Enterobacter aerogenes BMA10), KU555927 (Klebsiella oxytoca BMA13) e KU555928 (Bacillus pumilus BLD-07).
 Figura 1. Dendograma obtido em decorrência do alinhamento múltiplo das sequências 16S rDNA dos isolados BMA-05, 10, 13 e BLD-07, os quais tiveram 99 % de identidade com as bactérias Pseudomonas fluorescens P10-1, Enterobacter aerogenes KCTC 2190, Klebsiella oxytoca KCTC 1686 e Bacillus pumilus SAFR-032, respectivamente
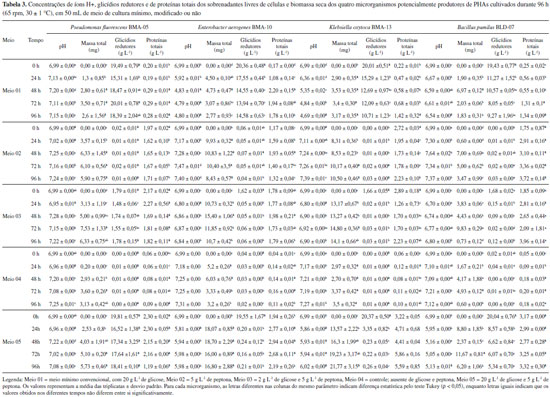
Conhecer a espécie precisa dos microrganismos isolados é essencial para direcionar futuros estudos para uma determinada finalidade. Dos quatro gêneros identificados no presente trabalho, Pseudomonas e Bacillus sao os mais largamente utilizados na produçao de PHAs. Enquanto o gênero Pseudomonas é conhecido por produzir PHAs contendo monômeros de cadeia média, algumas bactérias Gram-positivas, tais como espécies de Bacillus, Clostridium, Corynebacterium, Nocardia e Rhodococcus, têm sido estudadas tanto na produçao de PHB como também de copolímeros. O gênero Bacillus compreende mais de 50 espécies, cujo rendimento da produçao de PHAs varia de 11 a 69% (p/p de massa seca celular) e esse polímero biodegradável pode ser produzido em condiçoes de excesso ou limitantes de algum nutriente. Segundo Parvathi et al.,32 a espécie B. pumilus caracteriza-se por ser altamente resistente a condiçoes ambientais extremas, como a disponibilidade de baixo ou nenhum nutriente, dessecaçao, irradiaçao, presença de H2O2 e desinfecçao química. Seu papel ecológico é enfatizado pelo fato de que nao produz compostos de microrganismos patogênicos, aumentando o interesse na mesma. Representantes da família Enterobacteriaceae, por outro lado, apesar de nao serem frequentemente documentados como produtores de PHAs, sao capazes de hiperexpressar genes relacionados à síntese desse polímero e têm sido usados com sucesso para esse fim em experimentos de engenharia genética.7,33 Cinética de crescimento dos isolados Condiçoes de fermentaçao como a relaçao C:N, a concentraçao de oxigênio dissolvido e quantidade de células no meio de cultura34 aliada à otimizaçao das vias metabólicas35 sao fatores de extrema importância quando se busca maximizar a biossíntese de PHA pelos microrganismos. A presença da fonte orgânica de nitrogênio nos meios 02, 03 e 05 permitiu que os microrganismos utilizassem o substrato disponível gerando maior biomassa em comparaçao ao meio 04 (Tabela 3).
Esse resultado está de acordo com Sreekanth et al.,36 que estudaram como diferentes fontes de nitrogênio (0,2 g N L-1) afetam o rendimento da produçao de PHA e α-amilase numa cepa de Bacillus sp. cultivada em meio contendo 20 g L-1 de sacarose como fonte de carbono. Os autores observaram que fontes orgânicas de nitrogênio resultaram em maior biomassa e também maior quantidade de PHA dentro das células. Rendimentos de PHA diferenciados como reflexo da fonte nitrogenada presente no meio de cultura também foram documentados em outros experimentos semelhantes.36-39 Yüksekdağ et al.,38 por exemplo, demonstraram que em meio contendo peptona, duas linhagens de Bacillus apresentaram os maiores rendimentos de PHB em comparaçao com os meios contendo sulfato de amônio, nitrato de potássio, e os aminoácidos L-cisteína e L-glicina (sempre na concentraçao de 20 g L-1). Já Gomaa et al.39 avaliaram o efeito de 19 fontes de nitrogênio na produçao de PHA em Bacillus subtilis e verificaram que sulfato de amônio e nitrato de amônio na concentraçao de 1 g L-1 foram as melhores fontes para o crescimento da cepa. No presente trabalho, é possível afirmar que a adiçao de hidrolisado de proteínas favoreceu o crescimento de Enterobacter aerogenes BMA-10 Klebsiella oxytoca BMA-13. Todavia, os crescimentos observados no meio 01 foram equiparáveis aos do meio 04. Nesta última condiçao, as células bacterianas conseguiram se multiplicar em meio ausente de glicose provavelmente devido à presença de alguma fonte de carbono que ainda tenha restado no meio de cultura que foi introduzido juntamente com o inóculo, pois quando qualquer microrganismo se multiplica no meio de cultura, há um decréscimo proporcional na quantidade de nutrientes até o ponto em que a concentraçao de nutrientes por organismo atinja um nível crítico e a multiplicaçao cesse. Pseudomonas fluorescens BMA-05 foi o único isolado que nao acidificou nenhum dos meios de cultura, em nenhum dos tempos. Para os outros três isolados, os meios 02, 03 e 04 mantiveram-se próximos da neutralidade em todos os intervalos de tempo amostrados. Os meios 01 e 05, por sua vez, sofreram acidificaçao já após 24 h de cultivo, permanecendo na mesma faixa de pH até o final do experimento. Entretanto, como o meio 01 é limitado em nitrogênio [0,2 g L-1 de (NH4)2SO4], os microrganismos consumiram somente até 50% da glicose após 96 h de cultivo. Esse meio de cultura é comumente utilizado por pesquisadores que buscam selecionar possíveis bactérias produtoras de PHAs, entretanto, tais condiçoes nutricionais nao sao válidas para todos os microrganismos. Shi et al.,40 por exemplo, verificaram que para Brachymonas sp. P12 as condiçoes de excesso de carbono tiveram efeito negativo no acúmulo de PHA quando comparado a condiçoes limitantes de carbono. Nessa cultura, a aeraçao, limitaçao de C, excesso ou limitaçao de N e excesso ou limitaçao de P afetam a produçao de PHA. No meio 05, grande parte da glicose foi rapidamente consumida tanto por Enterobacter aerogenes BMA-10 como Klebsiella oxytoca BMA-13 e resultaram em maior biomassa desses microrganismos. Por sua vez, tanto Bacillus pumilus BLD-07 como Pseudomonas fluorescens BMA-05 apresentaram comportamento semelhante nesses dois meios de cultura. Encontrar a condiçao ideal para a produçao do biopolímero varia nao somente de acordo com a espécie bacteriana, mas também entre as diferentes cepas da mesma espécie,41 e como o nitrogênio faz parte da composiçao de macromoléculas como proteínas e ácidos nucleicos, a adiçao dele faz-se necessária à multiplicaçao microbiana.
CONCLUSAO Quatro bactérias (Enterobacter aerogenes BMA-10, Pseudomonas fluorescens BMA-05, Klebsiella oxytoca BMA-13 e Bacillus pumilus BLD-07) entre um total de 24 isoladas de solo de Mata Atlântica e Lodo de ETE de agroindústria sucroalcooleira foram selecionadas por serem capazes de turvar acentuadamente o meio mínimo mineral indutor de produçao de PHAs, mediante diferentes fontes de carbono, além de reterem o corante preto de Sudao e apresentarem em seu genoma o gene phaC, codante da PhaC sintase. Sob luz ultravioleta, culturas sólidas contendo o corante azul do Nilo só apresentaram fluorescência característica de acúmulo de PHA para um desses três isolados, isto é, Enterobacter aerogenes BMA-10. O meio mínimo acrescido de peptona demonstrou ser um meio de cultivo favorável ao crescimento dos isolados.
AGRADECIMENTOS Ao Conselho Nacional de Desenvolvimento Científico e Tecnológico e à S. A. Usina Coruripe Açúcar e Alcool, pelo apoio financeiro. A Plataforma de Sequenciamento de DNA da Universidade Federal de Pernambuco pelo serviço prestado.
REFERENCIAS 1. Tokiwa, Y.; Calabia, B. P.; Ugwu, C. U.; Aiba, S.; Int. J. Mol. Sci. 2009, 10, 3722; Mano, E. B.; Mendes, L. C.; A natureza e os polímeros: meio ambiente, geopolímeros, fitopolímeros e zoopolímeros, 1ª ed., Blücher: Sao Paulo, 2013. DOI: http://dx.doi.org/10.3390/ijms10093722 PMID: 19865515 2. Khanna, S.; Srivastava, A. K.; Process Biochem. 2005, 40, 607. DOI: http://dx.doi.org/10.1016/j.procbio.2004.01.053 3. Silva, L. F.; Gomez, J. G. C; Rocha, R. C. S.; Taciro, M. K.; Pradella, J. G. C.; Quim. Nova 2007, 30, 1732; Brämer, C. O.; Vandamme, P.; Silva, L. F.; Gomez, G. C.; Steinbüchel, A.; Int. J. Syst. Evol. Microbiol. 2001, 51, 1709; Mantelatto, P. E.; Duzzi, A. M.; Sato, T.; Durao, N. A. S.; Nonato, R. V.; Rocchiccioli, C.; Kesserlingh, S. M.; Br PI 0405622-1 A, 2006. DOI: http://dx.doi.org/10.1590/S0100-40422007000700040 4. Laycock, B.; Halley, P.; Pratt, S.; Werker, A.; Lant, P.; Prog. Polym. Sci. 2013, 38, 536. DOI: http://dx.doi.org/10.1016/j.progpolymsci.2012.06.003 5. Squio, C. R.; Aragao, G. M. F.; Quim. Nova 2004, 27, 615. DOI: http://dx.doi.org/10.1590/S0100-40422004000400016 6. Jendrossek, D.; J. Bacteriol. 2009, 191, 3195. DOI: http://dx.doi.org/10.1128/JB.01723-08 PMID: 19270094 7. Naheed, N.; Jamil, N.; Hasnain, S.; Afr. J. Microbiol. Res. 2011, 5, 4097. 8. Silva, N. Informes Técnicos Nº1. Testes Bioquímicos Para Identificaçao de Bactérias em Alimentos. Campinas: Instituto de Tecnologia de Alimentos. 1996. 9. Neder, R. N.; Microbiologia. Manual de Laboratório, Livraria Nobel S. A.: Sao Paulo, 1992. 10. Romo, D. M. R.; Grosso, M. V.; Solano, N. C. M.; Castaño, C. M.; Electron. J. Biotechnol. 2007, 10, 348. 11. Sheu, D.; Wang, Y.; Lee, C.; Microbiology 2000, 146, 2019. DOI: http://dx.doi.org/10.1099/00221287-146-8-2019 PMID: 10931906 12. Winsley, T.; van Dorst, J. M.; Brown, M. V.; Ferrari, B. C.; Appl. Environ. Microbiol. 2012, 78, 5938. DOI: http://dx.doi.org/10.1128/AEM.01299-12 PMID: 22685133 13. Larkin M. A.; Blackshields, G.; Brown, N. P.; Chenna, R.; McGettigan, P. A.; McWilliam, H.; Valentin, F.; Wallace, I. M.; Wilm, A.; Lopez, R.; Thompson, J. D.; Gibson, T. J.; Higgins, D. G.; Bioinformatics 2007, 23, 2947. DOI: http://dx.doi.org/10.1093/bioinformatics/btm404 PMID: 17846036 14. Lowry, O. H.; Rosebrough N. J.; Farr A. L.; Randall R. J.; J. Biol. Chem. 1951, 193, 265; Miller, G. L.; Anal. Chem. 1959, 31, 426. PMID: 14907713 15. Amoli, R. I.; Baei, M. S.; Pirouz, F.; Middle East J. Sci. Res. 2013, 14, 1035. 16. López-Cuellar, M. R.; Alba-Flores, J.; Rodríguez, J. N.; Pérez-Guevara, F.; Int. J. Biol. Macromol. 2011, 48, 74. DOI: http://dx.doi.org/10.1016/j.ijbiomac.2010.09.016 PMID: 20933541 17. Choi, J.; Lee, S. Y.; Appl. Microbiol. Biotechnol. 1999, 51, 13. DOI: http://dx.doi.org/10.1007/s002530051357 18. Huang, Y.; Chen, P.; Semblante, G. U.; You, S.; J. Microbiol. Biotechnol. 2012, 22, 1141. DOI: http://dx.doi.org/10.4014/jmb.1111.11040 PMID: 22713992 19. Marang, L.; Jiang, Y.; van Loosdrecht, M.; Kleerebezem, R.; Bioresour. Technol. 2013, 142, 232. DOI: http://dx.doi.org/10.1016/j.biortech.2013.05.031 PMID: 23743427 20. Koller, M.; Bona, R.; Chiellini, E.; Fernandes, E.; Horvat, P.; Kutschera, C.; Hesse, P.; Braunegg, G.; Bioresour. Technol. 2008, 99, 4854. DOI: http://dx.doi.org/10.1016/j.biortech.2007.09.049 PMID: 18053709 21. Tyo, K. E.; Zhou, H.; Stephanopoulos, G. N.; Appl. Environ. Microbiol. 2006, 72, 3412. DOI: http://dx.doi.org/10.1128/AEM.72.5.3412-3417.2006 PMID: 16672485 22. Legat, A.; Gruber, C.; Zangger, K.; Wanner, G.; Stan-Lotter, H.; Appl. Microbiol. Biotechnol. 2010, 87, 1119. DOI: http://dx.doi.org/10.1007/s00253-010-2611-6 PMID: 20437233 23. Serafim, L. S.; Lemos, P. C.; Reis, M. A. M.; Boletim da Sociedade Portuguesa de Biotecnologia 2003, 76, 16. 24. Ostle, A. G.; Holt, J. G.; Appl. Environ. Microbiol. 1982, 44, 238. PMID: 6181737 25. Mesquita, D. P.; Amaral, A. L.; Leal, C.; Oehmen, A.; Reis, M. A. M; Ferreira, E. C.; Anal. Chim. Acta 2015, 865, 8. DOI: http://dx.doi.org/10.1016/j.aca.2015.01.018 PMID: 25732579 26. Rehm, B. H.; Biochem. J. 2003, 376, 15. DOI: http://dx.doi.org/10.1042/bj20031254 PMID: 12954080 27. López, N. I.; Pettinari, M. J.; Méndez, B. S.; FEMS Microbial Ecology 1997, 22, 129. DOI: http://dx.doi.org/10.1016/S0168-6496(96)00083-9 28. Inoue, D.; Suzuki, Y.; Uchida, T.; Morohoshi, J.; Sei, K.; J. Biosci. Bioeng. 2015, 121, 47. DOI: http://dx.doi.org/10.1016/j.jbiosc.2015.04.022 PMID: 26071670 29. Hai, T.; Lange, D.; Rabus, R.; Steinbüchel, A.; Appl. Environ. Microbiol. 2004, 70, 4440; Han, J.; Lu, Q.; Zhou, L.; Liu, H.; Xiang, H.; Appl. Environ. Microbiol. 2009, 75, 6168. DOI: http://dx.doi.org/10.1128/AEM.70.8.4440-4448.2004 PMID: 15294771 30. Shamala, T. R.; Chandrashekar, A.; Vijayendra, S. V. N.; Kshama, L.; J. Appl. Microbiol. 2003, 94, 369. DOI: http://dx.doi.org/10.1046/j.1365-2672.2003.01838.x PMID: 12588544 31. Solaiman, D. K. Y.; Ashby, R. D.; Biomacromolecules 2005, 6, 532. DOI: http://dx.doi.org/10.1021/bm0493640 PMID: 15762609 32. Parvathi, A.; Krishna, K.; Jose, J.; Joseph, N.; Nair, S.; Braz. J. Microbiol. 2009, 40, 269. DOI: http://dx.doi.org/10.1590/S1517-83822009000200012 PMID: 24031357 33. Zhang, H.; Obias, V.; Gonyer, K.; Dennis, D.; Appl. Environ. Microbiol. 1994, 60, 1198. PMID: 8017916 34. Manangan, T.; Shawaphun, S.; ScienceAsia 2010, 36, 199. DOI: http://dx.doi.org/10.2306/scienceasia1513-1874.2010.36.199 35. Magdouli, S.; Brar, S. K.; Blais, J. F.; Tyagi, R. D.; Biomass Bioenergy 2015, 74, 268. DOI: http://dx.doi.org/10.1016/j.biombioe.2014.12.017 36. Sreekanth, M. S.; Vujavendra, S. V.; Joshi, G. J.; Shamala T. R.; J. Food Sci. Technol. 2013, 50, 404. DOI: http://dx.doi.org/10.1007/s13197-012-0639-6 PMID: 24425935 37. Lee, W-H.; Loo, C-Y.; Nomura, C. T.; Sudesh, K.; Bioresour. Technol. 2008, 99, 6844. DOI: http://dx.doi.org/10.1016/j.biortech.2008.01.051 PMID: 18325764 38. Yüksekdağ, N.; Aslim, B.; Beyatli, Y. Mercan, N.; Afr. J. Biotechnol. 2004, 3, 63. DOI: http://dx.doi.org/10.5897/AJB2004.000-2011 39. Gomaa, E. Z.; Braz. Arch. Biol. Technol. 2014, 57, 145. 40. Shi, H. P.; Lee, C. M.; Ma, W. H.; World J. Microbiol. Biotechnol. 2007, 23, 625. DOI: http://dx.doi.org/10.1007/s00253-007-1184-5 41. Saharan, B. S; Grewal, A.; Kumar, P.; Chin. J. Biol. 2014, 2014, 1. DOI: http://dx.doi.org/10.1155/2014/396708 |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access