Revisão
|
|
| Potencial catalítico de lipases ligadas ao micélio de fungos filamentosos em processos de biotransformação Potential catalytic of mycelium-bound lipase of filamentous fungi in biotransformation processes |
|
Daniela V. CortezI; Heizir F. de CastroI; Grazielle S. S. AndradeII,*
IDepartamento de Engenharia Química, Escola de Engenharia de Lorena, Universidade de São Paulo, 12.602-810 Lorena - SP, Brasil Recebido em 11/06/2016 *e-mail: email@domain_name_here.com Over the past decades, mycelium-bound lipase of filamentous fungus has been extensively studied as an alternative biocatalyst in biotransformation processes. Mycelium-bound lipase can be used directly as suspended free cells or immobilized within biomass support particles as whole cells biocatalyst. In the latter, cells can be immobilized in situ or extra situ using different support materials avoiding the purification step. This represents an attractive, cost-effective technology to enhance chemical reaction efficiency. The present review covers the great versatility of mycelium-bound lipase to mediate biotransformation processes, given particular emphasis in its use as biocatalyst for biodiesel production. INTRODUÇÃO Reações de biotransformação ou biocatálise, uma das ferramentas da química verde, são amplamente usadas em síntese orgânica. Este tipo de processo emprega enzimas purificadas ou células íntegras, em especial, células microbianas livres ou imobilizadas e apresenta notável destaque como uma tecnologia suplementar útil para a indústria química, fornecendo em alguns casos, reações que não são facilmente conduzidas pela química orgânica clássica ou, em outros casos, promovendo reações que podem substituir etapas químicas severas.1-2 Adicionalmente, outros atributos benéficos da biotransformação incluem versatilidade reacional, especificidade, redução no número de etapas no processo e alto rendimento.3 Diferente da fermentação, na biotransformação o biocatalisador transforma moléculas precursoras pré-formadas (naturais ou sintéticas) em limitado número de etapas. Além disto, o produto formado não é limitado pelo metabolismo do biocatalisador.4-5 As enzimas são catalisadores notavelmente eficazes, responsáveis por milhares de reações químicas coordenadas, envolvidas nos processos biológicos dos sistemas vivos e podem agir in vitro.6-7 Os processos enzimáticos apresentam uma série de vantagens quando comparados com os processos químicos convencionais catalisados ou não. Segundo Faber,4 tipicamente, as velocidades das reações mediadas por enzimas são superiores em relação aquelas não catalisadas, por um fator de 108 - 1010. Entretanto, a literatura ainda aponta outras perspectivas sobre o vasto poder catalitico das enzimas, podendo alcançar, por exemplo, velocidades de reação aproximadamente 1010 a 1026 vezes superiores aos encontrados na ausência do catalisador.8 Diferente dos catalisadores metálicos nos processos químicos, as enzimas agem como um reagente ambientalmente favorável, uma vez que são completamente degradáveis e os processos enzimáticos são realizados sob condições operacionais mais brandas. Ainda, as reações biocatalíticas podem transformar uma grande variedade de substâncias naturais ou não, em ambiente aquoso ou orgânico.4 A ação de células íntegras (whole cells), por sua vez, pode ser comparada com aquela de enzimas em células que se encontram em estado estacionário (resting cells) ou mortas (não viáveis), mas com a maquinaria enzimática de interesse mantida ativa.9 Na forma mais simples, a célula íntegra é usada como suporte, evitando as etapas de extração, isolamento e purificação das enzimas de interesse, as quais são normalmente trabalhosas e contribuem para tornar o processo oneroso. Ademais, o ambiente natural das células íntegras garante a estabilidade da enzima porque sua localização espacial otimizada permanece intacta.10-12 As células íntegras podem ser imobilizadas como um fenômeno natural ou por meio de procedimentos artificiais. As células imobilizadas são preferidas em relação às células livres, uma vez que são mais convenientes de manusear e de separar do meio reacional, além de serem mais estáveis.3 Cerca de dois terços das pesquisas em síntese orgânica por biotransformação envolvem a ação de enzimas hidrolíticas como as lipases, as proteases e as esterases.4 Impulsionadas por sua versatilidade, as lipases apresentam destaque no cenário mundial de enzimas pois permitem a catálise de reações de hidrólise e de síntese, muitas vezes de forma quimio, regio e enantiosseletiva.13 Esta característica faz com que as lipases apresentem aplicação em vários setores, como por exemplo, na indústria alimentícia, farmacêutica, de química fina, oleoquímica, energia (biodiesel) e na formulação de detergentes, dentre outros. Com base em indicadores, científico e tecnológico, Daiha et al.,14 relataram que a importância das lipases como biocatalisadores, identificada em 1930, ainda mantem elevado grau de interesse para aplicação em diversos segmentos industriais, com projeção de crescimento na demanda mundial estimada em 6,2% anualmente, atingindo US$ 345 milhões em 2017. A disponibilidade e os sólidos conhecimentos de seleção, purificação, preservação, manipulação bem como condições de cultivo de fungos filamentosos e a busca constante de novas e alternativas fontes de lipases, criam oportunidades para o aprimoramento e/ou descobertas de novas formas de obtenção e aplicação da biomassa como fonte da enzima. Dentro deste contexto, a presente revisão tem como objetivo apresentar dados de estudo e aplicação de lipase ligada ao micélio de fungos filamentosos, iniciando com os principais aspectos relacionados à enzima, bem como a obtenção e o preparo do biocatalisador na forma de células íntegras.
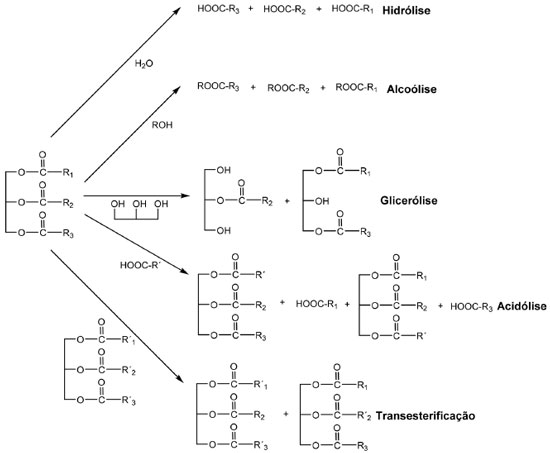
LIPASE Aspectos gerais As lipases (glicerol éster hidrolases, EC 3.1.1.3) fazem parte do grupo das α/β-hidrolases, as quais possuem um padrão de dobramento característico. A descrição original deste dobramento foi baseada no estudo comparativo feito por Ollis et al.,15 das estruturas tridimensionais de hidrolases de origem filogenética e função catalítica diferentes. As α/β-hidrolases são compostas por um núcleo de oito folhas β, principalmente paralelas, rodeadas em ambos os lados por α-hélices.16 Com poucas exceções, as lipases apresentam ativação interfacial. A presença de um grande volume de agregado lipídico próximo à superfície da enzima altera o ambiente dielétrico nesta região e os resíduos carregados da proteína interagem mais fortemente com um substrato lipídico do que com a água. Isto faz com que muitas lipases apresentem uma alteração conformacional.17 Nestas enzimas, uma tampa na forma de laço ou de uma pequena α-hélice, bloqueia o acesso ao sítio ativo. Quando em presença de um substrato lipídico (ou inibidor), a tampa se adapta a uma nova conformação de forma a permitir o acesso ao sítio ativo e ao mesmo tempo, uma grande área hidrofóbica também fica exposta, o que facilita a ligação da lipase à interface água-óleo.17-18 Entretanto, nem todas lipases apresentam a tampa que controla o acesso do substrato ao sítio ativo, tornando, a presença desta estrutura e a "ativação interfacial", critérios não adequados para sua classificação. Desta forma, lipases podem ser melhor definidas como carboxilesterases que catalisam a hidrólise de acilgliceróis de cadeia longa.19 A hidrólise de um substrato lipolítico catalisada por uma lipase se inicia com a ligação do éster do lipídeo à enzima e o ataque do resíduo serina da biomolécula à carbonila do substrato, formando um intermediário tetraédrico. A clivagem deste intermediário tetraédrico libera o álcool, permanecendo um intermediário correspondente à enzima acilada. Em uma reação de hidrólise a água ataca esta enzima acilada para formar um segundo intermediário tetraédrico e a clivagem deste intermediário libera o ácido. Alternativamente, outro nucleófilo tal como um álcool pode atacar a enzima acilada, resultando, então, um novo éster (uma reação de transesterificação). Na maioria dos casos, a formação da enzima acilada é uma etapa rápida, portanto, a remoção do grupo acila é a etapa determinante.16 As lipases catalisam uma série de diferentes reações. Os processos básicos de hidrólise e formação de ésteres (esterificação) podem ser combinados em uma sequência lógica para resultar em reações denominadas interesterificações.20 Dependendo dos materiais de partida, pode ocorrer uma reação de acidólise, na qual ocorre a transferência do grupamento acila entre um acilglicerol e um ácido carboxílico. Ainda pode ocorrer uma alcoólise, na qual o grupamento acila é transferido entre um acilglicerol e um álcool. Outra reação também possível é a transesterificação, caracterizada pela troca de grupamentos acila entre dois acilgliceróis (Figura 1). As lipases também catalisam a reação de glicerólise. Neste caso, ocorre a interesterificação do triacilglicerol com glicerol.22
A especificidade é uma característica importante das lipases. Estas enzimas podem ser específicas com relação à molécula ácida ou alcoólica do substrato.13 As lipases não específicas clivam as moléculas de acilglicerol na posição randômica, produzindo ácidos graxos livres, glicerol, monoacilgliceróis e diacilgliceróis como intermediários. As lipases 1,3 específicas liberam ácidos graxos das posições 1 e 3 e formam, por esta razão, produtos com composições diferentes daquelas obtidas pelas lipases não regiosseletivas, ou mesmo pelo catalisador químico. As lipases ácido graxo específicas, por sua vez, apresentam ação específica na hidrólise de ésteres cujos ácidos graxos são de cadeia longa insaturada com duplas ligações, em cis no carbono 9.13,23 Outra característica importante é a estereoespecificidade, ou seja, a capacidade que algumas lipases possuem de discriminar os enantiomêros de uma mistura racêmica. A especificidade estrutural ou regiosseletividade é decorrente da orientação imposta pelas dimensões e pela estrutura do centro ativo à ligação do substrato. Estas restrições levam à distinção e à transformação seletiva de funções quimicamente similares na mesma molécula. A seletividade e a estereoquímica advém da própria quiralidade da enzima, ou seja, de sua simetria estrutural, que limita a ação em substratos que não satisfaçam determinadas relações espaciais. Desse modo, a catálise enzimática permite transferir ou criar centros quirais nas moléculas, assim como distinguir formas enantioméricas. Lipases têm sido empregadas para resolução de racematos, resultando em rendimentos elevados, para obtenção de ésteres, álcoois e ácidos opticamente puros.12-13,24 Essa versatilidade faz destas enzimas, biocatalisadores potenciais devido a sua grande aplicabilidade em vários setores, conforme exemplificado na Figura 2.
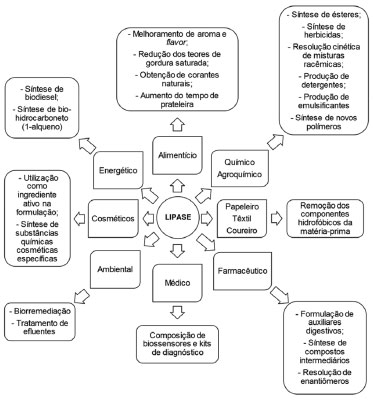 Figura 2. Alguns exemplos de aplicação de lipases nos diversos setores
Em um estudo muito interessante visando avaliar se o uso das lipases como biocatalisadores ainda é relevante no contexto tecnológico, Daiha et al.,14 analisaram o número de publicações e patentes relacionados, em um período de trinta anos (1983 a 2012), com base em quatro setores industriais de diferentes estágios de desenvolvimento e com diferentes níveis tecnológicos. Durante a busca de patentes, foram obtidos 671 documentos relacionados à resolução cinética, 456 na formulação de detergente, 165 para a produção de biodiesel e 544 para produtos do setor alimentício e veterinário. Com relação a publicações científicas, o número de documentos encontrados para cada setor correspondeu a 1352, 117, 439, e 315, respectivamente. Neste estudo, os autores concluíram que a aplicação destes biocatalisadores para o setor industrial ainda é significativo, entretanto, ressaltam a necessidade do desenvolvimento de lipases que alcancem seu potencial catalítico total. Fontes produtoras As lipases são encontradas na natureza em tecidos animais e vegetais e em microrganismos (fungos, leveduras e bactérias). Dependendo da sua origem, podem apresentar massa molecular variando de 19 a 75 kDa e atuar numa ampla faixa de pH (4,0-9,0) e de temperatura, a qual varia do ambiente até 70 ºC.23 Dentre as fontes produtoras de lipase, as microbianas são as mais utilizadas industrialmente, porque as enzimas apresentam uma grande diversidade em suas propriedades catalíticas e especificidades por substratos.25 Os microrganismos com potencial para produzir lipases podem ser encontrados em vários habitats, incluindo, por exemplo, ambiente marinho,26 ambiente antártico,27-28 resíduos de óleos vegetais e de petróleo29 e indústrias de produtos lácteos,30 solos contaminados,31 plantas,32 e alimentos deteriorados33. Desta forma, a natureza e os ambientes oferecem um extraordinário e potencial acervo de fontes de lipase, as quais podem apresentar novas propriedades e aplicações. Os fungos filamentosos possuem a habilidade de crescer em substratos bastante simples e de baixo custo, além da excepcional capacidade de expressar e excretar proteínas.34 Estas características e graças ao avanço do conhecimento da fisiologia, bioquímica e genética dos fungos filamentosos tornou possível a exploração de seu imenso potencial para a produção de lipase, assim como outras enzimas de aplicação industrial.35 Alguns dos fungos produtores de lipase com destacada aplicação comercial são aqueles pertencentes dos gêneros Rhizopus sp., Aspergillus sp., Penicillium sp., Geotrichum sp., Mucor sp. e Rhizomucor sp. Entretanto, a Figura 3 representa uma listagem de diferentes espécies desses gêneros, bem como de outros gêneros descritos na literatura como fontes promissoras da enzima. Essas espécies foram isoladas de amostras de solo, fezes de animais, partes de plantas e em frutos (endofíticos).
 Figura 3. Exemplos de fungos filamentosos avaliados para obtenção de lipases
Os fungos endofíticos habitam o interior das plantas, sendo encontrados nos órgãos e tecidos vegetais como sementes, folhas e ramos, sem causar danos aos seus hospedeiros, mas agindo como controladores de patógenos e predadores por meio da produção de substâncias antagônicas e enzimas.82-83 A Figura 4 destaca alguns exemplos de estudos de detecção e seleção de fungos endofíticos isolados a partir de amostragens originadas do Brasil, da Espanha e da Índia.
 Figura 4. Exemplos de estudos de detecção e seleção de fungos endofíticos, citando as potenciais fontes de lipase
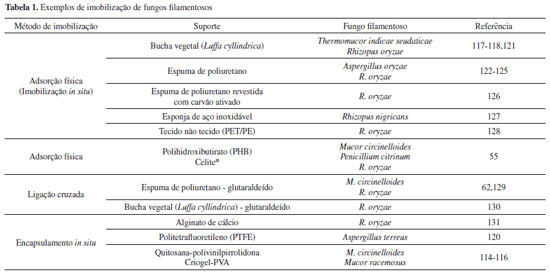
Condições para a produção de lipases ligadas ao micélio de fungos filamentosos A otimização das condições de cultivo é de grande importância uma vez que influenciam nas propriedades do microrganismo produtor da enzima, bem como na razão das lipases retidas no micélio e excretadas para meio de cultivo.25 Em geral, as lipases são excretadas para o meio de cultivo com o objetivo de captar os nutrientes para a célula microbiana, clivando o substrato lipídico em componentes mais polares e, portanto, mais fáceis de absorção.93 Entretanto, a excreção da lipase para meio de cultivo depende do gênero do fungo filamentoso, como por exemplo para Geotrichum candidum no qual a lipase é formada na região do envelope celular e retida por um período, antes de ser liberada.94 Esse comportamento foi também confirmado no trabalho desenvolvido por Loo et al.,95 que constataram em 54 h de incubação elevada atividade lipolítica nas células íntegras de G. candidum, seguida de redução da atividade após este período, provavelmente devido à liberação da enzima para o meio de cultivo. Em estudo com Rhizopus oryzae, Hama et al.,96 constataram a presença de dois tipos da enzima, sendo uma lipase de 34 kDa (ROL 34) encontrada ligada à parede celular e uma lipase de 31 kDa (ROL 31), ligada à membrana e parede celular. Neste mesmo estudo, os autores também observaram um decréscimo acentuado na quantidade de lipase ligada à membrana com o tempo de cultivo. O crescimento e as distintas atividades metabólicas dos organismos são respostas às condições do ambiente físico-químico que os rodeia.97 A produção de enzimas em escala industrial se faz majoritariamente por fermentação submersa, embora haja uma tradição estabelecida na utilização de fermentação sólida ou semissólida.98-99 Os processos submersos são aqueles em que a célula produtora se desenvolve no seio do meio de cultivo, sob agitação. Os mecanismos que regulam a biossíntese de lipases variam de forma significativa entre os diferentes microrganismos produtores. Em alguns casos, a produção de lipase parece ser constitutiva e independente da adição de substratos lipídicos no meio de cultivo. Entretanto, sua presença pode aumentar o nível de atividade lipolítica. Por outro lado, há microrganismos que preferem, por exemplo, óleo, triacilgliceróis ou qualquer outro indutor como ácidos graxos, do que fontes de carbono simples como açúcares, para a produção da enzima. Deste modo, as fontes de carbono lipídicas podem ser consideradas essenciais para a obtenção de um alto rendimento em lipase. Fontes de nitrogênio e micronutrientes também devem ser considerados para o crescimento microbiano e a otimização da produção da enzima.100 A literatura relata diversos estudos de otimização do meio e das condições de cultivo de microrganismos visando obter biomassa com elevada atividade lipolítica (ligada ao micélio). Por exemplo, Essamri et al.,101 verificaram que as variáveis pH, temperatura, concentração de óleo e período de incubação foram significativas sobre o crescimento celular de Rhizopus oryzae e a atividade lipolítica no interior das células. Os resultados mostraram que os rendimentos mais elevados foram obtidos quando utilizados pH 5,0, temperatura de 25 ºC, 3% de concentração de óleo de colza e 120 h de incubação. Yang et al.,102 Zeng et al.,103 Wang et al.,104 observaram que os diferentes efeitos dos óleos na produção de lipase por R. oryzae e Rhizopus arrhizus são causados pelos ácidos graxos presentes em sua composição, e a produção da enzima está relacionada aparentemente com a quantidade de ácido oleico presente em cada óleo. A utilização de óleos refinados como substrato no meio de cultivo aumentou a atividade catalítica das células íntegras, e dentre diversos óleos testados, o óleo de oliva foi o que promoveu atividades lipolíticas mais elevadas. Em outro estudo, as células de R. oryzae provenientes de meio de cultivo enriquecido com os ácidos oleico e linoleico mostraram atividade de metanólise superior à observada com as células cujo meio foi enriquecido com ácidos graxos saturados. Além disto, células provenientes de meio enriquecido com ácido palmítico mostraram maior estabilidade do que células enriquecidas com ácidos graxos insaturados.105 Utilizando também R. oryzae, Sun et al.,106 observaram que diferentes óleos indutores resultaram em composições variadas de membrana celular, o que influenciou diretamente na atividade catalítica e na estabilidade operacional do biocatalisador. A adição de óleos vegetais ricos em ácidos graxos insaturados promoveu um aumento na produção de lipase ligada ao micélio. Os autores também observaram que as células cultivadas em meio enriquecido com ácidos graxos saturados exibiram maior estabilidade operacional do que as células enriquecidas com ácidos graxos insaturados. Durante estudo com Rhizopus chinensis para verificar a produção e atividade da lipase ligada ao micélio, Wang et al.,104 constataram que o óleo de oliva foi melhor indutor de enzima dentre uma variedade de substratos lipídicos. Com a suplementação do meio cultivo com peptona (56 g L-1), maltose (13 g L-1), K2HPO4 (3 g L-1), MgSO4.7H2O (5 g L-1) e pH inicial 6,0, a atividade da biomassa celular proporcionou um incremento da ordem 90%. Ainda com relação ao R. chinensis, Teng e Xu,107 verificaram dentre uma série de variáveis experimentais que as mais significativas foram volume de inóculo, agitação, concentração de óleo de oliva e volume do meio de fermentação. Os autores também constataram a importância do óleo de oliva na produção da lipase ligada ao micélio. Em 2014, Andrade et al.,62 avaliaram o efeito dos óleos vegetais de soja, de girassol, de linhaça, castanha-do-pará e de oliva sobre o crescimento celular e a atividade lipolítica de Mucor circinelloides. Todos os óleos favoreceram as variáveis respostas, sendo os valores máximos obtidos quando foi utilizado o óleo de oliva numa concentração de 30 g L-1. Segundo os autores, estes resultados podem estar relacionados à alta proporção de ácido oleico (70%) no óleo de oliva. Ainda neste estudo, a adição de fontes de nitrogênio ao meio, bem como o nível de inóculo e a aeração do meio também foram parâmetros importantes. O cultivo na presença e ausência de óleo de oliva também apresentou efeito sobre a atividade lipolítica das células íntegras de Aspergillus niger, conforme verificado por Romero et al.,70. As especificidades dos biocatalisadores variaram com o tipo de reação e o substrato utilizado (ácido graxo, éster de ácido graxo ou álcool). O cultivo do fungo filamentoso na presença do indutor resultou em uma lipase ligada ao micélio mais reativa em reações de transesterificação e esterificação com ésteres de ácido graxo de cadeia longa na presença de etanol. Imobilização de células íntegras O processo de biotransformação pode ser conduzido por células íntegras no estado livre, entretanto a imobilização facilita o manuseio e separação do biocatalisador do meio reacional, além de promover maior estabilidade do sistema.3 Imobilização é um termo geral que descreve as muitas formas de fixação, encapsulamento ou aprisionamento de células ou enzimas em um suporte sólido. Especificamente, a imobilização de células íntegras representa uma forma particular de adesão celular baseada na habilidade de certos microrganismos de se fixarem em suportes sólidos por meio de ligações naturais ou físico-químicas.108 O conceito de células íntegras imobilizadas em biocatálise é diferente do conceito de células imobilizadas utilizadas em processos fermentativos. As células são normalmente tratadas antes ou após imobilização, de forma a perder a capacidade de multiplicação (viabilidade), mas com as enzimas desejadas mantidas estáveis e em estado ativo. No caso de processos fermentativos convencionais, as células imobilizadas são mantidas em estado de crescimento dentro da matriz por um suprimento contínuo dos nutrientes, favorecendo a manutenção de suas funções biológicas necessárias para o processo.9 Para a imobilização de células são utilizados os métodos amplamente utilizados na tecnologia de imobilização de enzimas, tais como adsorção, ligação covalente, ligação cruzada e aprisionamento (encapsulamento). Entretanto, devido a morfologia das células, alguns critérios devem ser impostos para seleção adequada do suporte e do método de imobilização. A literatura disponível referente a imobilização de células íntegras é escassa, podendo ser destacado apenas revisões abrangentes publicadas por Fukui e Tanaka,9 Jack e Zajic,109 e Karel et al.,110 abordando aspectos importantes que serviram de base para elaboração dos conceitos descritos neste tópico. As principais técnicas normalmente utilizadas para imobilização de células integras são baseadas na adsorção em suportes inertes, na encapsulação e aprisionamento em matrizes poliméricas ou ligação covalente, cruzada e iônica, em suportes previamente ativados.110 Os sistemas em que as células são imobilizadas por ligação na superfície são populares devido à facilidade de execução. Neste caso, a força com a qual as células estão ligadas ao suporte varia de acordo com o tipo de células e o tipo de suporte.110 Neste tipo de imobilização podem ocorrer interações iônicas ou adsortivas naturais, ou ligações covalentes entre grupos reativos do suporte e do biocatalisador.111 As células podem ser quimicamente ligadas a uma superfície por uma variedade de métodos que incluem a ligação cruzada com glutaraldeído, silanização a um suporte de sílica e quelação de óxidos de metal.110 Em relação ao aprisionamento em matriz porosa, há dois métodos possíveis para este tipo de imobilização: no primeiro caso, as células se difundem em uma matriz porosa pré-formada e a retenção ocorre no momento em que a mobilidade fica dificultada pelo aumento do número de células. Os suportes pré-formados são mais vantajosos porque são mais resistentes à compressão e desintegração que podem ocorrer em leitos empacotados ou em tanques agitados.110 Normalmente, utilizam-se este tipo para a imobilização de fungos filamentosos, visto que ocorre adesão das células microbianas à superfície do suporte. Quando os poros são maiores, as células em crescimento (hifas) penetram no suporte, preenchendo seu espaço interno.112 No segundo caso de imobilização por aprisionamento, a matriz porosa é sintetizada em torno das células a serem imobilizadas. Neste procedimento são utilizados compostos (polímeros) que gelificam em forma matrizes porosas hidrofílicas.113 Entre as várias técnicas disponíveis para imobilização células de fungos filamentosos para aplicação em biocatálise, a que utiliza partículas porosas para suporte de biomassa constituídas por cubos de espuma de poliuretano (do inglês, Biomass Support Particles - BSPs) é a mais amplamente utilizada porque possui muitas vantagens sobre outras técnicas, em termos de aplicação industrial. Estes suportes são constituídos de material inerte com boas propriedades mecânicas e baixo custo comercial, além de possuírem elevada porosidade e ampla superfície de adsorção.108 Entretanto, a literatura aponta outros tipos de suportes, como por exemplo, esferas de criogel obtidas a partir do álcool polivinílico (PVA) e esferas de quitosana adicionados de polivinilpirrolidona e polisfosfatos,114-116 esferas de polihidroxibutirato - PHB e terra diatomácea - Celite,55 partículas de bucha vegetal,117-118 esferas de alginato de cálcio,119 membranas de politetrafluoretileno - PTFE,120 conforme apresentado na Tabela 1.
APLICAÇÃO DE LIPASES LIGADAS AO MICÉLIO DE FUNGOS FILAMENTOSOS EM PROCESSOS DE BIOTRANSFORMAÇÃO Síntese de ésteres Ésteres de sacarídeos e ácidos graxos são surfactantes não-iônicos naturais, com propriedades prebióticas e aplicações na produção de alimentos (gordura dietética), como emulsificante, agente antimicrobiano e revestimento de proteção para frutas, dentre outros exemplos. Em 2004, Antczak et al.,114 produziram os ésteres de açúcar caprilato de glicose, caprilato de sacarose, oleato de sacarose e linoleato de sacarose por células íntegras de Mucor circinelloides imobilizadas em criogel-PVA e meio reacional composto pelos açúcares e ácidos graxos solubilizados em mistura de éteres de di-n-pentila e de petróleo, adicionado de antioxidantes naturais. Os autores avaliaram a atividade de água do meio reacional e a otimização da composição do meio reacional e os rendimentos em ésteres produzidos variaram de 0 a 66%, aproximadamente. Lipase ligada ao micélio de Rhizopus chinensis mostrou atividade sintética superior a outras dez lipases comerciais, na esterificação direta do etanol com diferentes ácidos carboxílicos (C2 a C8). O tamanho da cadeia do ácido carboxílico influenciou o grau de esterificação, sendo obtido o valor mais elevado (98,5%) para o ácido hexanóico a 40 ºC adotando relação molar ácido:etanol de 1:1,3.132 Em 2003, Torres et al.,84 isolaram um total de 94 fungos endofíticos a partir 27 amostras de plantas provenientes do Mediterrâneo. Os autores verificaram que 80% das cepas isoladas se mostraram como potenciais fontes de lipase. Os micélios liofilizados destas cepas foram submetidos à esterificação para produção de oleato de isopropila e a cepa codificada como 10A foi a que apresentou melhor resultado, sendo posteriormente identificada como Rhizopus oryzae. Visando avaliar o potencial deste biocatalisador em reação de esterificação de ácidos graxos, os autores verificaram que a atividade catalítica foi inversamente proporcional ao conteúdo de água adicionada ao meio reacional. Ainda foi verificado que a enzima se manteve ativa em uma faixa de pH de 3,0 a 8,0, com atividade máxima entre 4,0 e 7,0, além de mostrar termoestabilidade, com máxima atuação à 60 ºC. Recentemente, Zanotto et al.,85 isolaram fungos de plantas da Região Amazônica, Brasil, os quais foram avaliados como fontes de lipase ligada ao micélio, visando aplicação em reações de biotransformação. Neste estudo, foram isolados um total de 212 fungos, sendo 77 fungos endofíticos, 45 fungos fitopatogênicos e 90 fungos degradadores de madeira, extraídos de fontes como sementes, frutas, hastes, folhas e raízes. Após seleção prévia, 64 fungos potencialmente produtores de lipase foram submetidos à reações de síntese do oleato de pentila e linoleato de dodecila (Figura 5 A-C). Os resultados das reações de esterificação e transesterificação em meio orgânico mostraram que as lipases ligadas ao micélio dos isolados UEA_001, UEA_007 e UEA_115 foram capazes de sintetizar oleato de pentila, linoleato de dodecila e palmitato de etila, com rendimentos que variaram de 87 a 98%.
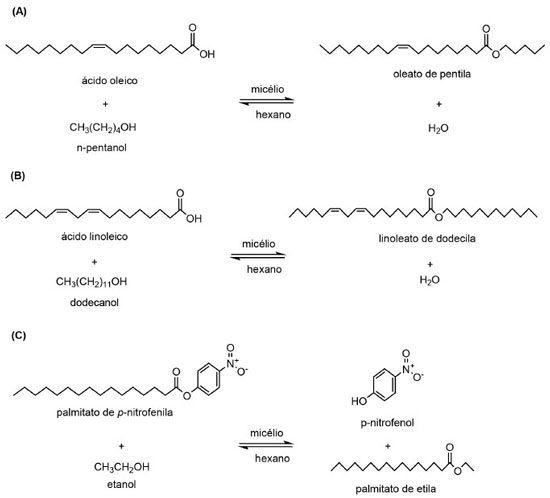 Figura 5. Reações de (A) esterificação de ácido oleico com n-pentanol e de (B) ácido linoleico com dedocanol e (C) transesterificação de palmitato de p-nitrofenila com etanol, catalisadas por lipases ligadas ao micélio de fungos isolados da Região Amazônica (Adaptado de Zanotto et al.,85)
Em seu estudo, Solarte et al.,133 reportaram que células íntegras de uma cepa selvagem de Aspergillus flavus, permitiu o preparo de laurato de etila, com rendimento de 96% (Figura 6A) e uma mistura de acrilato de 2-cloro-1-(cloro metil)etila e acrilato de 2,3-dicloro-1-propila, com rendimento similar ao encontrado quando utilizadas lipases comerciais (Figura 6B).
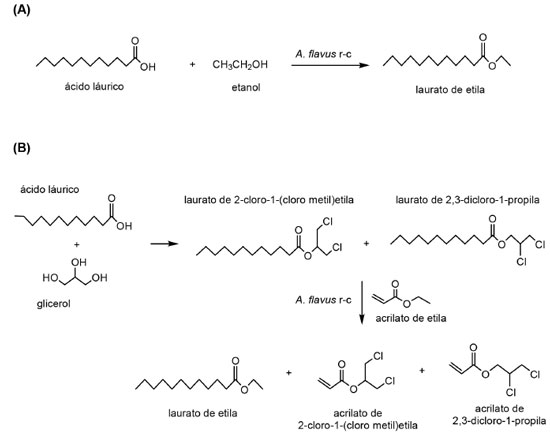 Figura 6. (A) Síntese de laurato de etila e (B) obtenção de uma mistura de régio-isômeros por células íntegras de Aspergillus flavus (Adaptado de Solarte et al.,133)
Resolução cinética Os mesmos fungos isolados por Zanotto et al.,85 testados para síntese de ésteres, foram avaliados na resolução cinética do racemato (R,S)-2-octanol, sendo verificado que as cepas UEA_053 e UEA_115 mostraram capacidades enantiosseletivas promissoras. Com o fungo UEA_053, foram obtidos valores de excessos enantioméricos eesubstrato de 22%, eeproduto de 99%, porcentagem de conversão (c) de 18% e razão enantiomérica (E) superior a 200, enquanto utilizando células íntegras de UEA_115 os resultados obtidos foram 29%, 99%, 22% e superior a 200, respectivamente (Figura 7).
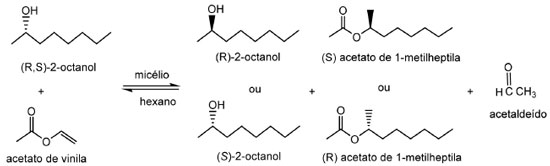 Figura 7. Resolução de (R,S)-2-octanol com acetato de vinila, catalisadas por lipases ligadas ao micélio de fungos isolados da Região Amazônica (Adaptado de Zanotto et al.,85)
Alta preferência enantiomérica da lipase ligada ao micélio de Aspergillus flavus permitiu o preparo de (R)-1-fenilacetato de etila a partir da esterificação de rac-1-feniletanol (Figura 8). O produto foi formado com um excesso enantiomérico (ee) de 94,6% em 24 h de reação a 40 ºC e (S)-1-feniletanol permaneceu no meio reacional com um valor de ee superior a 99%.133
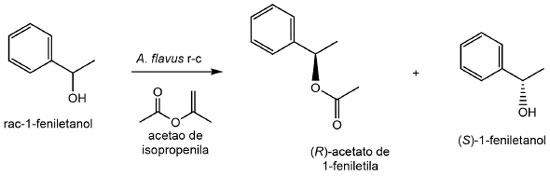 Figura 8. Resolução cinética do rac-1-feniletanol por lipase ligada ao micélio de Aspergillus flavus (Adaptado de Solarte et al.,133)
Biodiesel O uso de lipase ligada ao micélio como biocatalisador na produção de biodiesel tem atraído considerável atenção, principalmente porque evita o custo elevado de preparação da enzima purificada. O biodiesel é produzido por meio da reação de transesterificação de triacilgliceróis (óleos e gorduras) com álcool de cadeia curta (metanol ou etanol). A transesterificação dos triacilgliceróis (TGA) catalisada por lipase é realizada em três reações sucessivas: TGA a diacilglicerol (DAG), DAG a monoacilglicerol (MAG) e MAG em éster alquílico gerando glicerol como coproduto, sendo que cada etapa de reação libera uma molécula de interesse (éster alquílicos). A clivagem enzimática inicial do TAG para DAG pela lipase parece ser a etapa limitante das três reações reversíveis, porque a clivagem do DAG é mais rápida que a reação anterior. A migração do grupo acila de 1,2-DAG para 1,3-DAG e 2-MAG para 1-MAG, que precedem a reação, promovem um rendimento elevado de biodiesel (Figura 9).134
 Figura 9. Esquema da transesterificação de TGA catalisada por lipase 1,3-específica com a migração do grupo acila (Adaptado de Li et al.,134)
As lipases sn-1,3-específicas, tais como Rhizopus oryzae,135 e Mucor circinelloides,64 catalisam eficientemente a transesterificação do óleo vegetal para ésteres de etila com rendimentos superiores a 90%. Este elevado rendimento de transesterificação enzimática resulta da migração de resíduos acila para posições específicas na molécula de glicerol, pois mesmo sendo enzimas sn-1,3-específica, estas lipases são eficientes para clivar ácidos graxos de TAG, nas posições sn-1,3 promovendo a migração de resíduos acila da posição sn-2 para posições terminais (sn-1 e sn-3) no glicerol.136 Por outro lado, a migração de grupo acila pode ser influenciada pela polaridade do solvente usado na reação de transesterificação. De acordo com Li et al.,134 a redução da polaridade do solvente aumenta as constantes de velocidade de migração do grupo acila devido à influência favorável da dispersão da carga sobre o estado de transição, conduzindo a rendimentos variados de biodiesel. É crescente o número de trabalhos publicados na literatura a respeito da utilização de células íntegras de fungos filamentosos imobilizados na produção de biodiesel. Apesar da utilização das células íntegras reduzir as etapas de obtenção do biocatalisador purificado, acarretando redução do custo final do produto, alguns inconvenientes devem ser levados em consideração. As células íntegras possuem baixa estabilidade operacional em sistemas livres de solvente.137 Além disto, a imobilização celular torna a síntese do biodiesel mais lenta do que no processo em que se emprega lipase purificada.138 Neste aspecto, diversos estudos estão sendo realizados com o objetivo de promover a estabilidade operacional das células íntegras de forma a reduzir a desativação da lipase ligada ao micélio, causada pelo excesso de metanol no meio reacional, além de propor novas estratégias de processo visando o aumento da eficiência de conversão e ganhos em produtividade. A Tabela 2 apresenta alguns exemplos. A grande maioria dos trabalhos publicados reporta o emprego de células íntegras de R. oryzae imobilizadas em partículas de espuma de poliuretano. Este fato se deve a comprovada eficiência catalítica da lipase deste fungo filamentoso juntamente com a simplicidade operacional da técnica de imobilização. Entretanto, existem relatos de outros tipos de fungos filamentosos e de suportes também avaliados para a produção de biodiesel.
CONCLUSÕES O uso de células íntegras de fungos filamentosos com atividade lipolítica ligada ao micélio vem despertando grande interesse de investigação pelo seu potencial de uso na biotecnologia, pois sua aplicação pode reduzir consideravelmente os custos de um processo enzimático. Em especial, a utilização de células íntegras imobilizadas em suportes porosos de baixo custo como biocatalisadores em reações de biotransformação tem revelado resultados promissores de rendimento em produto. A simplicidade da técnica de imobilização, que se baseia na habilidade natural de adesão celular ao suporte durante o crescimento, e a possibilidade de utilização do biocatalisador em processos contínuos, confere as células íntegras imobilizadas vantagens do ponto de vista comercial e industrial.
AGRADECIMENTOS À CAPES (Coordenação de Aperfeiçoamento de Pessoal de Ensino Superior) e ao CNPq (Conselho Nacional de Desenvolvimento Científico e Tecnológico), pelo apoio financeiro.
REFERÊNCIAS 1. Hernáiz, M. J.; Alcántara, A. R.; García, J. I.; Sinisterra, J. V.; Chem.- Eur. J. 2010, 16, 9422. 2. Vasic-Racki, D.; Em Industrial Biotransformations; Liese, A.; Seelbach, K.; Wandrey, C., eds.; Wiley-VCH: Weinheim, 2006, cap. 1. 3. Perkins, C.; Siddiqui, S.; Puri, M.; Demain, A. L.; Crit. Rev. Biotechnol. 2015, 18, 1. 4. Faber, K.; Biotransformation in Organic Chemistry, 6th ed., Springer-Verlag: Heidelberg, 2011. 5. Held, M.; Schmid, A.; van Beilen, J. B.; Witholt, B.; Pure Appl. Chem. 2000, 72, 1337. 6. Borriss, R. Em Biotechnology volume 7a: Enzyme Technology; Rehm, H.-J.; Reed, G., eds.; Wiley-VCH: Weinheim, 1987, cap. 2. 7. Kaushik, N.; Biswas, S.; Singh, J.; The Scitech Journal 2014, 1, 15. 8. Edwards, D. R.; Lohman, D. C.; Wolfenden, R.; J. Am. Chem. Soc. 2012, 134, 525. 9. Fukui, S.; Tanaka, A.; Ann. Rev. Microbiol. 1982, 36, 145. 10. Přenosil, J. E.; Dunn, I. J.; Heinzle, E. Em Biotechnology volume 7a: Enzyme Technology; Rehm, H.-J.; Reed, G., eds.; Wiley-VCH: Weinheim, 1987, cap. 10. 11. Brodelius, P.; Vandamme, E. J. Em Biotechnology volume 7a: Enzyme Technology; Rehm, H.-J.; Reed, G., eds.; Wiley-VCH: Weinheim, 1987, cap. 8. 12. Milner, S. E.; Maguire, A. R.; Arkivoc 2012, i, 321. 13. Kapoor, M.; Gupta, M. N.; Process Biochem. 2012, 47, 555. 14. Daiha, K. G.; Angeli, R.; de Oliveira, S. D.; Almeida, R. V.; PLoS One (2015), doi: 10.1371/journal.pone.0131624. 15. Ollis, D. L.; Cheah, E.; Cygler, M.; Dijkstra, B.; Frolow, F.; Franken, S. M.; Harel, M.; Remington, S. J.; Silman, I.; Schrag, J.; Sussman, J. L.; Verschueren, K. H. G.; Goldman, A.; Protein Eng. 1992, 5, 197. 16. Kazlauskas,R. J.; Bornscheuer, U. T. Em Biotechnology: a multi volume comprehensive treatise; Rehm, H.J., Reed, G., Pühler, A., Stadler, P.J.W., eds., vol. 8ª: Biotransformations I; Kelly, D. R., ed.; Wiley-VCH: Weinheim, 1998, cap. 3. 17. Petersen, S. B. Em Engineering of/with lipases; Malcata, F. X. ed.; Kluwer Academic Publishers:Netherlands, 1996. 18. Brzozowski, A. M.; Nature 1991, 351, 491. 19. Verger, R.; Trends Biotechnol. 1997, 15, 32. 20. Balcão, V. M.; Paiva, A. L.; Malcata, F. X.; Enzyme Microb. Technol. 1996, 18, 392. 21. Vulfson, E. N. Em Lipases - their structure, biochemistry and application; Woolley, P.; Petersen, S. B., eds.; Cambridge University Press: Cambridge, 1994, cap. 13. 22. Fregolente, P. B. L.; Pinto, G. M. F.; Wolf-Maciel, M. R.; Maciel Filho, R.; Batistella, C. B.; Quim. Nova 2009, 32, 1539. 23. de Castro, H. F.; Mendes, A. A.; Santos, J. C.; Aguiar, C. L.; Quim. Nova 2004, 27, 146. 24. Khan, N. R.; Rathod, V. K.; Process Biochem. 2015, 50, 1793. 25. Aires-Barros, M. R.; Taipa, M. Â.; Cabral, J. M. S. Em Lipases - their structure, biochemistry and application; Woolley, P.; Petersen, S. B., eds.; Cambridge University Press: Cambridge, 1994, cap. 12. 26. Bonugli-Santos, R. C.; Vasconcelos, M. R. S.; Passarini, M. R. Z.; Vieira, G. A. L.; Lopes, V. C. P.; Mainardi, P. H.; dos Santos, J. A.; Duarte, L. A.; Otero, I. V. R.; Yoshida, A. M. S.; Feitosa, V. A.; Pessoa Junior, A.; Sette, L. D.; Front. Microbiol. (2015), doi: 10.3389/fmicb.2015.00269. 27. Nobre, F. S. Tese de Doutorado, Universidade de São Paulo, Brasil, 2012. 28. Duarte, A. W. F.; Dayo-Owoyemi, I.; Nobre, F. S.; Pagnocca, F. C.; Chaud, L. C. S.; Pessoa, A.; Felipe, M. G. A.; Sette, L. D.; Extremophiles 2013, 17, 1023. 29. Sperb, J. G. C.; Costa, T. M.; Vaz, D. A.; Valle, J. A. B.; Valle, R. C. S. C.; Tavares, L. B. B.; Engevista 2015, 17, 385. 30. Facchin, S.; Alves, P. D. D.; Siqueira, F. F.; Barroca, T. M.; Victória, J. M. N.; Kalapothakis, E.; Open Journal of Ecology 2013, 3, 34. 31. Nwuche, C. O.; Ogbonna, J. C.; Braz. Arch. Biol. Technol. 2011, 54, 113. 32. Oliveira, A. C. D.; Fernandes, M. L.; Mariano, A. B.; Braz. J. Microbiol. 2014, 45, 1503. 33. Ribeiro Junior, J. C.; Lima, J. B. A.; de Lemos, K. L.; da Silva, L. C. C.; Tamanini, R.; Beloti, V.; Semina: Cienc. Agrar. 2015, 36, 4289. 34. Meyer, V.; Biotechnol. Adv. 2008, 26, 177. 35. Torres, F. A. G.; de Moraes, L. M. P.; De Marco, J. L.; Poças-Fonseca, M. J.; Felipe, M. S. S. Em Enzimas em biotecnologia. Produção, aplicação e mercado; Bon, E. P. S.; Ferrara, M.A.; Corvo, M. L.; Vermelho, A. B.; Paiva, C. L. A.; de Alencastro, R. B.; Coelho, R. R. R., organizadores; Editora Interciência: Rio de Janeiro, 2008, cap. 3. 36. Rapp, P.; Backhaus, S.; Enzyme Microb. Technol. 1992, 14, 938. 37. Dheeman, D. S.; Antony-Babu, S.; Frías, J. M.; Henehan, G. T. M.; J. Mol. Catal. B: Enzym. 2011, 72, 256. 38. Stöcklein, W.; Sztajer, H.; Menge, U.; Schmid, R.D.; Biochim. Biophys. Acta 1993, 1168, 181. 39. Ortiz-Vázquez, E.; Granados-Baeza, M.; Rivera-Muñoz, G.; Biotechnol. Adv. 1993, 11, 409. 40. de la Torre, M.E.; Diaz, A.; Ruiz, B.; Farres, A.; Aramburo, C.; Sanchez, S.; J. Ind. Microbiol. 1996, 17, 73. 41. Sugihara, A.; Shimada, Y.; Takada, N.; Nagao, T.; Tominaga, Y.; J. Ferment. Bioeng. 1996, 82, 498. 42. Yadav, R. P.; Saxena, R. K.; Gupta, R.; Davidson, S. Folia Microbiol. 1998, 43, 373. 43. Gombert, A. K.; Pinto, A. L.; Castilho, L. R.; Freire, D. M. G. Process Biochem. 1999, 35, 85. 44. Lima, V. M. G.; Krieger, N.; Mitchel, D. A.; Fontana, J. D.; Biochem. Eng. J. 2004, 18, 65. 45. Tan, T.; Zhang, M.; Xu, J.; Zhang, J. Process Biochem. 2004, 39, 1495 46. Godoy, M. G.; Gutarra, M. L. E.; Maciel, F. M.; Felix, S. P.; Bevilaqua, J. V.; Machado, O. L. T.; Freire, D. M. G. Enzyme Microb. Technol. 2009, 44, 317. 47. Gutarra, M. L. E.; Godoy, M. G.; Maugeri, F.; Rodrigues M. I.; Freire, D. M. G.; Castilho, L. R.; Bioresour. Technol. 2009, 100, 5249. 48. Kumar, S.; Katiyar, N.; Ingle, P.; Negi, S.; Bioresour. Technol. 2011, 102, 4909. 49. Kumar, S.; Mathur, A.; Singh, V.; Nandy, S.; Khare, S. K.; Negi, S.; Bioresour. Technol. 2012, 120, 300. 50. Cardenas, F.; Alvarez, E.; de Castro-Alvarez, M.-S.; Sanchez-Montero, J.-M.; Valmaseda, M.; Elson, S. W.; Sinisterra, J.-V.; J. Mol. Catal. B: Enzym. 2001a, 14, 111. 51. Cardenas, F.; de Castro, M. S.; Sanchez-Montero, J. M.; Sinisterra, J. V.; Valmaseda, M.; Elson, S. W.; Alvarez, E.; Enzyme Microb. Technol. 2001b, 28, 145. 52. Baffi, M. A.; Romo-Sánchez, S.; Úbera-Iranzo, J.; Briones-Pérez, A. I.; New Biotechnol. 2012, 29, 451. 53. Miranda, O. A.; Salgueiro, A.A.; Pimentel, M. C. B.; Lima Filho, J. L.; Melo, E. H. M.; Durán, N.; Bioresour. Technol. 1999, 69, 145. 54. D'Annibale, A.; Sermanni, G. G.; Federici, F.; Petruccioli, M.; Bioresour. Technol. 2006, 97, 1828. 55. Andrade, G. S. S.; Freitas, L.; Oliveira, P. C.; de Castro, H. F.; J. Mol. Catal. B: Enzym. 2012, 84, 183. 56. Marotti, B. S.; Gonçalves, D. B.; Cortez, D. V.; de Castro, H. F.; XX Simpósio Nacional de Bioprocessos, XI Simpósio de Hidrólise Enzimática de Biomassa, Fortaleza, Brasil, 2015, 10.17648/sinaferm-2015-33518. 57. Shimada, Y.; Koga, C.; Sugihara, A.; Nagao, T.; Takada, N.; Tsunasawa, S.; Tominaga, Y.; J. Ferment. Bioeng. 1993, 75, 349. 58. Rapp, P.; Enzyme Microb. Technol. 1995, 17, 832. 59. Nguyen, L. N.; Dao, T. T.; Živković, T.; Fehrholz, M.; Schäfer, W.; Enzyme Microb. Technol. 2010, 46, 479. 60. Hernández-Rodríguez, B.; Córdova, J.; Bárzana, E.; Favela-Torres, E.; J. Mol. Catal. B: Enzym. 2009, 61, 136. 61. Bancerz, R.; Osińska-Jaroszuk, M.; Jaszek, M.; Janusz, G.; Stefaniuk, D.; Sulej, J.; Janczarekm, M.; Jarosz-Wilkolazka, A.; Rogalski, J.; Biotechnol. Appl. Biochem. 2015. 1. 62. Andrade, G. S. S.; Carvalho, Ana K. F.; Romero, C. M.; Oliveira, P. C.; de Castro, H. F. Bioprocess Biosyst. Eng. 2014, 37, 2539. 63. Faria, E. L. P.; Cortez, D. V.; Oliveira, P. C.; de Castro, H. F.; XX Simpósio Nacional de Bioprocessos, XI Simpósio de Hidrólise Enzimática de Biomassas, Fortaleza, Brasil, 2015, 10.17648/sinaferm-2015-33476. 64. Carvalho, A. K. F. Tese de Doutorado, Universidade de São Paulo, Brasil, 2015. 65. Marchut-Mikolajczyk, O.; Kwapisz, E.; Wieczorek, D.; Antczak, T.; Int. Biodeterior. Biodegrad. 2015, 104, 142. 66. Ülker, S.; Karaoğlu, S. A.; J. Biosci. Bioeng. 2012, 114, 385. 67. Ionita, A.; Moscovici, M.; Popa, C.; Vamanu, A.; Popa, O.; Dinu, L.; J. Mol. Catal. B: Enzym. 1997, 3, 147. 68. Mhetras, N. C.; Bastawde, K. B.; Gokhale, D. V.; Bioresour. Technol. 2009, 100, 1486. 69. Romero, C. M.; Pera, L. M.; Loto, F.; Vallejos, C.; Castro, G.; Baigori, M. D.; Biocatal. Agric. Biotechnol. 2012a, 1, 25. 70. Romero, C. M.; Pera, L. M.; Olivaro, C.; Vazquez, A.; Baigori, M. D.; Fuel Process. Technol. 2012b, 98, 23. 71. Colla, L. M.; Rizzardi, J.; Pinto, M. H.; Reinehr, C. O.; Bertolin, T. E.; Costa, J. A. V.; Bioresour. Technol. 2010, 101, 8308. 72. Basheer, S. M.; Chellappan, S.; Beena, P. S.; Sukumaran, R. K.; Elyas, K. K.; New Biotechnol. 2011, 28, 627. 73. Pereira, M. G.; Vici, A. C.; Facchini, F. D. A.; Tristão, A. P.; Cursino-Santos, J. R.; Sanches, P. R.; Jorge, J. A.; Polizeli, M. L. T. M.; Biocatal. Biotransform. 2014, 32, 74. 74. Dobrev, G.; Zhekova, B.; Dobreva, V.; Strinska, H.; Doykina, P.; Krastanov, A.; Biocatal. Agric. Biotechnol. 2015, 4, 77. 75. Morinaga, T.; Kanda, S.; Nomi, R.; J. Ferment. Technol. 1986, 64, 451. 76. Kmecz, I.; Simándi, B.; Poppe, L.; Juvancz, Z.; Renner, K.; Bódai, V.; Tőke, E. R.; Csajági, C.; Sawinsky, J.; Biochem. Eng. J. 2006, 28, 275. 77. Romdhane, I. B.-B.; Fendri, A.; Gargouri, Y.; Gargouri, A.; Belghith H.; Biochem. Eng. J. 2010, 53, 112. 78. Supakdamrongkul, P.; Bhumiratana, A.; Wiwat, C.; J. Invertebr. Pathol. 2010, 105, 228. 79. Jermsuntiea, W.; Aki, T.; Toyoura, R.; Iwashita, K.; Kawamoto, S.; Ono, K.; New Biotechnol. 2011, 28, 158. 80. Florczak, T.; Daroch, M.; Wilkinson, M. C.; Białkowska, A.; Bates, A. D.; Turkiewickz, M.; Iwanejko, L. A.; Enzyme Microb. Technol. 2013, 53, 18. 81. Cazarolli, J. C.; Guzatto, R.; Samios, D.; Peralba, M.C. R.; Cavalcanti, E. H. S.; Bento, F.M.; Int. Biodeterior. Biodegrad. 2014, 95, 364. 82. Azevedo, J. L.; Araujo, W. L. Em Fungi multifaceted microbes; Ganguli, B. N.; Deshmukh, S. K. eds.; Anamaya Publishers: Nova Délhi, 2007, cap. 12. 83. Orlandelli, R. C.; Specian, V.; Felber, A. C.; Pamphile, J. A.; SaBios: Rev. Saúde e Biol. 2012, 7, 97. 84. Torres, M.; Dolcet, M. M.; Sala, N.; Canela, R.; J. Agric. Food Chem. 2003, 51, 3328. 85. Zanotto, S. P.; Romano, I. P.; Lisboa, L. U. S.; Duvoisin Jr., S.; Martins, M. K.; Lima, F. A.; Silva, S. F.; Albuquerque, P. M.; J. Braz. Chem. Soc. 2009, 20, 1046. 86. Nascimento, C. S.; dos Santos, V. L.; Andrade, M.H.C.; Trabalhos completos do XX Congresso Brasileiro de Engenharia Química, Florianópolis, Brasil, 2014. 87. Venkatesagowda, B.; Ponugupaty, E.; Barbosa, A. M.; Dekker R. F. H.; World J. Microbiol. Biotechnol. 2012, 28, 71. 88. Gulhane, P. A.; Gomashe, A. V.; Patne, M. K.; International Journal of Recent Scientific Research 2016, 7, 8226. 89. Amirita, A.; Sindhu, P.; Swetha, J.; Vasanthi, N. S.; Kannan, K. P.; World J. Sci. Technol. 2012, 2, 13. 90. Sunitha, V. H.; Nirmala Devi, D.; Srinivas C.; World J. Agric. Sci. 2013, 9, 1. 91. Silva, R. L. O.; Luz, J. S.; Silveira, E. B.; Cavalcante, U. M. T.; Acta bot. bras. 2006, 20, 649. 92. Colen, G.; Junqueira, R. G.; Moraes-Santos, T.; World J. Microbiol. Biotechnol. 2006, 22, 881. 93. Frost, G. M.; Moss, D. A. Em Biotechnology volume 7a: Enzyme Technology; Rehm, H.-J.; Reed, G., eds.; Wiley-VCH: Weinheim, 1987, cap. 3. 94. Tsujisaka, Y.; Iwai, M.; Fukumoto, J.; Okamoto, Y; Agric. Biol. Chem. 1973, 37, 837. 95. Loo, J. L.; Khoramnia, A.; Lai, O. M.; Long, K.; Ghazali, H. M.; Molecules 2014, 19, 8556. 96. Hama, S.; Tamalampudi, S.; Fukumizu, T.; Miura, K.; Yamaji, H.; Kondo, A.; Fukuda, H.; J. Biosci. Bioeng. 2006, 101, 328. 97. Galvagno, M. A.; Forchiassin, F. Em Fungos: uma introdução à biologia, bioquímica e biotecnologia; Esposito, E.; Azevedo, J. L., organizadores; Educs: Caxias do Sul, 2010. 98. Sant'Anna Jr, G. L. Em Biotecnologia Industrial. Volume 3: Processos fermentativos e enzimáticos; Lima, U. A.; Aquarone, E.; Borzani, W.; Schmidell W., coordenadores; Edgard Blücher: São Paulo, 2005, cap. 14. 99. Bon, E. P. S.; Pereira Jr, N.; Gottschalk, L. M. F.; Sá-Pereira, P.; Roseiro, J. C.; Ferrara, M. A. Em Enzimas em biotecnologia. Produção, aplicação e mercado; Bon, E. P. S., Ferrara, M. A., Corvo, M. L., Vermelho, A. B., Paiva, C. L. A., de Alencastro, R. B., Coelho, R. R. R., orgs; Editora Interciência: Rio de Janeiro, 2008, cap. 5. 100. Zarevúcka, M.; Em Olive Oil - Constituents, Quality, Health Properties and Bioconversions; Boskou, D., ed.; InTech: Rijeka, 2012, cap. 25. 101. Essamri, M.; Deyris, V.; Comeau, L.; J. Biotechnol. 1998, 60, 97. 102. Yang, X.; Wang, B.; Cui, F.; Tan, T.; Process Biochem. 2005, 40, 2095. 103. Zeng, J.; Du, W.; Liu, X.; Liu, D.; Dai, L.; J. Mol. Catal. B: Enzym. 2006, 43, 15. 104. Wang, D.; Xu, Y.; Shan, T.; Biochem. Eng. J. 2008, 41, 30. 105. Hama, S.; Yamaji, H.; Kaieda, M.; Oda, M.; Kondo, A.; Fukuda, H.; Biochem. Eng. J. 2004, 21, 155. 106. Sun, T.; Du, W.; Zeng, J.; Dai, L.; Liu, D.; Process Biochem. 2010, 45, 514. 107. Teng, Y.; Xu, Y.; Bioresour. Technol. 2008, 99, 3900. 108. de Ory, I.; Cabrera, G.; Ramirez, M.; Blandino, A.; Em Methods in Biotechnology: Immobilization of Enzymes and Cells; Guisan, J. M., ed.; Humana Press Inc.:Totowa, 2006, cap. 31. 109. Jack, T. R.; Jazic, J. E. Em Advances in Biochemical Engineering - volume 5; Ghose, T. K.; Fiechter, A.; Blakerbrough, N., eds.; Springer-Verlag: Berlin Heidelberg, 1977, cap. 6. 110. Karel, S. F.; Libicki, S. B.; Robertson, C. R.; Chem. Eng. Sci. 1985, 40, 1321. 111. Canilha, L.; de Carvalho, W.; de Almeida e Silva, J. B.; Biotecnologia Ciência & Desenvolvimento 2006, 36, 48. 112. Covizzi, L. G.; Giese, E. C.; Gomes, E.; Dekker, R. F. H.; da Silva, R.; Semina: Cienc. Exatas Tecnol. 2007, 28, 143. 113. Ramakrishna, S. V.; Prakasham, R. S.; Curr. Sci. 1999, 77, 87. 114. Antczak, T.; Patura, J.; Szczȩsna-Antczak, M.; Hiler, D.; Bielecki, S.; J. Mol. Catal. B: Enzym. 2004, 29, 155. 115. Szczȩsna-Antczak, M.; , Anczak, T.; Rzyska, M.; Bielecki, S.; J. Mol. Catal. B: Enzym. 2002, 19-20, 261. 116. Szczȩsna-Antczak, M.; Antczak, T.; Rzyska, M.; Modrzejewska, Z.; Patura, J.; Kalinowska, H.; Bielecki, S.; J. Mol. Catal. B: Enzym. 2004, 29, 163. 117. Ferrarezi, A. L.; Ohe, T. H. K.; Borges, J. P.; Brito, R. R.; Siqueira, M. R.; Vendramini, P. H.; Quilles Jr., J. C.; Nunes, C. C. C.; Bonilla-Rodriguez, G. O.; Boscolo, M.; Da-Silva, R.; Gomes, E.; J. Mol. Catal. B: Enzym. 2014, 107, 106. 118. Sattari, S.; Vahabzadeh, F.; Aghtaei, H. K.; Braz. J. Chem. Eng. 2015, 32, 367. 119. Peart, P. C.; Reynolds, W. F.; Reese, P. B.; J. Mol. Catal. B: Enzym. 2016, 125, 16. 120. Ahmed, E. M.; Braz. J. Microbiol. 2007, 38, 224. 121. Arabmarkadeh, A.; Jalili, F.; Chegini, P.; Nationalpark-Forschung in der Schweiz (Switzerland Research Park Journal) 2014, 103, 671. 122. Oda, M.; Kaieda, M.; Hama, S.; Yamaji, H.; Kondo, A.; Izumoto, E.; Fukuda, H.; Biochem. Eng. J. 2005, 23, 45. 123. Hama, S.; Yamaji, H.; Fukumizu, T.; Numata, T.; Tamalampudi, S.; Kondo, A.; Noda, H.; Fukuda, H.; Biochem. Eng. J. 2007, 34, 273. 124. Arai, S.; Nakashima, K.; Tanino, T.; Ogino, C.; Kondo, A.; Fukuda, H.; Enzyme Microb. Technol. 2010, 46, 51. 125. Yoshida, A.; Hama, S.; Tamadani, N.; Fukuda, H.; Kondo, A.; Biochem. Eng. J. 2012, 63, 73. 126. Kyeong, J. S.; Yeom, S. H.; Bioprocess Biosyst. Eng. 2014, 37, 2189. 127. Ahmed, E. M.; Ammar, M. I.; Journal of Advancement in Medical and Life Sciences, (2015), doi: 10.15297/JALS.V3I1.02. 128. Chen, J.-P.; Lin, G.-H.; Appl. Biochem. Biotechnol. 2010, 161, 181. 129. Zhou, G.; Chen, G.; Yan, B.; Biotechnol. Lett. 2015, 37, 1959. 130. He, Q.; Shi, H.; Gu, H.; Naka, G.; Ding, H.; Li, X.; Zhang, Y.; Hu, B.; Wang, F. BioResources 2016, 11, 850. 131. Ganesan, D.; Rajendran, A.; Thangavelu, V.; Biomass Conv. Bioref. 2012, 2, 11. 132. Xu, Y.; Wang, D.; Mu, X. Q.; Zhao, G. A.; Zhang, K. C.; J. Mol. Catal. B: Enzym. 2002, 18, 29. 133. Solarte, C.; Yara-Varón, E.; Eras, J.; Torres, M.; Balcells, M.; Canela-Garayoa, R.; J. Mol. Catal. B: Enzym. 2014, 100, 78. 134. Li, W.; Du, W.; Li, Q.; Li, R.-W.; Liu, D.; Bioresour. Technol. 2010, 101, 5737. 135. Ban, K.; Hama, S.; Nishizuka, K.; Kaieda, M.; Matsumoto, T.; Kondo, A.; Noda, H.; Fukuda, H.; J. Mol. Catal. B: Enzym. 2002, 17, 157. 136. Antczak, M. S.; Kubiak, A.; Antczak, T.; Bielecki, S.; Renewable Energy 2009, 34, 1185. 137. Li, W.; Du, W.; Liu, D.; Yao, Y.; Biochem. Eng. J. 2008, 41, 111. 138. Robles-Medina, A.; González-Moreno, P. A.; Esteban-Cerdán, L.; Molina-Grima, E.; Biotechnol. Adv. 2009, 27, 398. 139. Arai, S.; Nakashima, K.; Tanino, T.; Ogino, C.; Kondo, A.; Fukuda, H.; Enzyme Microb. Technol. 2010, 46, 51. 140. Talukder, M. M. R.; Lee, H. Z. S.; Low, R. F.; Pei-Lyn, L. C.; Warzecha, D.; Wu, J.; J. Mol. Catal. B: Enzym. 2013, 89, 108. 141. Xiao, M.; Mathew, S.; Obbard, P. A.; Global Change Biology Bioenergy 2010, 2, 45. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access