Artigo
|
|
| Biodegradacion fungica de poliuretano rígido Fungal biodegradation of rigid polyurethane |
|
Araceli LoredoI,*; Andres ArgüelloII; Raul Rodríguez-HerreraII; Gerardo Gutiérrez-SánchezIII; Alondra EscamillaIV; Cristóbal AguilarII
IInstituto Politécnico Nacional, Centro Interdisciplinario de Investigación para el Desarrollo Integral Regional, Unidad Durango, 34220, Durango, México Recebido em 22/11/2016 *e-mail: alotre@gmail.com Polyurethane (PU) is one of the most widely used polymer materials. This generates a serious environmental problem because of its poor biodegradability since most of the native microorganisms cannot use it as a source of nutrients. Several techniques involving mechanical and chemical processes have been used for its recovery and reuse; however, the problem of contamination has not been solved, and recently research has focused on developing strategies that use different microorganisms for the biodegradation of PU. In this work, the biodegradation and enzymatic capacity of three fungal strains were evaluated. Thermogravimetric and infrared spectrophotometry analyzes were used to evaluate the ability of the three fungal strains to biodegrade PU and also their enzymatic activities were studied. It was found that a strain of Trichoderma was able to modify the PU structure enough that the changes were detected by both TGA and FTIR. These changes can be caused by urease activity that breaks down carbon-nitrogen bonds in the structure because the signals suggesting the formation of carboxylic acids and decreasing corresponding to carbon-nitrogen bonds of the urethane bond are increased. INTRODUCCION En el mundo, los polímeros son los principales responsables del deterioro del medio ambiente.1 Dentro de estos, los poliuretanos (PU) son ampliamente usados como materia prima en diversas industrias, desde muebles y automóviles, a aislamientos térmicos de barcos y materiales médicos.2 Son sintetizados a partir de polioles y poliisocianatos y se caracterizan por ser insolubles en agua.3 El problema principal radica en su acumulación y no biodegradación por parte de los microorganismos existentes, que no cuentan con estrategias de biodegradación contra esta estructura polimérica.4 Cada año, más de 5 millones de toneladas de residuos que contienen diferentes plásticos y espumas de PU son generados en los Estados Unidos y Canadá,3 por lo que la degradación del PU y el descubrimiento de microorganismos capaces de aprovecharlo como fuerte de carbono o nitrógeno, se ha convertido en un pilar dentro de la investigación científica hacia una química más verde.5 Los polímeros pueden ser sustratos potenciales para microorganismos quimioheterotrofos.6 Se han aislado tanto bacterias como hongos filamentosos capaces de degradar PU.7 Los géneros de hongos encontrados han sido Geomyces pannorum y Nectria sp, principalmente, además de Cylindrocladiella parva, Penicillium inflatum, Plectosphaerella cucumerin y P. inflatum, Alternaria, Penicillium venetum, Neonectria ramulariae y Penicillium viridicatum. Los poliurteanos están conformados por regiones C-C, C-N y C-O y poseen un enlace característico, el enlace uretano, similar al enlace peptídico y a la urea. La principal funcion del enlace uretano es unir cadenas orgánicas de polioles y poliisocianatos, para formar la unidad polimérica del poliuretano, la cual diferencia a cada poliuretano e influencia la manera en la es degradado en el ambiente.8 Diversas investigaciones han empleado microorganismos capaces de aprovechar PU como fuente de carbono o nitrógeno en biodegradaciones fortuitas o mediante procesos co-metabólicos,9 encontrándose que las principales enzimas encargadas de este proceso son proteasas, esterasas y ureasas.8,10 Akutsu et al.10 sugieren que la degradación enzimática de los polímeros por hidrólisis es un proceso en dos pasos: 1) Unión al polímero por medio de un dominio hidrofóbico y 2) Hidrólisis. En la biodegradación del poliuretano se han aislado y caracterizado dos enzimas poliuretanasas (PUasas) bacterianas11 y dos esterasas.12,13 La esterasa unida a membrana se adhiere a la superficie hidrófoba del PU, para que la esterasa extracelular empieze con la hidrólisis, hasta degradar al poliuretano a metabolitos. Algunos autores,11,14 indican que la biodegradación del poliuretano es debida a la hidrólisis de los enlaces éster de las cadenas unidas al enlace uretano y no a la hidrólisis del enlace en sí. La mayoría de los trabajos sobre degradación de PU están enfocados en estudios con bacterias. Sin embargo, en años recientes la investigación se ha centrado en el uso de hongos.15-19 Algunas de las enzimas fúngicas descritas en la década de 1980 fueron dos esterasas de Aspergillus terreus y las actividades atribuidas a ellas fueron endo y exo poliuretanesterasa. Así que considerando que los hongos sólo son capaces de usar fuentes de carbono orgánicas, el poliuretano puede ser empleado como una fuente potencial de carbono para el crecimiento fúngico. El objetivo principal de este estudio fue evaluar las actividades enzimáticas de tres hongos filamentosos, Trichoderma sp DIA-T, Aspergillus ustus PSS y Paecelomyces sp NK12, como posibles candidatos para llevar a cabo la biodegradación de un residuo sólido de PU. Además fue posible rastrear a la enzima responsable de la biodegradación e identificar cambios en la estructura del polímero evaluado. Este trabajó se realizó en la Facultad de Ciencias Químicas de la Universidad Autónoma de Coahuila en Saltillo, Coahuila, México.
PARTE EXPERIMENTAL Poliuretano Las muestras de poliuretano a biodegradar fue sintetizado por la empresa Nemak S.A. de C.V., a partir de metil diisocianato y metil difenol. La muestra de poliuretano rígido fue triturada hasta alcanzar un tamaño de partícula entre 0.5-2 mm y homogenizada. No se sometió a ningún lavado. Microorganismos Los microorganimos empleados fueron Trichoderma sp (DIA-T), Aspergillus ustus (PSS), provistas por el Departamento de Investigación en Alimentos (DIA) de la Universidad Autónoma de Coahuila y Paecelomyces sp (NK12), aislada a partir de una muestra de suelo contaminada con PU en un trabajo previo por este mismo grupo de trabajo en dónde se encontró que son capaces de producir enzimas ureasa, proteasa y esterasa, las cuales están relacionadas con la degradación de poliuretano así como crecer en un medio de cultivo suplmentado con el polímero.20 Condiciones de cultivo Trichoderma sp (DIA-T), Aspergillus ustus (PSS) y Paecelomyces sp (NK12) se emplearon como posibles candidatas para llevar a cabo la biodegradación de PU. El inóculo se preparó incubando las cepas en agar papa-dextrosa a 30 °C por 5 días. Las esporas se cosecharon usando una solución de Tween 80 al 0.1% y fueron contadas empleando una cámara de Neaubauer. Las condiciones de cultivo así como el tamaño del inóculo fueron determinadas en experimentos previos en dónde se encontró que a los 30 días había producción de enzimas hidrolasas por parte de los hongos, por lo que se tomó este tiempo como adecuado para los fines del trabajo de investigación.20 Se emplearon matraces Erlenmeyer con 50 mL del medio de cultivo mineral2the enzyme is heat labile and not inhibited by phenylmethylsulfonylfluoride (PMSF a un pH inicial de 4.5, a 200 rpm, 30 °C e incubados por 30 días y el inóculo fue de 5x105 esporas/mL. Como fuente de carbono alternas se emplearon glucosa y glicerol y como fuente de nitrógeno, urea y sulfato de amonio, en dos relaciones (1:1 y 1:2). Se utilizó un diseño experimental factorial 22 con un total de 8 tratamientos y un Blanco (cultivo sólo con poliuretano). Los cultivos se llevaron a cabo a una concentración de PU molido de 3 g/L. Al final del tiempo de cultivo, los sobrenadantes se filtraron para separar la biomasa y el poliuretano remanente. La biomasa se analizó para determinar el contenido de glucosamina. El PU residual fue analizado mediante Espectrometrìa infrarroja (FTIR) y análisis termogravimetricos (TGA). El líquido restante se empleó para llevar a cabo los determinaciones de las actividades enzimáticas presentes al final del tiempo de cultivo. Análisis bioquímico Diversas metodologías se emplearon para llevar a cabo el análisis bioquimico de las muestras. El contenido de proteína de las muestras fue evaluado empleando el método Bradford;21 el contenido total de fenoles mediante el método propuesto por Makkar et al;22 la actividad ureasa de acuerdo a la metodología propuesta por Witte y Medina-Escobar;23 la actividad esterasa de acuerdo a la propuesto por Coca y Dustet24 y la actividad lacasa con el método reportado por Membrillo et al.25 Para poder monitorear el crecimiento de los hongos evaluados se empleó un método indirecto mediante la determinación del contenido de glucosamina de acuerdo al trabajo de Blix.26 Análisis para la biodegradación del poliuretano El poliuretano fue caracterizado mediante análisis termogravimetrico (TGA) e infrarrojo (FTIR). Los análisis térmicos de TGA se llevaron a cabo empleando una rampa de calentamiento de 25 a 900 °C a 20 °C/min. Los análisis de FTIR se hicieron en un equipo Spectrum GX, Perkin Elmer con una resolución de 4 cm-1 y empleando la técnica de Atenuamiento del Reflejo Total (Attenuated Total Reflection, ATR) (SenrIR, número de parte 071-1305). Los escaneos se promediaron para obtener cada espectro. Es importante mencionar que el PU fue sintetizado a partir de precursores grado industrial que contenían principalmente metil difenil diisocianato (MDI) y metil difenol pero la estructura química exacta no se conoce debido a secreto de la empresa Nemak S.A. de C.V. Todos los análisis se realizaron por triplicado y todos los demás reactivos usados fueron grado analítico. En la Figura 1 se muestra la estructura de los precursores y la posible estructura del poliuretano usado.
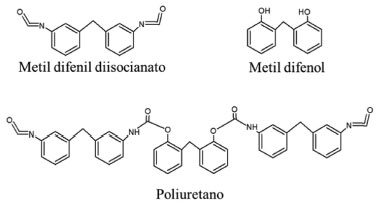 Figura 1. Estructuras de Metil difenil diisocianto, metil difenol y poliuretano
RESULTADOS Y DISCUSION La degradación del Poliuretano puede depender de varios factores intrínsecos al polímero, así como de las características bioquímicas del hongo a emplear por lo que fue importante evaluar de forma paralela dos fuentes de carbono y nitrógeno como nutrimentos coadyuvantes para la biodegradación fúngica de poliuretano. El proceso de biodegradación fue monitoreado mediante la medición de las actividades enzimáticas, mientras que la degradación del polímero se determinó mediante análisis de TGA y de FTIR. Análisis bioquímico Debido a la naturaleza fenólica del poliuretano usado, se determinó el contenido de fenol en el medio como indicador de la degradación de PU a los 30 dias de incubación. En los experimentos realizados con las tres cepas y a diversos tratamientos con nutrimentos adicionales, no se encontraron diferencias significativas (Figura 2). Es importante mencionar que existe la posibilidad de que cierta cantidad de fenol liberado al medio de cultivo haya sido usado por los microorganismos como fuente de nutrimentos o energía,2the enzyme is heat labile and not inhibited by phenylmethylsulfonylfluoride (PMSF lo cual se evidencia en el hecho de que los tratamientos que sólo tienen PU como fuente de nutrimentos mostraron menores cantidades de fenol liberado al medio que el PU sin tratar. Esta diferencia podría deberse al uso de fenoles liberados al sobrenadante como nutrimentos por parte de las cepas. Por lo que son necesarias pruebas complementarias de toxicidad para confirmar la inocuidad de los productos de degradación del PU hacia los hongos y otros organismos, y con esto desarrollar alguna estrategia de tratamiento biológico.
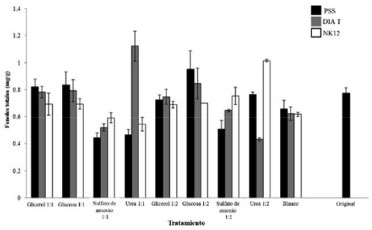 Figura 2. Fenoles liberados al medio de cultivo
Al evaluar las actividades enzimáticas, la actividad ureasa estuvo presente en todas las muestras, viéndose particularmente incrementada en el caso del tratamiento con sulfato de amonio en relación 1:2 (Figura 3). En el caso de las actividades esterasa y ureasa fueron detectadas en el tratamiento con poliuretano como única fuente de nutrimentos para los hongos, por lo que la producción de biomasa puede deberse a otra actividad enzimática o la actividad era muy pequeña como para ser detectada mediante la técnica empleada. No se detectaron actividades lacasa o proteasa en ninguno de los tratamientos. Otra posible explicación es que estos análisis fueron realizados a los 30 días de cultivo, así que es posible que las actividades enzimáticas estuvieran presentes antes en el cultivo. Estudios previos por otros autores, indican que la actividad ureasa es mayor antes de la liberación de los fenoles y que cuando estos eran detectados, la actividad disminuía,27 probablemente debido a que primero se producían las enzimas para actuar sobre el polímero y luego podrían ser degradadas. Esto también se observó en un estudio previo realizado por este grupo de trabajo.20
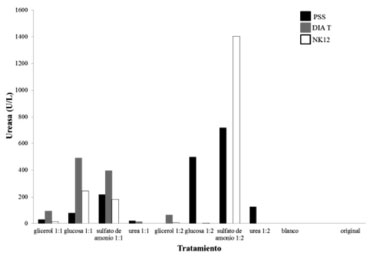 Figura 3. Actividad ureasa
No se detectó actividad esterasa cuando el poliuretano fue la única fuente de nutrimentos, detectándose sólo en el caso de emplear como nutrimento adicional sulfato de amonio en relación 1:2 con el poliuretano, seguido por el tratamiento con urea en la misma relación. No se encontró actividad importante en los otros tratamientos. La presencia y actividad de estas enzimas puede ser explicada porque algunas enzimas, a pesar de su especificidad, tienen cierta flexibilidad y son capaces de actuar sobre sustratos en los que normalmente no actúan. Es importante mencionar este comportamiento de inespecificidad enzimática porque cuando se trata de nuevos materiales en la naturaleza, los microorganismos deben adaptar sus mecanismos enzimáticos más que producir nuevas enzimas específicas.4,10 Análisis termogravimétricos (TGA) y de infrarrojo (FTIR) Para corroborar la degradación del poliuretano se llevaron a cabo análisis térmicos de TGA y espectroscópico de FTIR para identificar cambios estructurales del polímero. Los resultados de TGA (Figura 4) muestran la estabilidad térmica PU sin ningun tipo de tratamiento ("Sin tratar" en la figura) el PU Blanco ("Blanco" en la figura), y los tres tratamientos con las cepas fúngicas.
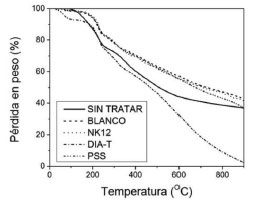 Figura 4. Análisis termogravimétricos
La estabilidad del PU frente a la degradación térmica depende de la relación que haya entre las pártes rígidas y flexibles de su molécula.28 El Pu sin tratar se caracteriza por presentar tres pérdidas en peso. La primera pérdida en peso, por debajo de los 100 °C se relaciona con la pérdida del agua contenida dentro del polímero y otros compuestos volátiles de bajo peso molecular empleados durante la síntesis del polímero. La segunda perdida en peso, más gradual, a los 250 °C se relaciona con la descomposición térmica de las cadenas poliméricas y después de 320 °C y hasta casi 400 °C se descompone el segmento suave y luego el rígido del polímero28 En el caso de las muestras de PU sometidas a la acción del hongo Trichoderma DIA-T se observa un comportamiento similar, una primera degradación entre los 100-300 °C relacionada con los compuestos volátiles, tales como aditivos usados en la síntesis del poliuretano o componentes como biomasa, seguida de una segunda degradación entre 300-400 °C, correspondiente a la degradación de enlaces uretano y a 400-500 °C para enlaces éster.18 El tratamiento con glucosa mostró un descenso apreciable en la temperatura a la cual el poliuretano se degrada desde el principio del análisis. Lo que probablemente indica que la pérdida de peso sea debida a modificaciones en la estructura del poliuretano por la acción enzimática de Trichoderma, especialmente sobre los enlaces uretano, que serían los de más fácil acceso, y se relaciona con la diferencia en la pérdida de peso en esa zona (300-400 °C) la cual, inicia antes en referencia al poliuretano sin tratar, lo que podría indicar modificaciones en la cristalinidad del polímero. La pérdida de peso del poliuretano tratado con microorganismo, comparado con el poliuretano sin tratar fue de 40%. Las curvas de TGA presentan pérdidas en peso relacionadas con reacciones químicas y transformaciones físicas (Figura 6). El termograma del PU sin tratar muestra una curva típica de una reacción de descomposición térmica con productos de reacción volátiles. De acuerdo a la gráfica obtenida de las derivadas en peso se pueden observar dos fases de degradación térmica. La primera fase, a los 200 grados, donde la muestra pierde cerca del 20% de su masa y una segunda fase a 360 grados Celsius donde pierde otro 10 % de su masa, quedando un residuo del 37.60%, por lo que la muestra no llega a ser degradada térmicamente en su totalidad. Lo que indica una descomposición multietapa, con productos intermedios relativamente estables.
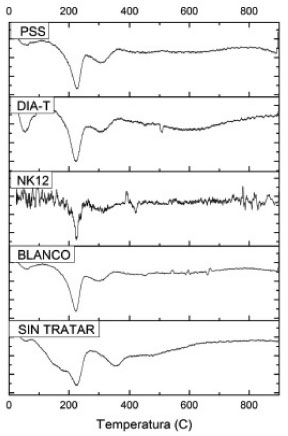 Figura 5. Derivadas en peso del PU tratado con hongos y sin tratar
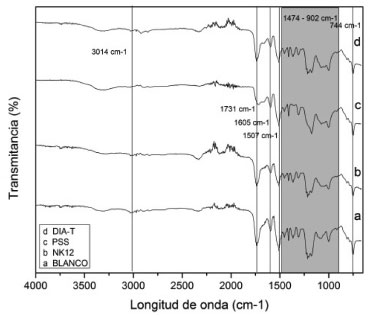 Figura 6. FTIR del PU tratado con hongos y sin tratar. a) Blanco; b Paece lomyces NK12 con glucosa); c)Aspergillus PSS con glicerol; d) Trichoderma DIA-T con glucosa
En el caso de las muestras tratadas por hongos así como en la muestra Blanco, también se logra observar una descomposición multifase. Diferenciándose de la curva del PU sin tratar, por la presencia de una pequeña pérdida en peso inicial por debajo de los 100 grados la cual podría asignarse a la perdida de agua absorbida por el material o residuos de biomasa. Además, como logra apreciarse en la gráfica de las derivadas en peso, ocurre un desplazamiento de la señal de la primera fase de descomposición del polímero hacia mayores temperaturas (230 grados) así como la presencia de nuevas señales probablemente relacionadas con subproductos de la biodegradación, lo que le da una imagen de ruido a la gráfica. La muestra blanco, NK12 y la PSS muestran un residuo no degradable entre 38 - 44%, muy similar al obtenido por el PU sin tratar. La diferencia más notable entre los tratamientos lo tendría aquel llevado a cabo por DIA-T, donde la descomposición térmica de la muestra se lleva casi a la totalidad. Esta diferencia en los porcentajes en el residuo no degradable entre las muestras puede ser atribuida a la presencia de un mayor grado de entrecruzamiento (mayor porcentaje de residuo) o a la presencia de diversos grupos funcionales dentro de la cadena polimérica (menor porcentaje de residuo). La estructura del PU tratado y sin tratar se analizó mediante FTIR (Figura 6). El PU se caracteriza por presentar señales asociadas con la ʋ(N-H) a 3320 cm-1 (1), la señal correspondiente a la vibración del enlace ester ʋ(C=O) a 1730 cm-1 (2), la vibración de los grupos uretanos ʋ(C=O) a 1620 cm-1 (3), y un doblete ubicado a 1513 cm-1 y a 1234 cm-1 (4 y 5) relacionados con δ(N-H) + ʋ(C-N).27,29 Al comparar este espectro con los PU tratados se puede observar ligeros desplazamientos de las señales y disminuciones en su intensidad. El grupo carbonilo de los uretanos se relaciona con una señal intensa ubicada alrededor de los 1700 cm-1. En las muestras evaluadas, esta señal es muy pequeña en comparación a las señales relacionadas con la cadena alifática del polímero (estiramientos C-H y flexiones C-H a 3000 cm-1). La presencia de señales a 1172 y 988 cm-1 se relacionan fuertemente con la presencia de éteres, ésteres y carbonatos, las cuales se relacionan con la señal a 1731 cm-1 (grupo carbonilo) presente en todas las muestras tratadas. Estos datos podrían indicar un nivel de rompimiento de la molécula polimérica y la posible formación de varios subproductos. Los resultados nos podrían indicar la ruptura parcial del enlace uretano debido a actividad ureasa previamente detectada, siendo estos cambios más pronunciados en el tratamiento con Trichoderma DIA-T y con Paecelomyces NK12, esto por la aparición de un "hombro" en la región de 1680 cm-1 que se ha relacionado con la degradación del enlace uretano.30 Así mismo, según Tavares et al,28 los enlaces N-H se encuentran en la región de 3300 cm-1 aproximadamente, y en la Figura 5 se puede observar que el ensachamiento de la dicha banda es menor para el caso del PU tratado con Trichoderma en presencia de glucosa, por lo que es posible suponer que el hongo utiliza el nitrógeno de la molécula del plástico para suplir sus necesidades de este elemento. Esto dejaría libres los grupos carbonilo del enlace uretano aumentando la señal en 1731 cm-1 como se puede ver en la figura.31 Algunos reportes indican que el poliuretano de tipo poliéster es susceptible a la biodegradación en los enlaces éster y que el de tipo poliéter es degradado química o físicamente debido a sus enlaces más estables,32 así que si la biodegradación de este tipo de poliuretano rígido ocurre, está podría iniciarse en los enlaces uretano, como algunos reportes4,33 4-dicarbamic acid dibutyl ester (TDCB lo indican.
CONCLUSIONES El presente estudio evaluó la capacidad de cepas fúngicas para usar PU como fuente de nutrimentos. A través de esto fue posible identificar que cuando se añade glucosa como fuente extra de carbono, la cepa Trichoderma sp DIA-T es capaz de modificar la molécula de poliuretano, como se demostró en los espectros de FTIR y análisis de TGA. Se propone que una enzima ureasa producida por este hongo es la responsable del inicio de la hidrólisis de este poliuretano, sin embargo, se necesitan más estudios para establecer el momento en el cual el hongo produce esta enzima.
AGRADECIMENTOS Los autores agradecen a Nemak S.A. de C.V. y a CONACYT por becas para alumnos. También, se agradece especialmente al Dr. A. Velasco y al Ing. G. García por las facilidades en la realización de este trabajo.
REFERENCIAS 1. Bhardwaj, H.; Gupta, R.; Tiwari, A.; J. Polym. Environ. 2013, 21, 575. 2. Rowe, L.; Howard, T.; Int. Biodeterior. Biodegrad. 2002, 50, 33. 3. Nomura, N.; Shigeno-Akutsu, Y.; Nakajima-Kambe, T.; Nakahara, T.; J. Ferment. Bioeng. 1998, 86, 339. 4. Akutsu-Shigeno, Y.; Adachi, Y.; Yamada, C.; Toyoshima, K.; Nomura, N.; Uchiyama H.; Nakajima-Kambe, T.; Appl. Microbiol. Biotechnol. 2006, 70, 422. 5. Oceguera-Cervantes, A.; Carrillo-García, A.; López, N.; Bolaños-Núñez, S.; Cruz-Gómez, J.; Wacher, C.; Loza-Tavera, H.; Appl. Environ. Microbiol. 2007, 73, 6214. 6. Barratt, R.; Ennos, R.; Greenhalgh, M.; Robson, D.; Handley, S.; J. Appl. Microbiol. 2003, 95, 78. 7. Cosgrove, L.; McGeechan, L.; Robson, D.; Handley, S.; Appl. Environ. Microbiol. 2007, 73, 5817. 8. Ruiz, C.; Main, T.; Hilliard, P.; Howard, T.; Int. Biodeterior. Biodegrad. 1999, 43, 43 9. Urgun-Demirtas, M.; Singh, D.; Pagilla, K.; Polym. Degrad. Stab. 2007, 92, 1599. 10. Akutsu, Y.; Nakajima-Kambe, T.; Nomura, N.; Nakahara, T.; Appl. Environ. Microbiol. 1998, 64, 62. 11. Howard, T.; Hilliard, P.; Int. Biodeterior. Biodegrad. 1999, 43, 23. 12. Allen, B.; Hilliard, P.; Howard, T.; Int. Biodeterior. Biodegrad. 1999, 43, 37. 13. Vega, E.; Main, T.; Howard, G.; Int. Biodeterior. Biodegradation. 1999, 43, 49. 14. Nakajima-Kambe, T.; Onuma, F.; Kimpara, N.; Nakahara, T.; FEMS Microbiol. Lett. 1995, 129, 39. 15. Amaral, S.; Sepulveda, M.; Cateto, A.; Fernandes, P.; Rodrigues E.; Belgacem, M.; Barreiro, F.; Polym. Degrad. Stab. 2012, 97, 2069. 16. Mathur, G.; Prasad, R.; Appl. Biochem. Biotechnol. 2012, 167, 1595. 17. Ibrahim, N.; Maraqa, A.; Hameed, M.; Saadoun, M.; Maswadeh, M.; Nakajima-Kambe, T.; Adv. Environ. Biol. 2009, 3, 162. 18. Matsumiya, Y.; Murata, N.; Tanabe, E.; Kubota, K.; Kubo, M.; J. Appl. Microbiol. 2010, 108, 1946. 19. Khan, S.; Nadir, S.; Ullah, Z.; Ali, A.; Hasan, F.; Environ Pollut. 2017, 225, 469. 20. Loredo-Treviño, A.; García, G.; Velasco-Téllez, A.; Rodríguez-Herrera, R.; Aguilar, N.; Adv. Biosci. Biotechnol. 2011, 2, 52. 21. Bradford, M.; Anal. Biochem. 1976, 72, 248. 22. Makkar, S.; Blümmel, M.; Borowy, K.; Becker, K.; J. Sci. Food Agric. 1993, 61, 161. 23. Witte, P.; Medina-Escobar, N.; Anal. Biochem. 2001, 290, 102. 24. Coca, J.; Dustet, C.; Biotecnol. Apl. 2006, 23, 224. 25. Membrillo, I.; Sánchez, C.; Meneses, M.; Favela, E.; Loera, O.; Bioresour. Technol. 2008, 99, 7842. 26. Blix, G.; Acta Chem. Scand. 1948, 2, 467. 27. Spontón, M.; Casis, N.; Mazo, P.; Raud, B.; Simonetta, A.; Ríos, L.; Estenoz, D.; Int. Biodeterior. Biodegrad. 2013, 85, 82. 28. Tavares, B.; Boas, V.; Schleder, R; Nacas, M.; Rosa, S.; Santos, J.; eXPRESS Polym. Lett. 2016, 927, 940. 29. Hatchett, W.; Kodippili, G.; Kinyanjui, M.; Benincasa, F.; Sapochak, L. Polym. Degrad. Stab. 2005, 87, 555. 30. Oprea, S.; Oprea, V.; e-Polymers 2016, 277, 286. 31. Das, S.; Pandey, P.; Mohanty, S.; Nayak K.; Int Biodeterior Biodegradation. 2017, 117, 278. 32. Cregut, M.; Bedas, M.; Durand, J.; Thouand, G.; Biotechnol. Adv. 2013, 31, 1634. 33. Nakajima-Kambe, T.; Onuma, F.; Akutsu, Y.; Nakahara, T.; J. Ferment. Bioeng. 1997, 83, 456. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access





