Revisão
|
|
| Metabolômica: definições, estado-da-arte e aplicações representativas Metabolomics: definitions, state-of-the-art and representative applications |
|
Gisele A. B. CanutoI,#; José Luiz da CostaII; Pedro L. R. da CruzI; Ana Rosa L. de SouzaI; Andréa T. FaccioI; Aline KlassenIII; Karina T. RodriguesI; Marina F. M. TavaresI,*
IDepartamento de Química Fundamental, Instituto de Química, Universidade de São Paulo, 05508-000 São Paulo - SP, Brasil Recebido em 12/06/2017 *e-mail: mfmtavar@iq.usp.br Metabolomics is an emerging and promising omics approach used to understand biological mechanisms. By untargeted and targeted metabolomics analyses, metabolites are determined in biological samples (fluids, cells, tissues, etc.) by comparison of control groups with altered groups, undergoing different therapies, submitted to differing stress levels, dietary modulation, or promoted by a disease, or specific condition, etc., using sophisticated analytical techniques, and advanced data treatment and statistical analyses. In this review, the concepts involved in metabolomics studies were presented, describing in details all steps involved in the metabolomics workflow, for untargeted and targeted strategies. Finally, the potential of metabolomics is illustrated by applications in representative areas: clinical, environmental, food and nutrition, forensic toxicology, microbiology, parasitology, plants, and sports. Relevant reviews were compiled to characterize each of these areas, and a corresponding application of untargeted and targeted metabolomics were described. INTRODUÇÃO As ciências ômicas buscam o entendimento do funcionamento celular dos organismos e suas alterações biológicas. Fazem parte deste conjunto de ciências, a genômica (estudo da alteração dos genes), a transcriptômica (estudo das alterações dos transcritos), a proteômica (estudo das alterações das proteínas), e a metabolômica (estudo das alterações dos metabólitos). Metabólitos são produtos intermediários ou finais do metabolismo em uma amostra biológica.1 O conjunto de todos os metabólitos de baixa massa molecular (até 1500 Da), presentes ou alterados em um sistema biológico, é chamado de metaboloma (do inglês, metabolome).2 A pesquisa relacionada a metabólitos vem sendo desenvolvida há décadas, mas em 1999, Nicholson et al. definiram a metabonômica (do inglês, metabonomics), como sendo a medida quantitativa da resposta metabólica de um sistema biológico após estímulos fisiopatológicos ou modificações genéticas.3 Já o termo metabolômica (do inglês, metabolomics) foi introduzido em 2001, por Oliver Fiehn, como sendo a análise abrangente e quantitativa do metaboloma de um sistema biológico.4 Além disso, a literatura nos apresenta outras denominações envolvendo esse campo da ciência, tais como: perfil metabólico (do inglês, metabolic profiling), descrito como sendo a análise de metabólitos previamente selecionados de rotas bioquímicas específicas,5 a impressão digital metabólica (do inglês, metabolic fingerprinting), que é definida como sendo "uma classificação de amostras de acordo com sua origem ou sua relevância biológica",3 e, por último, análise footprinting (ainda sem tradução para o português), para se referir aos metabólitos excretados por uma célula em condições controladas.6 Observa-se, portanto, uma divergência em relação às terminologias aplicadas às análises metabolômicas/metabonômicas, mas o que estas definições possuem em comum é que a resposta medida a nível molecular é obtida a partir de uma visão integrada da bioquímica em organismos complexos.7 Assim sendo, será adotado neste trabalho de revisão duas abordagens para classificar as análises metabolômicas: metabolômica alvo (do inglês, targeted metabolomics), definida como a análise quantitativa de um ou mais metabólitos pré-selecionados de determinada classe química, ou que estejam associados a rotas metabólicas específicas, e metabolômica global (do inglês, untargeted metabolomics), que está baseada na análise qualitativa do maior número de metabólitos possível, pertencentes a diversas classes químicas, contidas no sistema biológico em estudo. Cabe ressaltar que essas abordagens sempre se referem a medidas comparativas entre grupos de amostras. A comparação do metaboloma de um grupo sujeito a alterações ambientais ou genéticas, alimentares, de tratamento terapêutico, etc. (grupo teste), com um grupo de indivíduos não alterados (grupo controle) pode fornecer informações importantes no que diz respeito ao entendimento do fenótipo de um organismo, desempenhando um papel fundamental na biologia de sistemas.8 A metabolômica vem sendo aplicada em diferentes áreas do conhecimento, como análises clínicas, alimentos e nutrição, esportes, ambiental, toxicologia forense, ou análise de organismos patológicos (parasitas, bactérias, fungos), entre outras. Uma revisão detalhada de todas as definições e o fluxograma de trabalho das análises metabolômicas global e alvo foram apresentados em um capítulo de livro, recentemente publicado pelo grupo de pesquisa.9 O intuito deste artigo de revisão é apresentar em linhas gerais o fluxograma de trabalho da metabolômica, com ênfase em algumas particularidades e, por fim, destacar artigos de revisão e aplicações em análises global e alvo das grandes áreas estudadas sob o ponto de vista da metabolômica. Fluxograma de Trabalho Para se desenvolver estudos metabolômicos, global e alvo, recomenda-se seguir uma série de etapas, que estão apresentadas resumidamente na Figura 1, e que serão discutidas com mais detalhes nas próximas seções.
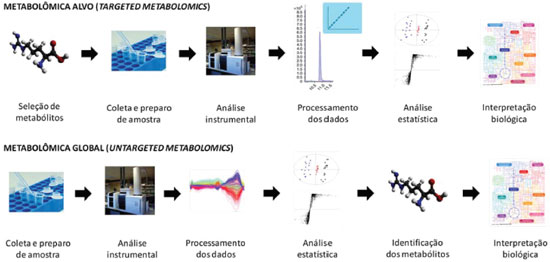 Figura 1. Esquema resumido do fluxograma de trabalho envolvido na análise metabolômica após definição do problema biológico
Com o aumento dos estudos utilizando abordagens metabolômicas, a Sociedade de Metabolômica (do inglês, Metabolomics Society) criou em 2005, o Metabolomics Standards Initiative (MSI) (http://www.metabolomics-msi.org/), que define uma padronização no procedimento adotado nos estudos e publicações de trabalhos em metabolômica, com o objetivo de fornecer uma descrição clara do sistema biológico estudado e todos os componentes do estudo, bem como permitir que os dados sejam efetivamente aplicados, compartilhados e reutilizados. Para desenvolver um estudo metabolômico, um problema biológico a ser estudado deve ser previamente definido, através da elaboração de uma ou mais perguntas a serem elucidadas ao final do estudo. Outro ponto importante é o tipo de abordagem metabolômica que será empregada (global ou alvo), pois a partir dessa escolha é que se definem as etapas do planejamento experimental e análise, uma vez que estas decisões estão inter-relacionadas, e a condução de todo o experimento metabolômico irá afetar diretamente o resultado final.10,11 O planejamento experimental, segunda etapa do fluxograma de trabalho, envolve a escolha do tipo de amostra (tipo de fluido biológico, células, tecido, etc.), a definição do número de amostras que serão avaliadas em cada grupo estudado, e como essas amostras serão coletadas e armazenadas, antes de nova manipulação. Um passo importante, geralmente realizado durante a coleta das amostras, é chamado de quenching metabólico, que se refere à interrupção imediata da atividade enzimática, através da adição de solventes orgânicos resfriados, ou do congelamento imediato das amostras com uso de gelo seco ou nitrogênio líquido. As amostras coletadas devem ser devidamente armazenadas, para evitar degradação ou ativação de enzimas; isso pode ser alcançado estocando-se as amostras a baixas temperaturas (-80 ºC é usualmente preferida).12 O preparo de amostra para análise metabolômica, terceira etapa do fluxograma de trabalho, é uma das etapas mais importantes e que demanda maior tempo de trabalho. Isso se deve à complexidade das amostras biológicas, sua heterogeneidade e grande faixa de concentração dos metabólitos presentes.12 Sua execução depende da abordagem metabolômica escolhida, tipo de amostra e técnicas de análise que serão de interesse no estudo. Para metabolômica global, um preparo de amostras mínimo e não-seletivo é, em geral, preferido. Para amostras líquidas, precipitação de proteínas e remoção de sais através da adição de solventes orgânicos são comumente empregadas. Adicionalmente, uma filtração ou diluição também são executadas. Por outro lado, a extração de amostras semi-sólidas ou sólidas, como células e tecidos, devido a sua maior rigidez, necessitam de aparatos mecânicos, como moinhos, ultrassom e misturadores, em conjunto com solventes orgânicos, para romper a parede celular ou o tecido e extrair os metabólitos de forma eficiente.12 Nas análises alvo, o procedimento é seletivo a um composto ou classe de compostos, envolvendo extrações do tipo líquido-líquido (LLE, do inglês, liquid-liquid extraction), sólido-líquido (SLE, do inglês, solid-liquid extraction), ou em fase sólida (SPE, do inglês, solid-phase extraction), objetivando a concentração dos analitos, remoção de interferentes e seleção das espécies de interesse.13 Como mencionado anteriormente, o preparo de amostra deve ser compatível com a técnica de análise empregada. Para tanto, quando a eletroforese capilar é aplicada, por exemplo, as amostras preferencialmente devem estar em fase aquosa, sendo necessária a evaporação completa dos solventes orgânicos e posterior ressuspensão do resíduo em água.14 Para análises por cromatografia gasosa as reações de derivatização, que convertem os analitos em espécies voláteis nas temperaturas de trabalho, são frequentemente empregadas.15 Por fim, experimentos conduzidos por ressonância magnética nuclear necessitam de diluição dos extratos em solventes deuterados. Sequencialmente ao preparo de amostras, amostras controle de qualidade (QC, do inglês, quality control), que são a mistura de volumes iguais de todas as amostras envolvidas no estudo (todos os grupos avaliados), devem ser preparadas. O objetivo dos QCs é avaliar o desempenho e a estabilidade instrumental durante a aquisição de dados, uma vez que a amostra QC é avaliada em intervalos pré-estabelecidos, sendo sua leitura intercalada à das amostras, durante toda a sequência analítica.16,17 Cobrir todo o metaboloma de um organismo biológico não é uma tarefa fácil, uma vez que existe uma grande diversidade química de metabólitos, com concentrações variadas. Não é possível, portanto, encontrar uma única técnica analítica capaz de fazê-lo.8,18 Assim, a aquisição dos dados dos estudos metabolômicos, vem sendo realizada com o uso de multiplataformas de análise, que proporcionam maior cobertura em termos de substâncias químicas detectadas e, consequentemente, proporcionam um maior entendimento biológico do organismo estudado.18,19 Ferramentas poderosas de análise como a ressonância magnética nuclear (NMR, do inglês, nuclear magnetic ressonance)20,21 e a espectrometria de massas (MS, do inglês, mass spectrometry),22 que fornecem informações estruturais de diversas classes químicas, são as técnicas analíticas mais empregadas nos estudos metabolômicos. Menos frequentemente, podem ser encontradas aplicações utilizando infravermelho por transformada de Fourier (FT-IR, do inglês, Fourier transform infrared spectroscopy), que demonstra grande potencial por ser uma técnica de elucidação estrutural de baixo custo, simples e não destrutiva.23 A NMR é uma técnica simples, robusta e abrangente, que requer pouca ou nenhuma manipulação de amostra, podendo ser analisadas amostras biológicas intactas (sólidos e semi-sólidos).24,25 Tem a vantagem de precisar de pouca quantidade de amostra e não destruir a mesma após análise, apesar de contaminá-la com solventes deuterados. Em contrapartida, apresenta baixa sensibilidade e seletividade, apresentando regiões espectrais com sobreposição de sinais, comprometendo a interpretação dos resultados; além disso, são necessários equipamentos de alta resolução.7,19,26 A MS é a técnica de análise mais usada na metabolômica, uma vez que proporciona análises rápidas, com alta sensibilidade e seletividade.8 Pode ser usada através da infusão direta dos extratos (DIMS, do inglês, direct infusion mass spectrometry), que muitas vezes dificulta a análise por problemas de supressão de ionização, ou ainda pode ser utilizada através da ionização e dessorção a laser assistida por matriz (MALDI-MS, do inglês, matrix-assisted laser desorption ionization mass spectrometry), esta última bastante aplicada especialmente na análise de compartimentos celulares.27,28 Os constantes problemas de supressão de ionização dos sinais obtidos nas análises por DIMS, decorrente de matrizes complexas, são contornados através do acoplamento desta com técnicas analíticas de separação, como a cromatografia gasosa (GC, do inglês, gas chromatography), a cromatografia líquida de alta eficiência (LC, do inglês, liquid chromatography) e a eletroforese capilar (CE, do inglês, capillary electrophoresis).8 Além disso, a informação de tempo de retenção, ou migração do metabólito previamente separado, auxilia na comprovação de identificação do metabólito, com uso de padrões analíticos autênticos. A GC-MS é uma das técnicas de análise mais empregadas em estudos metabolômicos,29-32 e apresenta alta robustez e repetibilidade. A análise de compostos voláteis por headspace vem sendo também empregada para acessar a porção volátil do metaboloma.31 Entretanto, grande parte dos metabólitos que compõem o metaboloma requerem derivatização, para que se tornem voláteis a baixas temperaturas.19 A derivatização é um procedimento tedioso, que pode introduzir erros por perdas por volatização durante o processo, e limita o número de amostras a serem trabalhadas em conjunto. Todavia, o uso de GC-MS em metabolômica apresenta uma grande vantagem, resultante da construção de bibliotecas de espectros, conferindo alta credibilidade na identificação dos metabólitos, que combina a informação de tempo de retenção e o padrão de fragmentação obtido, em decorrência da ionização por elétrons. Atualmente, a LC-MS vem sendo cada vez mais aplicada nos estudos envolvendo análises metabolômicas.16,33-36 É uma técnica analítica que apresenta alta robustez, sensibilidade e seletividade, além de ser de fácil operação.37 LC-MS é considerada uma técnica abrangente no que se refere à classe de compostos a serem analisadas, devido a grande variedade de fases estacionárias disponíveis, além dos diferentes modos de separação, como a eluição em fase reversa (fundamentalmente partição), com ou sem pareamento iônico, interação hidrofílica, e troca iônica. A CE-MS vem sendo usada como técnica complementar a LC-MS e GC-MS,38-40 pois promove a separação de compostos iônicos polares. Possui alta resolução e curtos tempos de análise. Além disso, utiliza volumes de injeção da ordem de nL, sendo vantajoso quando se trabalha com fluidos biológicos coletados em pequenas quantidades, como urina de rato e saliva de animais, por exemplo. Contudo, apresenta problemas de repetibilidade e sensibilidade, este último devido à diluição das amostras pelo uso de líquido auxiliar no acoplamento com MS. Novas tecnologias de interfaceamento CE-MS, desenvolvidas sem uso de líquido auxiliar (do inglês, sheathless), estão sendo desenvolvidas, promovendo melhora de até duas ordens de grandeza nos limites de detecção de algumas análises.41 Os dados gerados pelas análises metabolômicas são abundantes e complexos e, por isso, ferramentas adequadas de tratamento de dados devem ser empregadas no processamento destes, para evitar erros e manter a integridade das variações biológicas inspecionadas.42 Para análise metabolômica global, aplicam-se etapas de alinhamento, agrupamento, correção do tempo de retenção e de linha de base, deconvolução espectral, e normalização. Existem atualmente, diversos softwares livres e licenciados para tratamento de dados metabolômicos.43-47 Misra e van der Hooft (2016) descrevem uma série desses softwares de tratamento de dados, e bases de dados para correlações de massas e rotas metabólicas.48 Um trabalho interessante de Sugimoto et al. (2012) compara diversos softwares para análise metabolômica de dados obtidos por MS.42 Para análise metabolômica alvo, o resultado da análise se dá em termos da quantificação dos metabólitos pré-selecionados.49,50 Para aumentar a precisão das quantificações, padrões internos e padrões marcados isotopicamente são comumente utilizados.51 A análise estatística é usada para extrair a informação relevante dos dados, através de métodos de análise multivariada e univariada. Nas análises multivariadas, a classificação e discriminação de entidades ou metabólitos responsáveis por diferenciar os grupos de amostras são realizadas através da avaliação do conjunto da matriz de dados extraídos na etapa de trabalho anterior. São aplicados métodos não supervisionados, como análise de componentes principais (PCA, do inglês, principal component analysis), e métodos supervisionados, como análise discriminante por mínimos quadrados parciais (PLS-DA, do inglês, partial least squares discriminant analysis), e projeções ortogonais para estruturas latentes (OPLS-DA, do inglês, orthogonal partial least squares discriminant analysis). Já nas análises univariadas, as variáveis de estudo (entidades ou metabólitos) são avaliadas separadamente e, ao contrário dos métodos multivariados, são desprezadas as relações entre elas. Testes estatísticos como ANOVA, teste t de Student, ou de Mann-Whitney U, são frequentemente utilizados.42 A identificação dos metabólitos, apenas para estudos globais, é realizada utilizando-se bibliotecas de espectros construídas no próprio laboratório de análise, ou pelo uso de bibliotecas comerciais, como a NIST (do inglês, National Institute of Standards and Technology). Para determinação putativa dos metabólitos, buscas em bases de dados públicas online, como HMDB (do inglês, Human Metabolome Database),52 Metlin,53 MassBank,54 etc. são frequentemente realizadas. A confirmação da identificação desses metabólitos putativos pode ser posteriormente realizada através de análise metabolômica em fluxo, utilizando compostos marcados isotopicamente, ou ainda análise NMR-2D ou MS/MS para elucidação estrutural, além de adição por spiking de padrões analíticos às amostras. A interpretação biológica visa buscar as respostas para as questões levantadas no início do fluxograma de trabalho, ou possibilita gerar uma nova hipótese. Essa interpretação é realizada através da correlação dos metabólitos alterados com rotas metabólicas,55 utilizando-se bibliotecas como KEGG (do inglês, Kyoto Encyclopedia of Genes and Genomes),56 MetaCyc,57 MetaboLights,58 entre outras. Para confirmar a interpretação dos efeitos bioquímicos, uma validação biológica é recomendada,59,60 na qual novos experimentos são elaborados com novas amostras e estas são submetidas à análise metabolômica. Adicionalmente, metabolômica alvo, com a quantificação dos metabólitos discriminantes avaliados pela abordagem global, também pode ser realizada.
ÁREAS DE APLICAÇÕES DA METABOLÔMICA A seguir são apresentadas algumas compilações de artigos de revisão das maiores áreas de aplicação em metabolômica, com breve descrição de dois trabalhos (global e alvo), desenvolvidos por grupos de pesquisa de referência nas áreas em destaque, a fim de se conhecer quais tipos de estudos estão sendo executados na atualidade. Ambiental Estudos nos quais a metabolômica é aplicada a organismos obtidos diretamente de seu ambiente natural, ou criados em laboratório e submetidos a experimentos desenhados para reproduzir situações de ambientes naturais, são caracterizados como metabolômica ambiental.61 Esta definição foi apresentada em 2007, por cientistas da MSI, que recomendaram maneiras para reportar metadados de experimentos na área.61 A metabolômica ambiental envolve o estudo das repostas metabólicas de organismos a fatores bióticos (competição, mutualismo, relação parasita-hospedeiro, planta-herbívoro, entre outros), e abióticos (temperatura, umidade, disponibilidade de nutrientes, exposição a poluentes, etc.).62-67 A aplicação da metabolômica na área ambiental é muito ampla e envolve o estudo de organismos aquáticos,67-70 de plantas,71 e como estes respondem ao estresse oxidativo,72 estresse abiótico,73-75 fatores bióticos,76 ou fatores bióticos e abióticos simultaneamente.77 Também são encontrados trabalhos de exposição de organismos a poluentes,78 estudos de toxicidade aplicados à agricultura,79,80 modo de ação de pesticidas,79,81 toxicidade e função de metais em sistemas biológicos (também chamado de metallomics),82-85 biodisponibilidade de contaminantes no solo,78 biorremediação,18,86 entre outros. Estudos apontam a presença de fármacos em diferentes concentrações em águas de rios, em estações de tratamento de águas, de estuários, entre outros. Porém, não está bem estabelecido na literatura se a presença de fármacos em baixa concentração na natureza pode representar um risco para organismos microbianos. Nesse âmbito, Goodacre e colaboradores realizaram um estudo exploratório em culturas de Pseudomonas putida para avaliar os efeitos da exposição de seis fármacos no metabolismo desse organismo, empregando FT-IR e GC-MS. Alterações significativas no metabolismo de P. putida foram observadas para os medicamentos propranolol, diclofenaco, ibuprofeno e acetaminofeno. Como o propranolol apresentou maior quantidade de metabólitos alterados (43 no total), uma análise de correlação foi realizada, que indicou que as alterações desses metabólitos estão relacionadas à energia (Figura 2A). Os resultados globais desse estudo sugerem que bombas de efluxo dependentes de energia são empregadas na remoção de compostos tóxicos das células. Além disso, os lipídios da membrana estão alterados, numa tentativa de manter a integridade celular. A aplicação da metabolômica neste tipo de trabalho promove melhor entendimento dos mecanismos de respostas ao estresse abiótico de importantes microorganismos para o meio ambiente.87
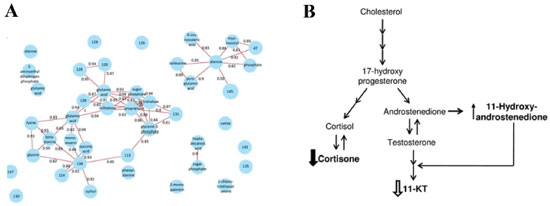 Figura 2. Exemplos de estudo em metabolômica ambiental. A) Análise de correlação de 43 metabólitos significativos de células de Pseudomonas putida expostas a propranolol. Reproduzido da referência 87 com permissão da The Royal Society of Chemistry. B) Alterações metabólicas na esteroidogênese de Rutilus rutilus causadas pelo pesticida fenotrotion. Reproduzido da referência 88 com permissão da ACS
O pesticida fenotrotion é empregado como um inseticida de amplo espectro e possui efeitos muito nocivos para o ambiente aquático. Para compreender a toxicidade desse pesticida a nível molecular, Viant e colaboradores empregaram duas abordagens metabolômicas, global e alvo, para estudar peixes das espécies Rutilus rutilus. A hipótese de que vias metabólicas da acetilcolina e dos esteroides poderiam estar alteradas pela resposta tóxica do organismo à ação do pesticida foi avaliada por metabolômica alvo. Para essa abordagem, empregou-se DIMS, para quantificar acetilcolina e colina em amostras de cérebro, e LC-MS para determinar esteroides nas gônadas masculinas, além de um radioimunoensaio para quantificar 11-ketotestosterona (11-KT) no plasma. A metabolômica global foi realizada em amostras de gônadas masculinas e fígado, empregando-se NMR e DIMS. O principal mecanismo de toxicidade do pesticida foi confirmado pela alteração de acetilcolina no cérebro dos peixes expostos ao pesticida. Porém, esperava-se níveis baixos de acetilcolina, o que sugere que a exposição crônica ao pesticida pode levar a um certo grau de adaptação. Além disso, a análise metabolômica alvo dos esteroides apontou baixos níveis de cortisona e uma tendência de aumento de 11-hidroxiandrostenediona nas gônadas masculinas, além de níveis baixos de 11-KT no plasma dos peixes. Esses resultados indicam uma alteração no metabolismo de esteroides (Figura 2B). A metabolômica global revelou uma alteração no metabolismo da fenilalanina, que ainda não havia sido reportada. Com base nos resultados inesperados em relação a previsões de toxicidade aguda obtidos nesse estudo, os autores sugeriram que a exposição crônica ao pesticida provocou uma adaptação fenotípica nos peixes, o que deve ser levado em conta em estudos similares de exposição crônica.88 Clínica A metabolômica tem proporcionado um aumento na compreensão, em nível molecular, de diversas patologias. Inúmeras são as áreas de aplicações clínicas e o número de publicações relacionado ao tema cresce a cada dia, especialmente no que tange à detecção de biomarcadores,89,90 o desenvolvimento de novos tratamentos,91,92 diagnóstico,59 etc. Neste tópico são destacados alguns trabalhos de revisão de temas relevantes, como por exemplo na área oncológica,93-95 na qual estudos têm demonstrado alterações no metabolismo, fornecendo subsídios para o diagnóstico e monitoramento das doenças, como no caso do câncer de próstata,96 gástrico,97 colorretal98 e de mama.99 Outra doença crônica que vem sendo extensamente estudada é a diabetes tipo 2,100-102 que tem o número de casos aumentado ao longo dos anos, demandando a busca por biomarcadores que possam diagnosticar precocemente esta patologia. A área clínica também tem se beneficiado de investigações focadas em doenças cardiovasculares,103,104 uma das maiores causas de morte no mundo,105 e na identificação de alterações no metabolismo causadas pela doença de Alzheimer.106 Entre outras áreas promissoras destacam-se trabalhos relacionados a tuberculose,107 doenças renais,108,109 esclerose múltipla110 e doença celíaca.111 Com uma abordagem de análise global, amostras bacterianas do intestino e do plasma de pacientes infectados pelo HIV (do inglês, human immunodeficiency virus) que passaram por terapia antirretroviral foram analisadas por Serrano-Villar et al. Já é conhecida a relação entre a infecção por HIV e a composição da microbiota intestinal, porém, a investigação em nível molecular tem sido pouco explorada. Utilizando as técnicas de LC-MS e CE-MS, foram examinados grupos de pacientes infectados pelo HIV, inlcuindo pacientes sem tratamento prévio (VU), respondedor imunológico (IR) e não respondedor imunológico (INR), e um conjunto de indivíduos controle não infectados por HIV. Foram identificados metabólitos discriminantes entre os grupos, como pode ser observado no diagrama de Venn da Figura 3A. Este estudo permitiu mostrar que a infecção pelo HIV pode estar associada a mudanças no conjunto ativo de bactérias intestinais.112
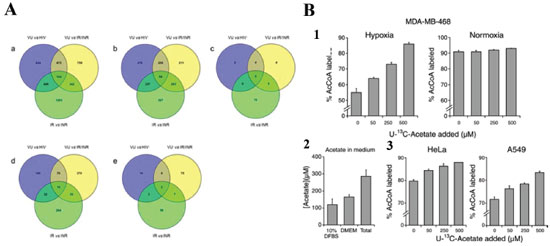 Figura 3. Exemplos de estudo em metabolômica clínica. A) Entidades discriminantes entre os diferentes grupos de indivíduos para estudo da ação de antirretrovirais em pacientes com HIV, em amostras de bactérias intestinais analisadas por (a) LC-MS modo positivo, (b) LC-MS modo negativo, e (c) CE-MS modo positivo; e amostras de plasma, analisadas por (d) LC-MS modo positivo, e (e) LC-MS modo negativo. Reproduzido da referência 112 com permissão da Elsevier. B) Porcentagem aumentada de acetil-CoA marcado com C13 na avaliação da proliferação de células cancerígenas. (1) célula normal em condição de hipóxia e rica em oxigênio (normoxia), com adição em concentrações crescentes de acetato marcado em meio contendo glicose e glutamina marcada; (2) quantificação de acetato em meio de cultura sem soro e em soro dialisado (controle); (3) células cancerígenas em condição de hipóxia, com adição em concentrações crescentes de acetato marcado. Reproduzido da referência 113 com permissão da BioMed Central
Para o entendimento da proliferação de células cancerígenas, Rabinowitz e colaboradores investigaram, por meio de abordagem metabolômica alvo, possíveis fontes da biossíntese de acetil-CoA. Em condições ricas em oxigênio, a acetil-CoA deriva da glicose. Porém, nas células cancerígenas hipóxicas, a contribuição da glicose diminui. Para investigar a origem da biossíntese nestas condições a quantificação de acetil-CoA foi realizada através da análise por MS e traçadores marcados com C-13, em células cancerígenas (HeLA e A549) e saudáveis (MDA MD468). Os resultados mostraram que a fonte de produção de acetil-CoA foi o acetato (Figura 3B) e permitiram identificar a enzima responsável por sua assimilação. Este estudo possibilitou vislumbrar inibidores desta enzima para impedir o crescimento tumoral.113 Esporte Exercícios físicos são capazes de oferecer proteção cardiovascular, revelar disfunções ocultas em órgãos, além de auxiliar no tratamento e prevenção de diversas doenças crônicas, como síndromes metabólicas, diabetes tipo 2 e doenças cardiovasculares. O conhecimento de mudanças metabólicas induzidas pela prática de esportes ou exercícios físicos pode auxiliar no esclarecimento dos fatores que influenciam tais processos.114 A metabolômica se apresenta como uma ferramenta inovadora para elucidar os complexos mecanismos associados à prática de exercícios físicos, relevantes para a saúde humana. Por muito tempo, essa abordagem foi especialmente aplicada à análise de controle do abuso de drogas proibidas no esporte, sendo muito utilizada para testes anti-doping em humanos,115-118 nas quais amostras de urina e plasma de atletas são analisadas; entretanto, um biofluido alternativo, o suor, tem sido proposto como um novo tipo de amostra para realização desses testes.119 Progressos nas técnicas de detecção e métodos de análise têm sido apresentados com o objetivo de se buscar biomarcadores de doping.120,121 Adicionalmente, a metabolômica também é aplicada no controle de abuso de medicamentos em animais, especialmente em cavalos de corrida no hipismo.122,123 Outros tipos de estudos da área reportam a avaliação de performance de exercícios físicos,124 além do entendimento das alterações metabólicas resultantes da realização de atividades físicas e seus impactos na saúde, especialmente na prevenção de doenças.125,126 O efeito da prática de exercícios é tópico de uma série de estudos, como alterações no metabolismo e envelhecimento,127-129 efeito no crescimento infantil130 e ainda, prevenção de doenças em mulheres grávidas e seus fetos.131,132 Bassini e Cameron introduziram, em 2014, o termo sportomics como sendo a "investigação das alterações metabólicas em indivíduos durante a prática de esportes e exercícios físicos", que visa auxiliar no entendimento do metabolismo na prática de esportes e aprimoramento da ciência do esporte, na busca de melhor performance, treinamento e recuperação.133 Apesar de ser uma aplicação relativamente recente, estudos envolvendo metabolômica e exercícios físicos podem ser facilmente encontrados na literatura, como por exemplo o trabalho de Britz-McKibbin e colaboradores, que explora a resposta metabólica adaptativa em plasma de mulheres com sobrepeso/obesas (BMI > 25), submetidas a 6 semanas de exercícios de alta intensidade. Neste trabalho, as análises foram realizadas por CE-MS, com injeção sequencial das amostras em única corrida analítica (Figura 4A). Os resultados revelaram que adaptações à prática do exercício físico estavam relacionadas com o aumento de L-carnitina no plasma, devido à melhora na capacidade oxidativa do músculo, induzida pelo exercício físico, enquanto que o status inferior de tiol plasmático redox circulante estava associado a uma maior capacidade antioxidante intracelular. Adicionalmente, alterações nas concentrações plasmáticas de O-acetil-L-carnitina e hipoxantina após prática de exercício correspondiam a um menor estresse energético e maior capacidade de acetilação para as mulheres treinadas. Os estudos demonstram que a busca por marcadores de capacidade de resposta ao exercício é importante para auxiliar na prevenção de doenças crônicas e na manutenção da saúde.134
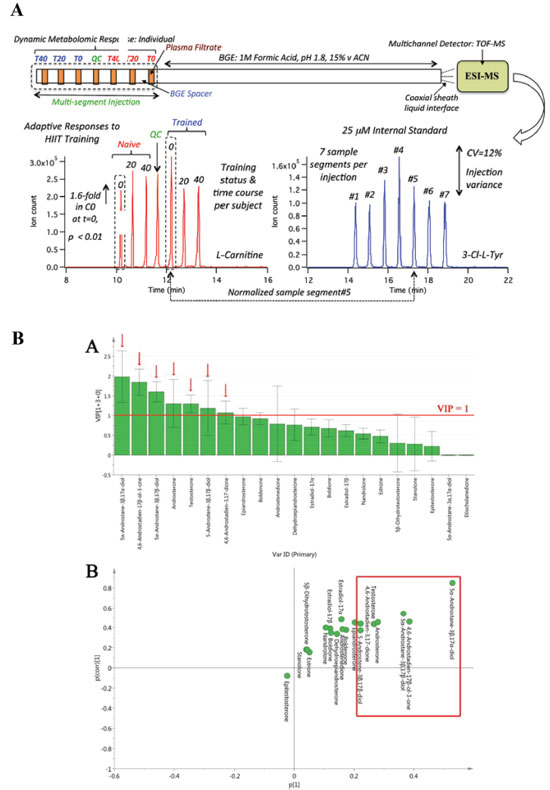 Figura 4. Exemplos de estudo em metabolômica no esporte. A) Apresentação de esquema de injeção sequencial de sete amostras em CE-MS para análise em única medida de separação e respectivos eletroferogramas extraídos de L-cartinina (vermelho) e padrão interno (azul) do estudo metabolômico da análise de plasma de mulheres submetidas a exercícios físicos de alta intensidade. Reproduzido da referência 134 com permissão da John Wiley and Sons. B) Resultados de (A) VIP score e (B) S-plot construído de modelos OPLS-DA, com destaque nos sete esteroides considerados potenciais biomarcadores para análise anti-doping em cavalos de corrida. Reproduzido da referência 135 com permissão da ACS
Outro estudo recente, utilizando abordagem metabolômica alvo foi publicado por Chan et al., cujo objetivo era estabelecer um novo modelo para a detecção de possíveis abusos de esteroides inibidores da aromatase, em cavalos usados no hipismo. Esteroides endógenos livres e conjugados foram quantificados em urina por GC-MS/MS. Através de análise estatística multivariada (Figura 4B), os autores reportaram que 7, dos 21 esteroides avaliados, foram detectados como possíveis biomarcadores para discriminar o grupo que recebeu as doses de esteroides do grupo controle. Adicionalmente, esse estudo revelou que através da abordagem proposta os inibidores 6-OXO (4-androsteno-3,6,17-triona) e ATD (1,4,6-androstatrieno-3,17-diona) podem ser detectados em urina por um tempo mais longo, em comparação ao método convencional de detecção direta já adotado para determinação de doping. Por fim, esses resultados demonstraram que a determinação de biomarcadores por metabolômica é uma ferramenta eficiente e viável para a detecção de abusos de diferentes drogas de mesma classe química, em diversas modalidades do hipismo.135 Foodomics e Nutrição Com o crescente número de trabalhos na área de alimentos sob a perspectiva das ciências ômicas (transcriptômica, proteômica e metabolômica), e com o intuito de relacionar os alimentos e seus componentes alimentares, a dieta e o indivíduo, a saúde e as doenças, um novo conceito foi inserido na comunidade científica, a foodomics, que é definida como uma nova abordagem que relaciona as tecnologias ômicas avançadas com a área de alimentos e nutrição, com o intuito de melhorar o bem-estar, a saúde e segurança dos consumidores, integrando pesquisadores de diversas áreas do conhecimento.136,137 É possível encontrar uma série de trabalhos de revisão na literatura sob perspectiva da metabolômica em ciência de alimentos e nutrição, seja com ênfase nas técnicas de análise empregada e suas diversas aplicações,138-140 ou com descrição em tratamento de dados,141,142 sugerindo o grande potencial desta abordagem na análise de alimentos.143 Sendo o intuito da foodomics integrar todas as grandes áreas emergentes em alimentos,136 diversas aplicações da área vem sendo compiladas, tais como: autenticação de alimentos,144,145 toxinas e segurança de alimentos,146-149 dieta e nutrição,150-152 química verde,153 produtos alimentícios diversos154 e oriundos de plantas,149,155 dentre outras. Dentro da ciência de alimentos, uma atenção mais detalhada é dada à pesquisa em nutrição, visando correlacionar dieta, estilo de vida e saúde, com genes, proteínas e metabólitos. Nesse contexto, surgem novas áreas do conhecimento que foram nomeadas de nutrigenética, nutrigenômica, nutriproteômica e nutrimetabolômica, respectivamente.156 A aplicação dessas novas áreas pode ser comprovada pela extensa compilação de trabalhos encontrados na literatura.157-162 A nutrimetabolômica, que se refere à aplicação da metabolômica na área de nutrição, permite a caracterização de fenótipos individuais, por meio da medida de centenas ou milhares de metabólitos, após a exposição a uma dieta específica, por exemplo. De um modo geral, a nutrimetabolômica está focada na descoberta de biomarcadores por meio da avaliação do efeito de uma dieta,163 estudos de intervenção de dieta em uma rota metabólica,164,165 e estudo da relação dieta-doença157 ou, mais recentemente, na busca de uma dieta personalizada.166,167 Em se tratando do estudo da relação dieta-doença, um trabalho interessante dos benefícios da dieta a base de extrato de alecrim contra câncer de cólon pode ser destacado, no qual foram aplicadas três abordagens ômicas (transcriptômica, proteômica e metabolômica), com mais de uma plataforma de análise para a metabolômica global (CE-MS, RPLC-MS e HILIC-MS). A Figura 5 apresenta um esquema com os resultados dos metabólitos, proteínas e genes alterados nas células de câncer após tratamento com o extrato de alecrim. Neste estudo, mais de 1300 genes, 17 proteínas e 30 metabólitos foram identificados, estes foram correlacionados a diferentes rotas metabólicas, sendo as principais responsáveis pelo desenvolvimento, manutenção e morte celular. Os autores afirmam que ainda há muito para investigar sobre os mecanismos envolvidos para os compostos alterados quando se faz uso do alecrim, entretanto, este trabalho auxilia no melhoramento do conhecimento dos efeitos quimiopreventivos dos polifenóis presentes no alimento, destacando a importância da dieta no combate e prevenção de doenças.168
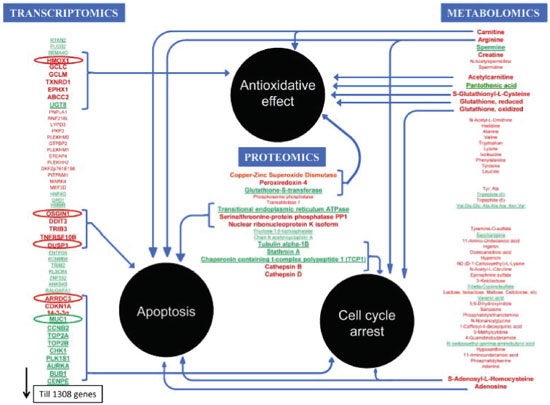 Figura 5. Exemplo de estudo em foodomics. Metabólitos, proteínas e genes envolvidos na ação de extratos de alecrim em células de câncer de cólon. Reproduzido da referência 168 com permissão da Elsevier
Em se tratando de intervenção de dieta, Brennan e colaboradores avaliaram o efeito da ingestão de vitamina D em síndrome metabólica através de análise global e alvo, em amostras de urina e sangue (soro e plasma). A determinação dos metabólitos foi realizada por 1H-NMR e os marcadores bioquímicos foram medidos por kits comerciais. A avaliação dos resultados de metabolômica alvo foi realizada por análise de agrupamento por k-means, baseada em 14 marcadores bioquímicos de síndrome metabólica e na concentração de 25-hidroxivitamina D (25(OH)D). Os resultados mostraram que houve um aumento da 25(OH)D nos indivíduos que receberam a suplementação da vitamina D, indicando menor risco de ocorrência de síndrome metabólica. A análise global dos metabólitos alterados mostrou que lactato, colina e lipoproteínas de baixa densidade (VLDL + LDL) são os metabólitos mais discriminantes entre grupos com e sem suplementação da vitamina D. Esses resultados suportam a hipótese de que o efeito da vitamina D é modulado, em parte, pelo metabolismo de lipídios. Os autores concluem que a vitamina D é um fenótipo responsivo, com grande potencial em estudos nutricionais, e que estudos complementares, incluindo outros fatores genéticos e fenótipos, devem ser executados para a compreensão da capacidade de resposta de dietas específicas.169 Microbiologia A microbiologia é a ciência que estuda os organismos microscópicos, como fungos, bactérias e vírus. Estudos da evolução, fisiologia e bioquímica desses organismos estão intimamente relacionados aos sistemas biológicos e assim, a metabolômica, juntamente com as outras ciências ômicas, tem um papel fundamental na compreensão dos fenômenos biológicos ocorridos na microbiologia.170 A metabolômica tem sido aplicada a organismos microbianos, especialmente estudos relacionados ao metabolismo da microbiota intestinal171-175 e interações organismo-hospedeiro,171,173,176 bem como marcadores de exposição a estresse ou alterações ambientais.177-180 Também são encontrados estudos de metabolismo secundário de microorganismos181 para produção de fármacos, biocombustíveis, agroquímicos etc. O metabolismo de diversos organismos microbianos,170,182 incluindo bactérias Mycobacterium tuberculosis183,184 e Staphylococcus aureus,185 além do conhecimento das novas vias metabólicas de Escherichia coli186 também tem sido revisados. Dentre as aplicações industriais, destacam-se a área de fermentação de alimentos187 e busca por alimentos funcionais, como os probióticos.188,189 Um segmento novo e que vem sendo explorado na indústria e estudado pelas ciências ômicas é o processo de biolixiviação, que consiste na conversão de metais, frutos de rejeitos industriais, catalisada por microorganismos.190 Na área médica o foco é voltado para pesquisas sobre diagnóstico de infecções,179,191 resistência ou desenvolvimento de novos fármacos,192,193 produção de vacinas185,194 e antibióticos195 a partir de microorganismos. Para entendimento do metabolismo e crescimento de bactérias Staphylococcus aureus, Sun et al. utilizaram metabolômica global através de análises por NMR para avaliar o efeito do oxigênio no crescimento bacteriano. Os experimentos foram executados em cultivos de S. aureus sob condições aeróbicas e anaeróbicas. Também foram comparados o crescimento em cultura controle (meio suplementado com glicose). Cerca de 50 compostos foram identificados, a separação entre os grupos de cultura aeróbica e anaeróbica podem ser observadas no modelo PCA da Figura 6A, assim como os metabólitos responsáveis por essa separação. Os metabólitos mais discriminantes entre os perfis foram isobutirato, isovalerato e succinato, em condições aeróbicas, e lactato, etanol e ornitina, em condições anaeróbicas. Os autores observaram que a presença de oxigênio é de fato muito importante para o crescimento dos microorganismos, e que alteram significativamente o metaboloma do S. aureus. Estes resultados podem auxiliar no desenvolvimento de novas estratégias para lidar com contaminações e infecções pelo microorganismo.196
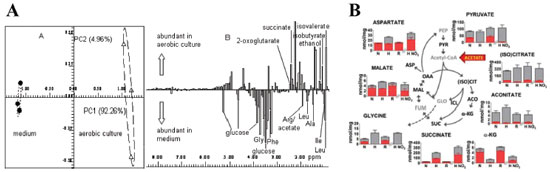 Figura 6. Exemplos de estudo em metabolômica em microbiologia. A) Gráfico de scores e loading de modelo PCA dos metabólitos responsáveis pela discriminação das condições de crescimento de S. aureus, grupos aeróbico e anaeróbico. Reproduzido da referência 196 com permissão da NRC Research Press. B) Remodelamento associado à hipóxia da atividade do ciclo TCA de Mycobacterium tuberculosis. Altura das barras indica a concentração do metabólito e a coloração, a extensão da marcação isotópica proveniente do acetato. Reproduzido da referência 197 com permissão da PNAS
Um estudo metabolômico alvo por LC-MS baseado em metabolômica em fluxo foi aplicado a amostras de Mycobacterium tuberculosis durante hipóxia, para avaliar as adaptações metabólicas, especificamente no ciclo do ácido tricarboxílico (TCA). Diferentes porcentagens de oxigênio foram avaliadas durante a replicação das bactérias. A análise em fluxo, utilizando acetato isotopicamente marcado com C-13, demonstrou diferenças em derivados de ácidos orgânicos, intermediários do TCA, como apresentado na Figura 6B. Os autores observaram que os teores de a-cetoglutarato diminuiu, enquanto que succinato, malato e aspartato aumentaram. Essas alterações indicam uma ativação na enzima isocitrato liase, a qual já se tem relatos de estar relacionada a sobrevivência de microorganismos. Por fim, concluem que as alterações demonstram que a M. tuberculosis se adapta facilmente à hipóxia, através de uma alteração em seu metabolismo bioenergético. Esse mecanismo pode ser facilmente extrapolado para outros microorganismos ou células (tumorais e hospedeiras), auxiliando no entendimento de adaptações em diferentes condições e concentrações de oxigênio.197 Parasitologia Parasitas são organismos que necessitam de um hospedeiro para se manterem vivos e em crescimento. Podem pertencer a três classes: protozoários (seres unicelulares, como Leishmania, Plasmódio, Giardia, etc.), helmintos (tipos de vermes, como Fasciolas, Ascarídeos, Esquistossomos, etc.) e ectoparasitas (parasitas externos, como piolho, carrapato, sanguessugas, etc.). As infecções parasitárias causam uma série de doenças em humanos, especialmente nos que vivem em países em desenvolvimento.198 As ciências ômicas têm se mostrado bastante adequadas para estudos da biologia parasitária; tecnologias de análise avançadas e tratamento de dados são frequentemente apresentados nos estudos metabolômicos de parasitas.199,200 Em 2010, uma edição especial da revista Parasitolgy (Insights into the metabolomes of parasites),201 contendo 14 publicações de especialistas na área, foi dedicada a artigos de revisão em metabolômica parasitológica. Além desta edição especial, frequentemente são encontradas compilações neste tema, dedicadas a aplicações na identificação de novas drogas-alvo,202,203 estudos de mecanismo de ação e resistência de intervenções farmacêuticas,204,205 e desenvolvimento de novas vacinas.206 Além disso, são encontrados trabalhos que revisam o aprimoramento de estratégias de diagnóstico e tratamento de doenças parasitárias,207 o conhecimento da interação hospedeiro-parasita208-210 e determinação de biomarcadores.211-213 Em uma busca rápida nas bases de dados de artigos científicos nos deparamos com centenas de trabalhos de revisões em metabolômica aplicada às doenças causadas por parasitas, como por exemplo, doenças negligenciadas,214 ou especificamente destacando gêneros parasitários, como: Leishmania215,216 e Tripanossoma217 ou ambas,218 Plasmódio,219-221 Entamoeba,222,223 Cryptosporidium,224 Nematódeos,225,226 entre outros. Dentre os trabalhos envolvendo análise metabolômica global, destaca-se um desenvolvimento sistemático de extração de metabólitos em amostras de Fasciola hepatica, um parasita que causa inflamação crônica no fígado de seus hospedeiros (boi, ovelha, porco e homem).227 A otimização da extração foi realizada por desenho experimental de misturas de solventes (metanol, água e clorofórmio), e envolveu o uso de multiplataformas de análise, com LC-MS (fase reversa e de interação hidrofílica, modos positivo e negativo), e CE-MS (modo positivo), para separação e detecção dos compostos extraídos diretamente do verme. Nestes estudos foram detectados 142 metabólitos oriundos de 14724 entidades moleculares. A Figura 7A apresenta um diagrama de Venn com a indicação dos 142 metabólitos significativos detectados pelas 5 plataformas analíticas, comparando-as entre si e demonstrando a capacidade de detecção de cada uma e seletividade das técnicas de análise utilizadas. Como observado na Figura 7A, não existe técnica de análise universal, uma vez que a análise por cromatografia em fase reversa facilita a separação de compostos com características mais apolares, como derivados de fosfolipídios, as colunas para interação hidrofílica, favorecem a separação de compostos de polaridade intermediária, como aminoácidos e seus derivados, nucleotídeos, etc., e a eletroforese capilar é responsável pela separação de compostos polares ionizáveis, e de cadeia curta, como colinas e alguns aminoácidos, sendo, portanto, essas técnicas complementares, acessando diferentes classes químicas.228 A complementaridade das plataformas analíticas em evidência proporciona uma maior cobertura do metaboloma, aprimorando o entendimento biológico do sistema estudado.
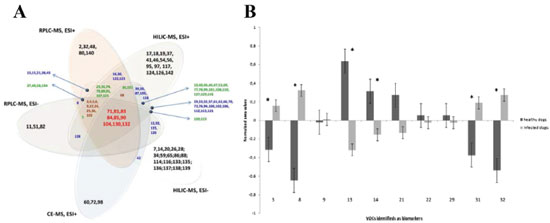 Figura 7. Exemplos de estudo em metabolômica em parasitologia. A) Diagrama de Venn dos 142 metabólitos identificados por RPLC-MS (modos positivo e negativo), HILIC-MS (modos positivo e negativo) e CE-MS (modo positivo). Metabólitos coloridos de acordo com o número de técnicas de análise em comum. Reproduzido da referência 228 com permissão da ACS. B) Comparação das áreas normalizadas de metabólitos discriminantes entre grupos de cães saudáveis e infectados com L. infantum. Metabólitos marcados com asterisco apresentam alta significância estatística (p-valor < 0,05) e são considerados biomarcadores. Reproduzido da referência 229 com permissão da Springer
A metabolômica alvo para análise de compostos orgânicos voláteis foi aplicada com a finalidade de diagnóstico de leishmaniose em cães, no trabalho de Magalhães Jr et al. Três grupos de amostras de pelos de cães saudáveis e infectados com Leishmania infantum (apresentando ou não sinais clínicos) foram analisados por GC-MS e revelaram diferentes perfis dos compostos detectados. Após tratamento e análise estatística multivariada dos dados normalizados, os autores encontraram seis candidatos a biomarcadores (octanal, nonanal, b-hidrixietil fenil éter, decanal, heptadecano e 2-etilhexil-salicilato), identificados com asterisco na Figura 7B. Esses seis compostos foram considerados biomarcadores por caracterizarem a presença ou ausência da doença. A diferença nos níveis desses biomarcadores foi atribuída a interações metabólicas entre o parasita e as células dos cães, e poderia ainda indicar a intensidade da doença.229 Um trabalho como este confirma a importância dos estudos de metabolômica na parasitologia, pois através de uma análise simples, com um método de coleta de amostra não invasivo, é possível diagnosticar a doença, facilitando seu controle e prevenção. Plantas Devido à grande complexidade química das plantas, onde estima-se a existência de mais de 200.000 metabólitos,230 a análise metabolômica é uma abordagem que se mostra muito interessante, pois além de auxiliar no entendimento do metabolismo desses seres vivos, também pode trazer melhorias em termos de rendimento nos cultivos.231 O número de publicações e o interesse na área é tão grande que, em 2013, um Handbook voltado para análise metabolômica em plantas foi publicado, no qual são destacados trabalhos interessantes apresentando protocolos e métodos de análise, tratamento de dados, e destaques para rotas metabólicas.232 Em uma busca rápida nas bases de dados de periódicos é possível encontrar nos últimos dezessete anos mais de 100 artigos de revisão na área, em diversas aplicações.233-239 Dentre estas revisões, destacam-se algumas com enfoque em estudos de alterações ambientais71,77 e genéticas,240-242 resposta ao estresse,243-245 saúde e tratamento de doenças,246-248 ecologia e evolução,63,76 agricultura,249,250 bem como trabalhos dedicados à metabolômica com uso de compostos isotópicos,251,252 que auxiliam na elucidação de rotas e melhoram a identificação e quantificação de metabólitos. Também são frequentemente encontrados estudos envolvendo mais de uma abordagem ômica, como proteômica e transcriptômica.253-255 Um trabalho interessante aplicado a alterações ambientais com análise metabolômica global em cultivos de tomate, beterraba, tremoço e pessegueiro, mantidos sob condições normais e sob deficiência de ferro, foi apresentado por Rellán-Álvarez et al. A análise de extratos das folhas e seiva do caule (xilema) foi real, izada por GC-TOF-MS, possibilitando a detecção de mais de 200 metabólitos para cada grupo de amostras. Diversas classes de metabólitos, como aminoácidos e carboidratos, foram encontradas significativamente alteradas nas comparações entre os grupos estudados. A Figura 8A apresenta uma compilação dos metabólitos alterados, correlacionando-os com suas respectivas rotas metabólicas. Foi observado que nas amostras de seiva (xilema) grande parte dos metabólitos alterados estão diminuídos, enquanto que nas amostras das folhas estão aumentados com a deficiência de ferro. Os autores concluíram que existe uma correlação significativa entre aminoácidos e metabólitos do ciclo do ácido cítrico (TCA) na sobrevivência a curto prazo das plantas analisadas em ausência de ferro, o que pode afetar diretamente a produtividade da cultura.256
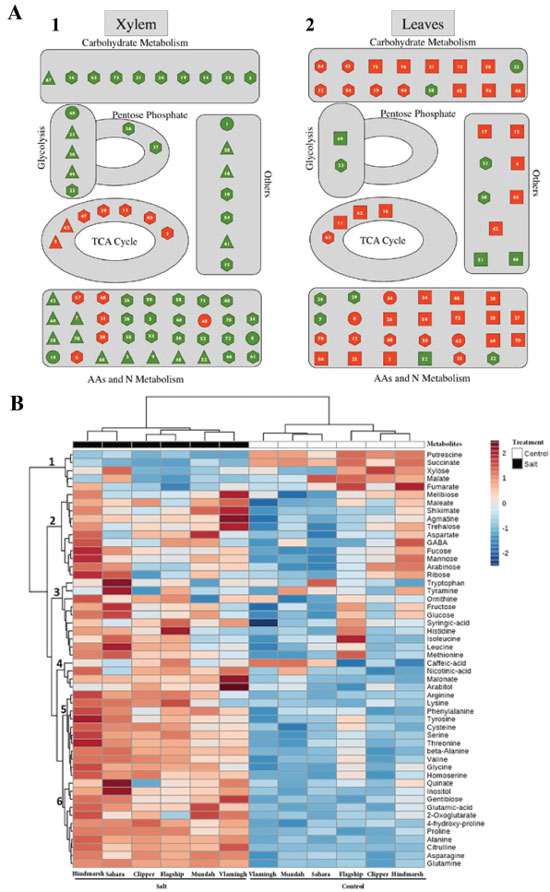 Figura 8. Exemplos de estudo em metabolômica de plantas. A) Alterações nas rotas metabólicas afetadas pela deficiência de ferro em cultivos de tomate, beterraba, tremoço e pessegueiro em 1) amostras de caule e 2) extratos de folhas. Símbolos em verde significam diminuídos e em vermelho aumentados. Legenda: (⬢) refere-se a amostras de tomate, (▲) tremoço, (■) beterraba e (●) pessegueiro. Reproduzido da referência 256 com permissão da Frontiers. B) Heatmap dos metabólitos detectados em seis variedades de raízes de cevada, sob interferência de estresse salino e suas alterações. Reproduzido da referência 257 com permissão da Frontiers
Um estudo recente do efeito da salinidade foi realizado por meio da avaliação do teor de dez fitohormônios e seus metabólitos em seis variedades de raízes de cevada, submetidas ou não a estresse salino. Para a quantificação dos metabólitos alvos, um método foi desenvolvido e validado por LC-MS. O método de análise se mostrou bastante rápido, com a separação dos dez analitos em menos de 10 min e com boa recuperação dos metabólitos. Na comparação entre os grupos estudados foram observadas diferentes alterações entre os fitohormônios e seus metabólitos (especialmente aminoácidos) (Figura 8B), indicando que essas mudanças metabólicas dependem da variedade da cevada. Estes estudos apresentaram resultados que contribuem para um melhor entendimento no que se refere à resposta ao estresse salino em plantas, em nível molecular, e como o estresse afeta a produção de fitohormônios, o crescimento, o desenvolvimento e o metabolismo de plantas.257 Toxicologia Forense A toxicologia é uma ciência secular, que estuda os efeitos adversos causados por substâncias químicas em organismos vivos. A intoxicação é um fenômeno complexo de alteração da homeostasia de um organismo, induzida por um xenobiótico (agente tóxico), e seu grau depende de fatores como a toxicidade da substância química (xenobiótico), quantidade (concentração ou dose), via (oral, dérmica, pulmonar), frequência e duração da exposição, além de características individuais do organismo exposto (espécie, idade, sexo, estado nutricional e de saúde). Ainda que um xenobiótico exerça sua ação tóxica em um órgão ou tecido específico (que geralmente é o foco principal dos estudos toxicológicos), deve-se considerar também sua ação secundária no indivíduo intoxicado. A metabolômica tem sido aplicada na toxicologia forense e trabalhos nessa área têm sido revisados frequentemente,258-262 mostrando que o estudo comparativo do perfil metabólico pode desempenhar um papel importante na descoberta e validação de novos biomarcadores, além de contribuir para o entendimento e consequente interpretação dos mecanismos de ação tóxica de xenobióticos.263 A dependência de drogas como cocaína, anfetaminas, opióides e etanol é um grave problema de saúde pública e, recentemente, estudos de metabolômica vem sendo conduzidos para tentar explicar (ou complementar) o entendimento da toxicologia das drogas de abuso,264-268 na busca por biomarcadores para hepatotoxicidade e nefrotoxicidade induzida pela ação de drogas269,270 e, ainda, métodos de quantificação de drogas e seus metabólitos vêm sendo desenvolvidos para aplicação em diversas matrizes biológicas.271 No que tange às ciências forenses, a metabolômica tem sido utilizada em diferentes aplicações, com análise em diversos biofluidos como urina, plasma, cabelo, unha, saliva, etc.,272 além de investigações de causas de morte273 e intervalo pós-morte.274 A avaliação da dependência de drogas por metabolômica global em amostras de plasma e urina de ratos foi realizada por Zaitsu et al. Os animais foram tratados com doses repetidas de morfina (4 mg/kg), metanfetamina (2 mg/kg) e cocaína (10 mg/kg). Amostras de urina foram coletadas antes do início da administração dos fármacos e no 12º dia após o início do tratamento, quando também foram coletadas as amostras de plasma. As amostras foram analisadas por GC-MS e os resultados da análise multivariada não-supervisionada (PCA) estão apresentados na Figura 9A. Dessas análises, os autores identificaram 57 metabólitos nas amostras de plasma e 70 metabólitos nas amostras de urina, e puderam observar alterações significativas no metaboloma dos animais tratados com morfina, porém, o mesmo não ocorreu nos animais tratados com as demais drogas de abuso estudadas, inferindo-se que a alteração do metaboloma na dependência está diretamente relacionada ao mecanismos de ação de cada substância no centro de recompensa no cérebro.275
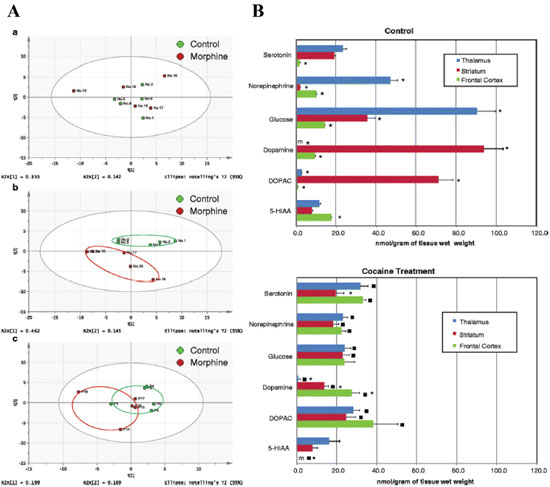 Figura 9. Exemplos de estudo em metabolômica em toxicologia forense. A) Gráfico de scores de modelo PCA de amostras de urina e plasma de ratos dos grupos controle (verde) e tratado com 4 mg/kg morfina (vermelho). (a) amostras de urina coletadas antes da administração de morfina; (b) amostras de urina coletadas após o tratamento (e dependência) de morfina; (c) amostras de plasma coletadas após o tratamento (e dependência) de morfina. Reproduzido da referência 275 com permissão da Springer. B) Concentrações de neurotransmissores e metabólitos (em nmol/g) em diferentes regiões do cérebro de ratos não tratados e tratados com cocaína. As diferenças estatisticamente significativas (intervalo de confiança de 95%) são representadas da seguinte forma: o único asterisco indica diferenças para uma substância determinada entre as três áreas anatômicas; os quadrados indicam diferenças entre os grupos tratados e não tratados com cocaína; (m) indica valores abaixo do limite de detecção. Reproduzido da referência 276 com permissão da Springer
Outro trabalho interessante que envolve a análise de drogas de abuso foi publicado por Kaplan et al. Neste trabalho, a espectrometria de massas por mobilidade iônica foi a técnica de análise para estudar o efeito da cocaína no metabolismo da glicose e de aminas biogênicas, em regiões específicas do cérebro de ratos. Utilizando uma abordagem metabolômica alvo, os autores determinaram a presença de diversos neurotransmissores e seus metabólitos, tais como noradrenalina, serotonina, ácido 5-hidroxi-indolacético, dopamina, e ácido 3,4-dihidroxifenilacético, no córtex frontal, estriado e tálamo de ratos controle e tratados com cocaína. A concentração destas substâncias foi medida por adição de padrão e permitiu distinguir entre as diferentes áreas anatômicas do cérebro, bem como observar que a concentração de alguns destes neurotransmissores (ou seus metabólitos) é alterada significativamente em animais que receberam cocaína, como exemplificado na Figura 9B. Na avaliação metabolômica global, os autores identificaram mais de 200 entidades moleculares que permitiram a distinção entre grupos tratados e controle.276
CONCLUSÃO Este artigo de revisão teve como objetivo apresentar as definições que permeiam as análises metabolômicas realizadas nos estudos de biologia de sistemas, bem como apresentar as etapas envolvidas em investigações focadas em análises global e alvo. As compilações de trabalhos publicados em áreas representativas de interesse, feitas nesta revisão, mostram de forma inequívoca o estabelecimento da metabolômica nos últimos anos. O amadurecimento desta ciência se deve ao crescimento na busca pela compreensão dos mecanismos envolvidos nos sistemas biológicos, além dos avanços das técnicas analíticas e o aprimoramento das ferramentas de tratamento de dados.
AGRADECIMENTOS Os autores agradecem a Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP - processos 2012/07361-6, 2012/51692-7, 2012/09822-0, 2015/10650-8), e ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq - processos 407354-2013-1, 306739-2014-3, 405677-2015-4, 152064/2016-8).
REFERÊNCIAS 1. Fiehn, O.; Plant Mol. Biol. 2002, 48, 155 2. Oliver, S. G.; Winson, M. K.; Kell, D. B.; Baganz, F.; Trends Biotechnol. 1998, 16, 373. 3. Nicholson, J.; Lindon, J. C.; Holmes, E.; Xenobiotica 1999, 11, 1181. 4. Fiehn, O.; Comp. Funct. Genom. 2001, 2, 155. 5. Horning, E. C.; Horning, M. G.; J. Chromatogr. Sci. 1971, 9, 129. 6. Kell, D. B.; Brown, M.; Davey, H. M.; Dunn, W. B.; Spasic, I.; Oliver, S. G.; Nat. Rev. Microbiol. 2005, 3, 557. 7. Nicholson, J. K.; Lindon, J. C.; Nature 2008, 455, 1054. 8. Kuehnbaum, N. L.; Britz-McKibbin, P.; Chem. Rev. 2013, 113, 2437. 9. Klassen, A.; Faccio, A. T.; Canuto, G. A. B.; da Cruz, P. L. R.; Ribeiro, H. C.; Tavares, M. F. M.; Sussulini, A.; Em Metabolomics: From Fundamentals to Clinical Applications;Sussulini, A., ed.; Springer: Switzerland, 2017, cap. 1. 10. Viant, M. R.; Kurland, I. J.; Jones, M. R.; Dunn, W. D.; Curr. Opin. Chem. Biol. 2017, 36, 64. 11. Vuckovic, D.; Anal. Bioanal. Chem. 2012, 402, 1523. 12. Álvarez-Sáchez, B.; Priego-Capote, F.; Luque de Castro, M. D.; Trends Anal. Chem. 2010, 29, 111. 13. Raterink, R.-J.; Lindenburg, P. W.; Vreeken, R. J.; Ramautar, R.; Hankemeier, T.; Trends Anal. Chem. 2014, 61, 157. 14. Kuehnbaum, N. L.; Gillen, J. B.; Kormendi, A.; Lam, K. P.; Dibattista, A.; Gibala, M. J.; Britz-Mckibbin, P.; Electrophoresis 2015, 36, 2226. 15. Garcia, A.; Barbas, C.; Em Gas Chromatography-mass Spectrometry (GC-MS)-based metabolomics;Metz, T. O., ed.; Spinger: New York, 2011. 16. Theodoridis, G. A.; Gika, H. G.; Want, E. J.; Wilson, I. D.; Anal. Chim. Acta 2012, 711, 7. 17. Dunn, W. B.; Wilson, I. D.; Nicholls, A. W.; Broadhurst, D.; Bioanalysis 2012, 4, 2249. 18. Villas-Bôas, S. G.; Bruheim, P.; OMICS 2007, 11, 305. 19. Dunn, W. B.; Ellis, D. I.; Trends Anal. Chem. 2005, 24, 285. 20. Beckonert, O.; Keun, H. C.; Ebbels, T. M. D.; Bundy, J.; Holmes, E.; Lindon, J. C.; Nicholson, J. K.; Nat. Protoc. 2007, 2, 2692. 21. Ludwig, C.; Viant, M. R.; Phytochem. Anal. 2010, 21, 22. 22. de Raad, M.; Fischer, C. R.; Northen, T. R.; Curr. Opin. Chem. Biol. 2016, 30, 7. 23. Dias, D. A.; Jones, O. A.; Beale, D. J.; Boughton, B. A.; Benheim, D.; Kouremenos, K. A.; Wolfender, J. L.; Wishart, D. S. Metabolites 2016, 6, E46. 24. Putri, S. P.; Yamamoto, S.; Tsugawa, H.; Fukusaki, E.; J. Biosci. Bioeng. 2013, 116, 9. 25. Lenz, E. M.; Wilson, I. D. J. Proteome Res. 2007, 6, 443. 26. Wishart, D. S.; Trends Anal. Chem. 2008, 27, 228. 27. Han, J.; Danell, R. M.; Patel, J. R.; Gumerov, D. R.; Scarlett, C. O.; Speir, J. P.; Parker, C. E.; Rusyn, I.; Zeisel, S.; Borchers, C. H.; Metabolomics 2008, 4, 128. 28. Mirnezami, R.; Spagou, K.; Vorkas, P. A.; Lewis, M. R.; Kinross, J.; Want, E.; Shion, H.; Goldin, R. D.; Darzi, A.; Takats, Z.; Holmes, E.; Cloarec, O.; Nicholson, J. K.; Mol. Oncol. 2014, 8, 39. 29. Fiehn, O.; Trends Anal. Chem. 2008, 27, 261. 30. Jonsson, P.; Gullberg, J.; Nordstrom, A.; Kusano, M.; Kowalczyk, M.; Sjostrom, M.; Moritz, T.; Anal. Chem. 2004, 76, 1738. 31. Koek, M. M.; Jellema, R. H.; van der Greef, J.; Tas, C. A.; Hankemeier, T.; Metabolomics 2011, 7, 307. 32. Hill, C. B.; Roessner, U.; Em Metabolic Profiling of Plants by GC-MS;Weckwerth, W.; Kahl, G., eds.; Wiley-VHC Verlag GmbH & Co: Weinheim, 2013. 33. Forcisi, S.; Moritz, F.; Kanawati, B.; Tziotis, D.; Lehmann, R.; Schmitt-Kopplin, P.; J Chromatogr. A 2013, 1292, 51. 34. Zhou, B.; Xiao, J. F.; Tuli, L.; Ressom, H. W.; Mol. BioSyst. 2012, 8, 470. 35. Cubbon, S.; Antonio, C.; Wilson, J.; Thomas-Oates, J.; Mass Spectrom. Rev. 2010, 29, 671. 36. Kloos, D.-P.; Lingeman, H.; Niessen, W. M. A.; Deelder, A. M.; Giera, M.; Mayboroda, O. A.; J. Chromatogr. B 2013, 927, 90. 37. Xiayan, L.; Legido-Quigley, C., Electrophoresis 2008, 29, 3724. 38. Hirayama, A; Wakayama, M.; Soga, T.; Trends Anal. Chem. 2014, 61, 215. 39. Barbas, C.; Moraes, E. P.; Viallaseñor, A.; J. Pharmaceut. Biomed. 2011, 55, 823. 40. Ramautar, R.; Somsen, G. W.; Jong, G. J.; Electrophoresis 2015, 36, 212. 41. Ramautar, R.; Busnel, J. M.; Deelder, A. M.; Mayboroda, O. A.; Anal. Chem. 2012, 84, 885. 42. Sugimoto, M.; Kawakami, M.; Robert, M.; Soga, T.; Tomita, M.; Curr. Bioinform. 2012, 7, 96. 43. Ludwig, C.; Günther, U. L.; BMC Bioinf. 2011, 12, 366. 44. Smith, C. A.; Want, E. J.; O'Maille, G.; Abagyan, R.; Siuzdak, G.; Anal. Chem. 2006, 78, 779. 45. Katajamaa, M.; Miettinen, J.; Oresic, M.; Bioinformatics 2006, 22, 634. 46. Lommen, A.; Anal. Chem. 2009, 81, 3079. 47. http://www.amdis.net, acessado em outubro de 2017. 48. Misra, B. B.; van der Hooft, J. J. J.; Electrophoresis 2016, 37, 86. 49. Koal, T.; Deigner, H.-P.; Curr. Mol. Med. 2010, 10, 216. 50. Lu, W.; Bennett, B. D.; Rabinowitz, J. D.; J. Chromatogr. B. 2008, 871, 236. 51. Guo, B.; Chen, B.; Liu, A.; Zhu, W.; Yao, S.; Curr. Drug Metab. 2012, 13, 1226. 52. Wishart, D. S.; Tzur, D.; Knox, C.; Eisner, R.; Guo, A. C.; Young, N.; Cheng, D.; Jewell, K.; Arndt, D.; Sawhney, S.; Fung, C.; Nikolai, L.; Lewis, M.; Coutouly, M. A.; Forsythe, I.; Tang, P.; Shrivastava, S.; Jeroncic, K.; Stothard, P.; Amegbey, G.; Block, D.; Hau, D. D.; Wagner, J.; Miniaci, J.; Clements, M.; Gebremedhin, M.; Guo, N.; Zhang, Y.; Duggan, G. E.; Macinnis, G. D.; Weljie, A. M.; Dowlatabadi, R.; Bamforth, F.; Clive, D.; Greiner, R.; Li, L.; Marrie, T.; Sykes, B. D.; Vogel, H. J.; Querengesser, L.; Nucleic Acids Res. 2007, 35, D521. 53. Tautenhahn, R.; Cho, K.; Uritboonthai, W.; Zhu, Z. J.; Patti, G. J.; Siuzdak, G.; Nat. Biotechnol. 2012, 30, 826. 54. Horai, H.; Arita, M.; Kanaya, S.; Nihei, Y.; Ikeda, T.; Suwa, K.; Ojima, Y.; Tanaka, K.; Tanaka, S.; Aoshima, K.; Oda, Y.; Kakazu, Y.; Kusano, M.; Tohge, T.; Matsuda, F.; Sawada, Y.; Hirai, M. Y.; Nakanishi, H.; Ikeda, K.; Akimoto, N.; Maoka, T.; Takahashi, H.; Ara, T.; Sakurai, N.; Suzuki, H.; Shibata, D.; Neumann, S.; Lida, T.; Tanaka, K.; Funatsu, K.; Matsuura, F.; Soga, T.; Taguchi, R.; Saito, K.; Nishioka, T.; J. Mass Spectrom. 2010, 45, 703. 55. Karp, P. D.; Caspi, R.; Arch. Toxicol. 2011, 85, 1015. 56. Kanehisa, M.; Goto, S.; Hattori, M.; Aoki-Kinoshita, K. F.; Itoh, M.; Kawashima, S.; Katayama, T.; Araki, M.; Hirakawa, M.; Nucleic Acids Res. 2006, 34, D354. 57. Caspi, R.; Altman, T.; Dreher, K.; Fulcher, C. A.; Subhraveti, P.; Keseler, I. M.; Kothari, A.; Krummenacker, M.; Latendresse, M.; Mueller, L. A.; Ong, Q.; Paley, S.; Pujar, A.; Shearer, A. G.; Travers, M.; Weerasinghe, D.; Zhang, P.; Karp, P. D.; Nucleic Acids Res. 2012, 40, D742. 58. Haug, K.; Salek, R. M.; Conesa, P.; Hastings, J.; Matos, P.; Rijnbeek, M.; Mahendraker, T.; Williams, M.; Neumann, S.; Rocca-Serra, P.; Maguire, E.; González-Beltrán, A.; Sansone, S.-A.; Griffin, J. L.; Steinbeck, C.; Nucleic Acids Res. 2013, 41, D781. 59. Madsen, R.; Lundstedt, T.; Trygg, J.; Anal. Chim. Acta. 2010, 659, 23. 60. Liesenfeld, D. B.; Habermann, N.; Owen, R. W.; Scalbert, A.; Ulrich, C. M.; Cancer Epidemiol. Biomarkers Prev. 2013, 22, 2182. 61. Morrison, N.; Bearden, D.; Bundy, J. G.; Collette, T.; Currie, F.; Davey, M. P.; Haigh, N. S.; Hancock, D.; Jones, O. A. H.; Rochfort, S.; Sansone, S. -A.; Stys, D.; Teng, Q.; Field, D.; Viant, M. R.; Metabolomics 2007, 3, 203. 62. Bundy, J. G.; Davey, M. P.; Viant; M. R.; Metabolomics 2009, 5, 3. 63. Jones, O. A. H.; Maguire, M. L.; Griffin, J. L.; Dias, D. A.; Spurgeon, D. J.; Svendsen, C.; Austral Ecol. 2013, 38, 713. 64. Lankadurai, B. P.; Nagato, E. G.; Simpson, M. J.; Environ. Rev. 2013, 21, 180. 65. Sardans, J.; Peñuelas, J.; Rivas-Ubach, A.; Chemoecology 2011,21, 191. 66. Viant, M. R.; Sommer, U.; Metabolomics 2013, 9, S144. 67. Gordon, B. R.; Leggat, W.; Mar. Drugs 2010, 8, 2546. 68. Goulitquer, S.; Potin, P.; Tonon, T.; Mar. Drugs 2012, 10, 849. 69. Samuelsson, L. M.; Larsson, D. G. J.; Mol. BioSyst. 2008, 4, 974. 70. Kumar, M.; Kuzhiumparambil, U.; Pernice, M.; Jiang, Z.; Ralph, P. J.; Algal Res. 2016, 16, 76. 71. Brunetti, C.; George, R. M.; Tattini, M.; Field, K.; Davey, M. P.; J. Exp. Bot. 2013, 13, 4011. 72. Noctor, G.; Lelarge-Trouverie, C.; Mhamdi, A.; Phytochemistry 2015, 112, 33. 73. Král'ová, K.; Jampílek, J.; Ostrovský, I.; Ecol. Chem. Eng. S. 2012, 19,133. 74. Jorge, T. F.; Rodrigues, J. A.; Caldana, C.; Schmidt, R.; van Dongen, J. T.; Thomas-Oates, J.; António, C.; Mass Spectrom. Rev. 2016, 35, 620. 75. Nakabayashi, R.; Saito, K.; Curr. Opin. Plant Biol. 2015, 24,10. 76. Macel, M.; Van Dam, N. M.; Keurentjes, J. J.; Mol. Ecol. Resour. 2010, 10, 583. 77. Park, S.; Seo, Y.-S.; Hegeman, A. D.; J. Plant Biol. 2014, 57, 137. 78. Simpson, M. J.; McKelvie, J. R.; Anal. Bioanal. Chem. 2009, 394, 137. 79. Aliferis, K. A.; Chrysayi-Tokousbalides, M.; Metabolomics 2011, 7, 35. 80. Wang, P.; Wu, Y. J.; Curr. Drug Metab. 2015, 16, 191. 81. Aliferis, K. A.; Jabaji, S.; Pestic. Biochem. Physiol. 2011, 100, 105. 82. García-Sevillano, M. A.; García-Barrera, T.; Abrild, N.; Pueyod, C.; López-Baread, J.; Gómez-Ariza, J. L. J.; Proteomics 2014, 104, 4. 83. Booth, S. C.; Workentine, M. L.; Weljie, A. M.; Turner, R. J.; Metallomics 2011, 3, 1142. 84. Garcia-Sevillano, M. A.; Garcia-Barrera, T.; Gomez-Ariza, J. L.; Electrophoresis 2015, 36, 2348. 85. Jones, O. A. H.; Dias, D. A.; Callahan, D. L.; Kouremenos, K. A.; Bealee, D. J.; Roessner, U.; Metallomics 2015, 7, 29 86. Singh, O. V.; Proteomics 2006, 6, 5481. 87. Currie, F.; Broadhurst, D. I.; Dunn, W. B.; Sellick, C. A.; Goodacre, R.; Mol. BioSyst. 2016, 12, 1367. 88. Southam, A. D.; Lange, A.; Hines, A.; Hill, E. M.; Katsu, Y.; Iguchi, T.; Tyler, C. R.; Viant, M. R.; Environ Sci Technol. 2011, 45, 3759. 89. Mamas, M.; Warwick, B.; Dunn, L. N.; Royston, G.; Arch. Toxicol. 2011, 85, 5. 90. Zhang, A.; Sun, H.; Yan, G.; Wang, P.; Wang, X.; BioMed Res. Int. 2015, 2015, 1. 91. Baldeep, K.; Ajay, P.; Rakesh, K.; Ruhela, B. M.; Pharmacol. Rep. 2014, 66, 956. 92. Mastrangelo, A.; Armitage, E. G.; García, A.; Barbas, C.; Curr. Top. Med. Chem. 2014, 14, 2627. 93. Spratlin, JL.; Serkova, N. J.; Eckhardt, S. G.; Clin. Cancer Res. 2009, 15, 431. 94. Beger, R.; Metabolites 2013, 3, 552. 95. Olivares, O.; Däbritz, J.M.; Kinga, A.; Gottlieb, E.; Halseya, C.; Semin. Cell Dev. Biol. 2015, 43, 52. 96. Lima, A. R.; Bastos, M. L; Carvalho, M.; Pinho, P. G.; Transl. Oncol. 2016, 9, 357. 97. Chan, A. W.; Gill, R. S.; Schiller, D.; Sawyer, M. B.; World J. Gastroenterol. 2014, 20, 12874. 98. Zhang, A.; Sun, H.; Yan, G.; Wang, P.; Han, Y.; Wang, X.; Cancer Lett. 2014, 345, 17. 99. Penkert, J.; Ripperger, T.; Schieck, M.; Schlegelberger, B.; Steinemann, D.; Illig, T.; Oncotarget 2016, 41, 67626. 100. Zhang, A.; Qiu, S.; Xu, H.; Sun, H.; Wang, X.; Clin. Chim. Acta 2014, 429, 106. 101. Klein, M. S.; Shearer, J.; J. Diabetes Res. 2016, ID 3898502 102. Pallares-Méndez, R.; Aguilar-Salinas, C. A.; Cruz-Bautista, I.; Del Bosque-Plata, L.; Ann. Med. 2016, 48, 89. 103. Djekic, D.; Nicoll, R.; Novo, M.; Henein, M.; IJC Metabolic & Endocrine 2015, 8, 26. 104. Lam, S. M.; Wang, Y.; Li, B.; Du, J.; Shui, G.; J. Genet. Genomics 2017, 44, 127. 105. D'Agostino, R. B.; Russell, M. W.; Huse, D. M.; Ellison, R. C.; Silbershatz, H.; Wilson, P. W.; Hartz, S. C.; Am. Heart J. 2000, 139, 272. 106. Trushina, E.; Mielke, M. M.; Biochim. Biophys. Acta 2014, 1842, 1232. 107. Preez, I.; Lutz, D. T.; Metabolomics 2014, 10, 877. 108. Hanna, M. H.; Brophy, P. D.; Pediatr. Nephrol. 2015, 30, 881. 109. Rhee, E. P.; Clish, C. B.; Wenger, J.; Roy, J.; Elmariah, S.; Pierce, K. A.; Bullock, K.; Anderson, A. H.; Gerszten, R. E.; Feldman, H. I.; Am. J. Nephrol. 2016, 43, 366. 110. Córdoba, D. L.; Castro, M. D. L.; J. Pharm. Biomed. Anal. 2017, 136, 22. 111. Ryan, D.; Newnham, E. D.; Prenzler, P. D.; Gibson, P. R.; Metabolomics 2015, 11, 980. 112. Serrano-Villar, S.; Rojo, D.; Martínez-Martínez, M.; Deusch, S.; Vázquez-Castellanos, J. F.; Bargiela, R.; Sainz, T.; Vera, M.; Moreno, S.; Estrada, V.; Gosalbes, M. J.; Latorre, A.; Seifert, J.; Barbas, C.; Moya, A.; Ferrer, M.; EBio Medicine 2016, 8, 203. 113. Kamphorst, J. J.; Chung, M. K.; Fan, J.; Rabinowitz, J. D.; Cancer Metab. 2014, 2, 23. 114. Heaney, L. M.; Deighton, K.; Suzuki, T.; J. Sports Sci. (2017), doi: 10.1080/02640414.2017.1305122. DOI: http://dx.doi.org/10.1080/02640414.2017.1305122. 115. Riedmaier, I.; Becker, C.; Pfaffl, M. W.; Meyer, H. H. D. J. Chromatogr. A 2009, 1216, 8192. 116. Pottigiesser, T.; Schumacher, Y. O.; Anal. Bioanal. Chem. 2013, 405, 9625. 117. Duntas, L. H.; Popovic, V.; Endocrine 2013, 43, 303. 118. Pitsiladis, Y. P.; Durussel, J.; Rabin, O.; Br. J. Sports Med. 2014, 48, 856. 119. Mena-Bravo, A.; Luque de Castro, M. D.; J. Pharm. Biomed. Anal. 2014, 90, 139. 120. Teale, P.; Barton, C.; Driver, P. M.; Kay, R. G.; Bioanalysis 2009, 1, 1103. 121. Barroso, O.; Handelsman, D. J.; Strasburger, C.; Thevis, M.; Bioanalysis 2012, 4, 1577. 122. Waller, C. C.; McLeod, M. D.; Drug Test Anal. (2016), doi: 10.1002/dta.2112. DOI: http://dx.doi.org/10.1002/dta.2112 123. Fragkaki, A. G.; Kioukia-Fougia, N.; Kiousi, P.; Kioussi, M.; Tsivou, M.; Drug Test Anal. 2017, 9, 1291. 124. van Hall, G.; Sports Med. 2015, 45, S23. 125. Weigert, C.; Lehmann, R.; Hartwig, S.; Lehr, S.; Proteomics Clin. Appl. 2014, 8, 5. 126. Riebe, D.; Franklin, B. A.; Thompson, P. D.; Garber, C. E.; Whitfield, G. P.; Magal, M.; Pescatello, L. S.; Med. Sci. Sports Exerc. 2015, 47, 2473. 127. Netzer, M.; Weinberger, K. M.; Handler, M.; Seger, M.; Fang, X.; Kugler, K. G.; Graber, A.; Baumgartner, C.; J. Clin. Bioinforma. 2011, 19, 34. 128. López-Otín, C.; Galluzzi, L.; Freije, J. M.; Madeo, F.; Kroemer, G.; Cell 2016, 11, 802. 129. Kim, I. Y.; Suh, S. H.; Lee, I. K.; Wolfe, R. R.; Exp. Mol. Med. 2016, 15, e203. 130. Radom-Aizik, S.; Cooper, D. M.; Pediatr. Exerc. Sci. 2016, 28, 194. 131. Cid, M.; González, M.; Early Hum. Dev. 2016, 94, 57. 132. May, L. E.; Allen, J. J. B.; Gustafson, K. M.; Early Hum. Dev. 2016, 94, 49. 133. Bassini, A.; Cameron, L. C.; Biochem. Biophys. Res. Commun. 2014, 445, 708. 134. Kuehnbaum, N. L.; Gillen, J. B.; Kormendi, A.; Lam, K. P.; Dibattista, A.; Gibala, M. J.; Britz-Mckibbin, P.; Electrophoresis 2015, 36, 2226. 135. Chan, G. H. M.; Ho, E. N. M.; Leung, D. K. K.; Wong, K. S.; Wan, T. S. M.; Anal. Chem. 2016, 88, 764. 136. Herrero, M.; Simó, C.; García-Cañas, V.; Ibázez, E.; Cifuentes, A.; Mass Spec. Rev. 2012, 31, 49. 137. Capozzi, F.; Bordoni, A.; Genes Nutr. (2013), doi 10.1007/s12263-012-0310-x. DOI: http://dx.doi.org/10.1007/s12263-012-0310-x 138. Laghi, L.; Picone, G.; Capozzi, F.; Trends Anal. Chem. 2014, 59, 93. 139. Wang, X.; Wang, S.; Cai, Z.; Trends Anal. Chem. 2013, 52, 170. 140. García-Cañas, V.; Simó, C.; Castro-Puyana, M.; Cifuentes, A.; Electrophoresis 2014, 35, 147. 141. Skov, T.; Honoré, A. H.; Jensen, H. M.; Naes, T.; Engelsen, S. B.; Trends Anal. Chem. 2014, 60, 71. 142. Zielinski, A. A. F.; Haminiuk, C. W. I.; Nunes, C. A.; Schnitzler, E.; van Ruth, S. M., Granato, D.; Compr. Rev. Food Sci. Food Saf. 2014, 13, 330. 143. Cevallos-Cevallos, J. M.; Reyes-De-Corcuera, J. I.; Etxeberria, E.; Danyluk, M. D.; Rodrick, G. E.; Trends Food Sci. Technol. 2009, 20, 557. 144. Cubero-Leon, E.; Peñalver, R.; Maquet, A.; Food Res. Int. 2014, 60, 95. 145. Ortea, I.; Pascoal, A.; Cañas, B.; Gallardo, J. M.; Barros-Velázquez, J.; Calo-Mata, P.; Electrophoresis 2012, 33, 2201. 146. Giacometti, J.; Tomljanović, A. B.; Josić, D. ; Food Res. Int. 2013, 54, 1042. 147. Giacometti, J.; Josić, D. ; Trends Anal. Chem. 2013, 52, 16. 148. Kuang, H.; Li, Z.; Peng, C.; Liu, L.; Xu, L.; Zhu, Y.; Wang, L.; Xu, C.; Crit. Rev. Food Sc. Nutr. 2012, 52, 761. 149. Oms-Oliu, G.; Odriozola-Serrano, I.; Martín-Belloso, O.; Food Res. Int. 2013, 54, 1172. 150. Scalbert, A.; Brennan, L.; Manach, C.; Andres-Lacueva, C.; Dragsted, L. O.; Draper, J.; Rappaport, S. M.; van der Hooft, J. J. J.; Wishart, D. S.; Am. J. Clin. Nutr. 2014, 99, 1286. 151. Puiggròs, F.; Solà, R.; Bladé, C.; Salvadó, M.-J.; Arola, L. J.; Chromatogr. A 2011, 1218, 7399. 152. Wishart, D. S.; Trends Food Sci. Technol. 2008, 19, 482-493. 153. Castro-Puyana, M.; Mendiola, J. A.; Ibañez, E.; Trends Anal. Chem. 2013, 52, 23. 154. Mannina, L.; Sobolev, A. P.; Capitani, D.; Electrophoresis 2012, 33, 2290. 155. Medina, S.; Domínguez-Perles, R.; Ferrenes, F.; Tomás-Barberán, F. A.; Gil-Izquierdo, Á.; Trends Anal. Chem. 2013, 52, 88. 156. Odrizola, L.; Corrales, F. J.; Int. J. Food Nutr. 2015, 66, S31. 157. Barron, R.; Bermingham, K.; Brennan, L.; Gibney, R. L.; Gibney, J. M.; Ryan, F. M.; O'Sullivan, A.; Nutr. Res. 2016, 36, 291. 158. Dessì, A.; Marincola, C. F.; Masili, A.; Gazzolo, D., Vassilios-Fanos, V.; BioMed Res. Int. (2014), doi: 10.1155/2014/981219. DOI: http://dx.doi.org/10.1155/2014/981219 159. Corthésy-Theulaz, I.; den Dunnen, T. J.; Ferré, P.; Geurts, M. W. J.; Müller, M.; van Belzen, N.; van Ommen, B.; Ann. Nutr. Metab. 2005, 49, 355. 160. Zduńczyk, Z.; Pareek, Ch. S.; J. Anim. Feed Sci. 2008, 17, 3. 161. Jones, P.D.; Park, Y.; Ziegler, R.T.; Annu. Rev. Nutr. 2012, 32, 183. 162. Astarita, G.; Langridge, J.; J. Nutrigenet. Nutrigenomics 2013, 6, 179. 163. Brennan, L.; Biochem. Soc. Trans. 2013, 41, 670. 164. Brennan, L.; Prog. Nucl. Magn. Reson. Spectrosc. 2014, 83, 42. 165. Gibbons, H.; O'Gorman, A.; Brennan, L.; Curr. Opin. Lipidol. 2015, 26, 30. 166. McNiven, M.S.E.; German, J. B.; Slupsky, M. C.; J. Nutr. Biochem. 2001, 22, 995. 167. Brennan, L.; Curr. Opin. Biotech. 2017, 44, 35. 168. Ibáñez, C.; Valdés, A.; García-Cañas, V.; Simó, C.; Celebier, M.; Rocamora-Reverte, L.; Gómez-Martínez, Á.; Herrero, M.; Castro-Puyana, M.; Segura-Carretero, A.; Ibáñez, E.; Ferragut, J. A., Cifuentes, A. J.; Chromatogr. A 2012, 1248, 139. 169. O'Sullivan, A.; Gibney, J. M.; Connor, O. A.; Mion, B.; Kaluskar, S.; Cashman, D. K.; Flynn, A.; Shanahan, F.; Brennan, L.; Mol. Nutr. Food Res. 2011, 55, 679. 170. Reaves, M. L.; Rabinowitz, J. D.; Curr. Opin. Biotechnol. 2011, 22, 17. 171. Vernocchi, P.; Vannini, L.; Gottardi, D.; Del Chierico, F.; Serrazanetti, D. I.; Ndagijimana, M.; Guerzoni, M. E.; Front. Cell. Infect. Microbiol. 2012, 2, 156. 172. Ottman, N.; Smidt, H.; De Vos, W. M.; Belzer, C.; Front. Cell. Infect. Microbiol. 2012, 2, 104. 173. Ursell, L. K.; Haiser, H. J.; Van Treuren, W.; Garg, N.; Reddivari, L.; Vanamala, J.; Dorrestein, P. C.; Turnbaugh, P. J.; Knight, R.; Gastroenterology 2014, 146, 1470. 174. Xu, Yong-Jiang.; Wang, C.; Ho, W. E.; Ong, C. N.; Trends Anal. Chem. 2014, 56, 37. 175. Smirnov, K. S.; Maier, T. V.; Walker, A.; Heinzmann, S. S.; Forcisi, A.; Martinez, I.; Walter, J.; Schmitt-Kopplin, P.; Int. J. Med. Microbiol. 2016, 306, 266. 176. Beltran, P. M. J.; Federspiel, J. D.; Sheng, X.; Cristea, I. M.; Mol. Syst. Biol. 2017, 13, 922. 177. Liebeke, M.; Lalk, M.; Int. J. Med. Microbiol. 2014, 304, 222. 178. Abram, F.; Comput. Struct. Biotechnol. J. 2015, 13, 24. 179. Bouchez, T.; Blieux, A. L.; Dequiedt, S.; Domaizon, I.; Dufresne, A.; Ferreira, S.; Godon, J. J.; Hellal, J.; Joulian, C.; Quaiser, A.; Martin-Laurent, F.; Mauffret, A.; Monier, J. M.; Peyret, P.; Schmitt-Koplin, P.; Sibourg, O.; D'oiron, E.; Bispo, A.; Deportes, I.; Grand, C.; Cuny, P.; Maron, P. A.; Ranjard, L.; Environ. Chem. Lett. 2016, 14, 423. 180. Geng, P.; Zhang, L.; Shi, G. Y.; World J. Microbiol. Biotechnol. 2017, 33, 94. 181. Nguyen, Q-T.; Merlo, M. E.; Medema, M. H.; Jankevics, A.; Breitling, R.; Takano, E.; FEBS Lett. 2012, 586, 2177. 182. Fondi, M.; Liò, P.; Microbiol. Res. 2015, 171, 52. 183. Swanepoel, C. C.; Loots, Du Toit.; Dis. Markers 2014, ID 124218. 184. Warner, D. F.; Cold Spring Harb. Perspect. Med. 2015, 5, a021121. 185. Holtfreter, S.; Kolata, J.; Stentzel, S.; Bauerfeind, S.; Schmidt, F.; Sundaramoorthy, N.; Bröker, B. M.; Proteomes 2016, 4, 11. 186. Guo, A. C.; Jewison, T.; Wilson, M.; Liu, Yifeng.; Knox, C.; Djoumbou, Y.; Lo, P.; Mandal, R.; Krishnamurthy, R.; Wishart, D. S.; Nucleic Acids Res. 2013, 41, D625. 187. Stefanovic, E.; Fitzgerald, G.; McAuliffeet, O.; Food Microbiol. 2017, 61, 33. 188. Klemashevich, C.; Wu, C.; Howsmon, D.; Alaniz, R. C.; Lee K.;Jayaraman, A.;. Curr. Opin. Biotechnol. 2014, 26, 85. 189. Mozzi, F.; Ortiz M. E.; Bleckwedel, J.; De Vuyst, L.; Pescuma, M.; Food Res. Int. 2013, 54, 1152. 190. Martinez, P.; Vera, M.; Bobadilla-Fazzini, R. A.; Appl. Microbiol. Biotechnol. 2015, 99, 8337. 191. Fleurbaaij, F.; Van Leeuwen, H. C.; Klychnikov, O. I.; Kuijper, E. J.; Hensbergen, P. J.; Chromatographia. 2015, 78, 379. 192. Xu, Y.; Zhang, Z.; Sun, Z.; Crit Rev Microbiol. 2015, 41, 399. 193. Jansen, R. S.; Rhee, K. Y.; Trends Pharmacol. Sci. 2017, 38, 393. 194. Schulz, C.; Hammerschmidt, S.; Expert Rev. Vaccines 2013, 12, 1061. 195. Wu, C.; Choi, Y. H.; van Wezel, G. P.; J. Ind. Microbiol. Biotechnol. 2016, 43, 299. 196. Sun, J.-L.; Zhang, S.-K.; Chen, J.-Y.; Han, B.-Z.; Can. J. Microbiol. 2012, 58, 709. 197. Eoh, H.; Rhee, K. Y.; Proc. Natl. Acad. Sci. U. S. A. 2013, 110, 6554. 198. https://www.cdc.gov/parasites/about.html, acessada em outubro de 2017. 199. Paget, T.; Haroune, N.; Bagchi, S.; Jarroll, E.; Acta Parasitol. 2013, 58, 127. 200. Cantacessi, C.; Campbell, B. E.; Jex, A. R.; Young, N. D.; Hall, R. S.; Ranganathan, S.; Gasser, R. B.; Parasite Immunol. 2012, 34, 265. 201. Edição Especial: Insights into the metabolomes of parasites, Parasitology 2010, 137, 1283. 202. Creek, D. J.; Barrett, M. P.; Parasitology 2014, 141, 83. 203. Mukherjee, S.; Mukherjee, N.; Gayen, P.; Roy, P.; Babu, S. P.; Curr. Drug Metab. 2016, 17, 937. 204. Kloehn, J.; Blume, M.; Cobbold, S. A.; Saunders, E. C.; Dagley, M. J.; McConville, M. J.; Curr. Opin. Microbiol. 2016, 32, 59. 205. Vincent, I. M.; Barrett, M. P.; J. Biomol. Screen. 2015, 20, 44. 206. Marcelino, I.; de Almeida, A. M.; Ventosa, M.; Pruneau, L.; Meyer, D. F.; Martinez, D.; Lefrançois, T.; Vachiéry, N.; Coelho, A. V.; J. Proteomics 2012, 75, 4232. 207. Keiser, J.; Duthaler, U.; Utzinger, J.; Curr. Opin. Infect. Dis. 2010, 23, 513. 208. Kafsack, B. F. C.; Llinás, M.; Cell Host Microbe 2010, 7, 90. 209. Swann, J.; Jamshidi, N.; Lewis, N. E.; Winzeler, E. A.; Wiley Interdiscip. Rev. Syst. Biol. Med. 2015, 7, 381. 210. Seeber, F.; Steinfelder, S.; F1000Research 2016, 5, 1369. 211. Requena-Méndez, A.; López, M. C.; Angheben, A.; Izquierdo, L.; Ribeiro, I.; Pinazo, M. J.; Gascon, J.; Muñoz, J.; Expert Rev. Anti Infect. Ther. 2013, 11, 957. 212. Sahu, P. K.; Satpathi, S.; Behera, P. K.; Mishra, S. K.; Mohanty, S.; Wassmer, S. C.; Front. Cell. Infect. Microbiol. 2015, 5, 75. 213. Balouz, V.; Agüero, F.; Buscaglia, C. A.; Adv. Parasitol. 2017, 97, 1. 214. Canuto, G. A. B.; da Cruz, P. L. R.; Faccio, A. T.; Klassen, A.; Tavares, M. F. M.; Electrophoresis 2015, 36, 2336. 215. Berg, M.; Mannaert, A.; Vanaerschot, M.; Van der Auwera, G.; Dujardin, J. C.; Parasitol. 2013, 140, 1492. 216. Kip, A. E.; Balasegaram, M.; Beijnen, J. H.; Schellens, J. H. M.; de Vries, P. J.; Dorlo, T. P. C.; Antimicrob. Agents Chemother. 2015, 59, 1. 217. Bringaud, F.; Biran, M.; Millerioux, Y.; Wargnies, M.; Allmann, S.; Mazet, M.; Mol. Microbiol. 2015, 96, 917. 218. Creek, D. J.; Anderson, J.; McConville, M. J.; Barrett, M. P.; Mol. Biochem. Parasitol. 2012, 181, 73. 219. Lakshmanan, V.; Rhee, K. Y.; Daily, J. P.; Mol. Biochem. Parasitol. 2011, 175, 104. 220. Salinas, J. L.; Kissinger, J. C.; Jones, D. P.; Galinski, M. R.; Mem. Inst. Oswaldo Cruz 2014, 109, 589. 221. Krungkrai, S. R.; Krungkrai, J.; Asian Pac. J. Trop. Med. 2016, 9, 525. 222. Jeelani, G.; Nozaki, T.; Curr. Opin. Infect. Dis. 2014, 20, 118. 223. Baumel-Alterzon, S.; Ankri, S.; Curr. Opin. Infect. Dis. 2014, 20, 139. 224. Ryan, U.; Hijjawi, N.; Int. J. Parasitol. 2015, 45, 367. 225. Wang, Y.; Li, J. V.; Saric, J.; Keiser, J.; Wu, J.; Utzinger, J.; Holmes, E.; Adv. Parasitol. 2010, 73, 369. 226. Edison, A. S.; Clendinen, C. S.; Ajredini, R.; Beecher, C.; Ponce, F. V.; Stupp, G. S.; Integr. Comp. Biol. 2015, 55, 478. 227. http://www.who.int/foodborne_trematode_infections/fascioliasis/en/, acesssada em outubro de 2017. 228. Saric, J.; Want, E. J.; Duthaler, U., Lewis, M.; Keiser, J.; Shockcor, J. P.; Ross, A. G.; Nicholson, J. K.; Holmes, E.; Tavares, M. F. M.; Anal. Chem. 2012, 21, 6963. 229. Magalhães Jr, J. T.; Mesquita, P. R. R.; Oliveira, F. S.; Franke, C. R.; Rodrigues, F. de M.; de Andrade, J. B.; Barrouin-Melo, S. M.; Anal. Bioanal. Chem. 2014, 406, 6691. 230. Ernst, M.; Silva, D. B.; Silva, R. R.; Vêncio, R. Z.; Lopes, N. P.; Nat. Prod. Rep. 2014, 31,784. 231. Fernandez, O.; Urrutia, M.; Bernillon, S.; Giauffret, C.; Tardieu, F.; Le Gouis, J.; Langlade, N.; Charcosset, A.; Moing, A.; Gibon, Y.; Metabolomics 2016, 12, 158. 232. Weckwerth, W.; Kahl, G.; The Handbook of Plant Metabolomics, Wiley-VCH Verlag GmbH & Co KGaA: Weinheim, 2013. 233. Allwood, J. W.; Goodacre, R.; Phytochem. Anal. 2010, 21, 33. 234. Hill, C. B.; Czauderna, T.; Klapperstück, M.; Roessner, U.; Schreiber, F.; Front. Bioeng. Biotechnol. 2015, 3, 167. 235. Sumner, L. W.; Lei, Z.; Nikolau, B. J.; Saito, K.; Nat. Prod. Rep. 2015, 32, 212. 236. Hong, J., Yang, L., Zhang, D., Shi, J.; Int. J. Mol. Sci. 2016, 17, 767. 237. Gemperline, E.; Keller, C.; Li, L.; Anal. Chem. 2016, 88, 3422. 238. Jorge, T. F.; Mata, A. T.; António, C.; Phil. Trans. R. Soc. A (2016), doi: 10.1098/rsta.2015.0370. DOI: http://dx.doi.org/10.1098/rsta.2015.0370 239. Boughton, B. A.; Thinagaran, D.; Sarabia, D.; Bacic, A.; Roessner, U.; Phytochem. Rev. 2016, 15, 445. 240. Simó, C.; Ibáñez, C.; Valdés, A.; Cifuentes, A.; García-Cañas, V.; Int. J. Mol. Sci. 2014, 15, 18941. 241. Hoekenga, O. A.; J. Biomol. Tech. 2008, 19, 159. 242. Harrigan, G. G.; Martino-Catt, S.; Glenn, K. C.; Metabolomics 2007, 3, 259. 243. Obata, T.; Fernie, A. R.; Cell. Mol. Life Sci. 2012, 69, 3225. 244. Pérez-Clemente, R. M.; Vives, V.; Zandalinas, S. I.; López-Climent, M. F.; Muñoz, V.; Gómez-Cadenas, A.; BioMed Res. Int. 2013, ID 654120. 245. Teneboim, H.; Brotman, Y.; Trends Plant Sci. 2016, 21, 781. 246. Gomez-Casati, D. F.; Zanor, M. I.; Busi, M. V.; BioMed Res. Int 2013, ID 792527. 247. Manach, C.; Hubert, J.; Llorach, R.; Scalbert, A.; Mol. Nutr. Food Res. 2009, 53, 1303. 248. Wang, L.; Chen, C.; AAPS J. 2013, 15, 941. 249. Nadella, K. D.; Marla, S. S. Kumar, P. A.; OMICS 2012, 16, 149. 250. Tian, H.; Lam, S. M.; Shui, G.; Int. J. Mol. Sci. 2016, 17, 1871. 251. Silva, W. B.; Daloso, D. M.; Fernie, A. R.; Nunes-Nesi, A.; Araújo, W. L.; Plant Sci. 2016, 249, 59. 252. Freund, D. M., Hegeman, A. D.; Curr. Opin. Microbiol. 2017, 43, 41. 253. Dinakar, C.; Bartels, D.; Front. Plant Sci. (2013), doi: 10.3389/fpls.2013.00482. DOI: http://dx.doi.org/10.3389/fpls.2013.00482 254. Fukushima, A.; Kusano, M.; J. Exp. Bot. 2014, 65, 5619. 255. Yang, D.; Du, X.; Yang, Z.; Liang, Z.; Guo, Z.; Liu, Y.; Eng. Life Sci. 2014, 14, 456. 256. Rellán-Álvarez, R.; El-Jendoubi, H.; Wohlgemuth, G.; Abadía, A.; Fiehn, O.; Abadía, J.; Alvarez-Fernández, A.; Front. Plant Sci. 2011, 2, 66. 257. Cao, D.; Lutz, A.; Hill, C. B.; Callahan, D. L.; Roessner, U.; Front. Plant Sci. 2017, 7, 2070. 258. Clarke, C. J.; Haselden, J. N.; Toxicol. Pathol. 2008, 36, 140. 259. Robertson, D. G.; Watkins, P. B.; Reily, M. D.; Toxicol. Sci. 2011, 120, S146. 260. Peters, F. T.; Clin. Biochem. 2011, 44, 54. 261. Oberacher, H.; Arnhard, K. Bioanalysis 2015, 7, 2825. 262. Castillo-Peinado, L. S.; Luque de Castro, M. D.; Talanta 2017, 15, 167. 263. Roux, A.; Lison, D.; Junto, C.; Heilier, J.-F.; Clin. Biochem. 2011, 44, 119. 264. Hazarika, P.; Russel, D. A.; Angew. Chem. Int. 2012, 51, 3524. 265. Dinis-Oliveira, R. J.; Bioanalysis 2014, 6, 3155. 266. Zaitsu, K.; Hayashi, Y.; Kusano, M.; Tsuchihashi, H.; Ishii, A.; Drug Metab. Pharmacokinet. 2016, 31, 21. 267. Dinis-Oliveira, R. J.; Curr. Drug Metab. 2016, 17, 327. 268. Dinis-Oliveira, R. J.; Drug Metab. Rev. 2016, 48, 568. 269. Beger, R. D.; Sun, J.; Schnackenberg, L. K. Toxicol. Appl. Pharmacol. 2010, 413, 154. 270. Weiler, S.; Merz, M.; Kullak-Ublick, G. A.; F1000Prime Rep. 2015, 7, 34. 271. Maurer, H. H.; Clin. Biochem. 2005, 38, 310. 272. Castillo-Peinado, L. S.; Luque de Castro, M. D.; Anal. Chim. Acta 2016, 925, 1. 273. Maeda, H.; Ishikawa, T.; Michiue, T.; Leg. Med. 2014, 16, 61. 274. Donaldson, A. E.; Lamont, I. L.; Aust. J. Forensic Sci. 2014, 46, 8. 275. Zaitsu, K.; Miyawaki, I.; Bando, K.; Horie, H.; Shima, N.; Katagi, M.; Tatsuno, M.; Bamba, T.; Sato, T.; Ishii, A.; Tsuchihashi, H.; Suzuki, K.; Fukusaki, E.; Anal. Bioanal. Chem. 2014, 406, 1339. 276. Kaplan, K. A.; Chiu, V. M.; Lukus, P. A.; Zhang, X.; Siems, W. F.; Schenk, J. O.; Hill Jr., H. H.; Anal. Bioanal. Chem. 2013, 405, 1959. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access





