Revisão
|
|
| Metabolômica microbiana: inovações e aplicações Microbial metabolomics: innovations and applications |
|
Joao Raul BelinatoI,II; Jaqueline Moraes BazioliI,III; Alessandra SussuliniI,II; Fabio AugustoI,II; Taícia Pacheco FillI,*
I. Instituto de Química, Universidade Estadual de Campinas, 13083-970 Campinas - SP, Brasil Recebido em 07/12/2018 *e-mail: taicia@iqm.unicamp.br Over the past few years, metabolomics has been employed in a broad range of applications in different research fields. Microbial metabolomics has been applied in several microbiological areas, such as identification of microorganisms, cell mutations, functional gene approach, identification of unique metabolic pathways and microbial engineering, leading to a better understanding of the global metabolism and metabolic regulation of certain systems. Metabolites produced by microorganisms constitute a large promising group with diverse applications; however, most of these systems are still unknown and underexplored. In this context, the overall analysis of metabolites involved in a biological system can be used to discover potential biomarkers or specific metabolic changes due to a biological phenomenon studied. In this sense, this review aims to understand the main challenges faced by each step of the workflow in the metabolic analysis of microorganisms and how these difficulties have been overcome. A critical analysis of the articles published within the last five years will be accomplished in this paper to understand how these studies have contributed to the microbial metabolomics research. Moreover, major trends observed in this area will be highlighted here in order to guide readers interested in microbial metabolomics in their future researches. INTRODUÇAO Ao longo dos últimos anos, a metabolômica tem desempenhado um papel essencial na pesquisa básica para elucidar os efeitos ambientais,1 entender as funçoes dos genes,2 e a definiçao dos processos celulares3 em busca de uma melhora na qualidade de vida humana. Ela pode ser definida como a análise abrangente (qualitativa e quantitativa) de metabólitos, tendo como objetivo principal reunir a maior quantidade possível de informaçoes metabólicas de um organismo ou sistema biológico.4,5 Na análise metabolômica o principal interesse está nas moléculas de baixa massa molar (< 1000 Da), que suportam funçoes vitais e participam de reaçoes metabólicas, sendo essa informaçao usada para obter uma melhor compreensao das vias bioquímicas.6 Os metabólitos sao classificados como produtos finais dos processos celulares e seus níveis nos sistemas biológicos podem ser considerados como a resposta final às mudanças genéticas, ambientais ou decorrentes de alguma patologia e/ou tratamento.7 Os principais desafios na análise química desses compostos estao relacionados com a complexidade química e a heterogeneidade, requerendo protocolos específicos quanto ao preparo de amostras, decisoes sobre a técnica de análise mais adequada, assim como a estratégia mais eficiente de processamento e interpretaçao dos dados analíticos.8 Quando se tratam de micro-organismos, os estudos no âmbito metabolômico têm contribuído para a descoberta de caminhos metabólicos únicos e interaçoes regulatórias, sendo essas descobertas extremamente úteis para a compreensao do metabolismo global das células.5,9 A metabolômica microbiana tem sido aplicada em vários campos microbiológicos, como identificaçao de micro-organismos,10 mutaçoes celulares,11 pesquisa de genes funcionais12 e identificaçao de vias metabólicas.13 Quando comparada a outros estudos, a principal desvantagem da metabolômica de micro-organismos está na alta complexidade dos metabólitos ainda pouco explorados e, em alguns casos, de difícil identificaçao. Além disso, a separaçao dos metabólitos intracelulares e extracelulares em micro-organismos nao é uma tarefa trivial.5 Apesar do avanço significativo em instrumentaçao analítica e no aumento do número de bancos de dados envolvendo metabólitos microbianos, muitos desafios envolvidos em metabolômica microbiana ainda precisam ser superados. Dessa forma, este artigo de revisao visa compreender os principais desafios enfrentados por cada etapa do fluxo de trabalho, e como essas dificuldades vêm sendo contornadas com base na literatura recente. Além disso, as principais tendências observadas nessa área serao destacadas aqui, a fim de orientar os leitores interessados em metabolômica microbiana em suas futuras pesquisas.
DEFINIÇOES EM METABOLOMICA Usualmente, duas abordagens principais e complementares sao usadas em investigaçoes metabolômicas: perfil metabólico (do inglês, metabolic profiling) e impressao digital metabólica (do inglês, metabolic fingerprinting. Além disso também sao encontrados os termos análise alvo (do inglês, targeted metabolomics) e análise global (do inglês, untargeted metabolomics), onde, a fim de padronizar as discussoes ao longo deste trabalho de revisao, essas serao as abordagens adotadas. A análise alvo concentra-se na análise de um grupo de metabólitos, seja esse grupo relacionado à uma via metabólica específica ou à uma classe de compostos.14,15 Na maioria dos casos, a análise alvo é uma abordagem orientada por hipóteses, e nao uma abordagem para geraçao de hipóteses. Dessa forma, métodos analíticos sao desenvolvidos visando a análise de compostos específicos. Por outro lado, os avanços tecnológicos nos últimos anos têm impulsionado uma expansao na quantificaçao simultânea de um amplo número de substâncias.16,17 A abordagem global está relacionada à análise qualitativa do maior número possível de metabólitos, pertencentes a diferentes classes químicas, encontrados em um determinado sistema biológico alvo e é geralmente utilizada como uma ferramenta de triagem para discriminar amostras de diferentes estados biológicos ou origem. Nesse tipo de abordagem, etapas de preparaçao da amostra podem ser simplificadas ou até mesmo eliminadas.18 A principal vantagem da abordagem global em relaçao à abordagem direcionada é que a primeira permite que novas áreas do metabolismo sejam identificadas.14,15,19 Ainda, análises do tipo exometabolômica (do inglês, footprinting) têm sido empregadas para se referir aos estudos dos metabólitos excretados por uma célula (metaboloma extracelular) ou determinado sistema.15,17
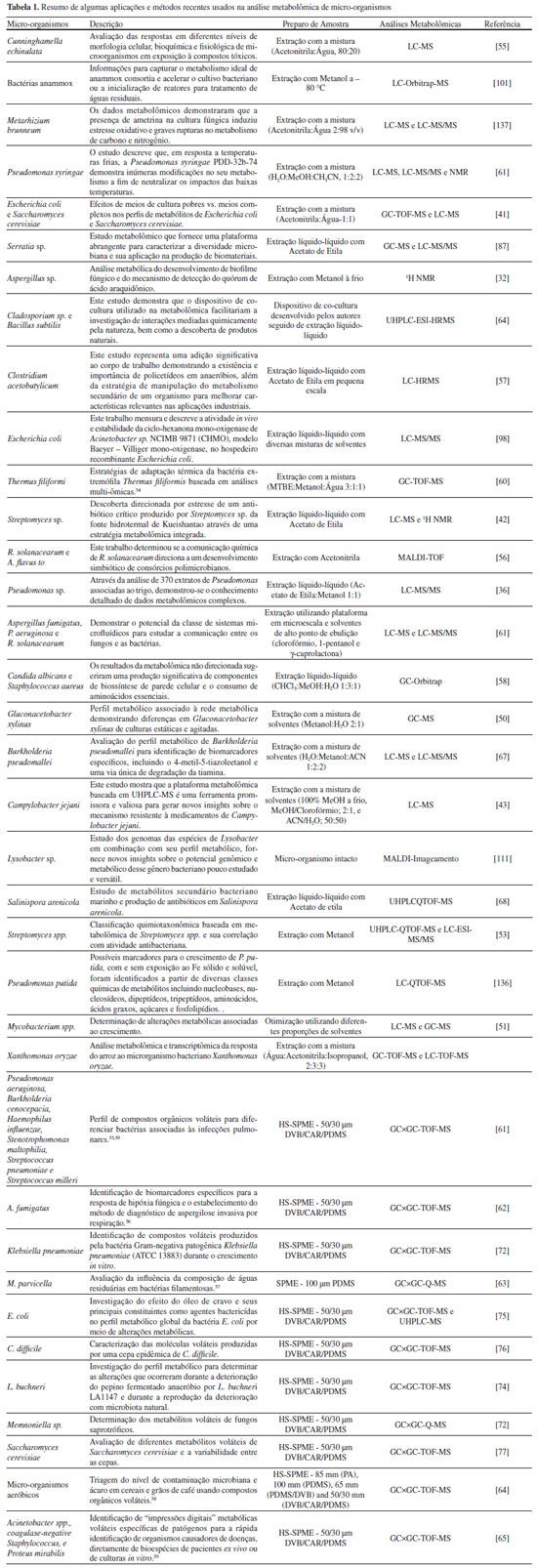
FLUXOGRAMA DE TRABALHO APLICADO A ANALISES DE MICRO-ORGANISMOS A metabolômica microbiana tem se desenvolvido rapidamente nos últimos anos e esse desenvolvimento foi paralelizado e apoiado por avanços importantes em instrumentaçao analítica e tecnologias, em particular métodos cromatográficos e de espectrometria de massas, juntamente com novas e poderosas ferramentas computacionais.20-22 No desenvolvimento desses estudos, sejam eles globais ou alvo-orientados, recomenda-se seguir uma série de etapas, que estao apresentadas resumidamente na Figura 1. Tais abordagens podem ser aplicadas em ambos os tipos de estudo, requerendo particularidades experimentais inerentes ao tipo de aplicaçao.
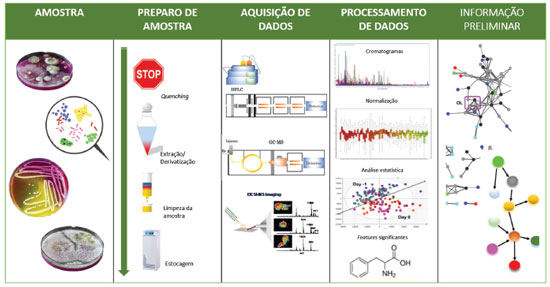 Figura 1. Fluxo de trabalho para abordagem metabolômica nao direcionada. Adaptado de Khoomrung, et al. 2017.23 Copyright 2017, com autorizaçao da revista mediante as normas de open-access
De forma geral, a sequência de trabalho constitui-se da etapa de preparo de amostra, que envolve principalmente o quenching e a extraçao de metabólitos, análise instrumental, processamento e análise dos dados. Para entender melhor cada uma dessas etapas aplicadas às análises de micro-organismos, elas serao descritas individualmente buscando exemplos de aplicaçoes na área microbiológica, além de considerar as devidas particularidades para cada abordagem quando necessário. A definiçao do problema biológico a ser estudado é a primeira etapa no desenvolvimento do estudo, para a determinaçao do tipo de abordagem empregada (global ou alvo). Após essa escolha, as etapas de planejamento experimental e análise química sao delineadas e o experimento metabolômico é conduzido.15,23,24 A partir daí, segue-se para o fluxograma de trabalho propriamente dito. Amostragem e preparo de amostras Os procedimentos de amostragem e preparo de amostras sao componentes vitais de qualquer estudo metabolômico, pois os metabólitos provenientes dos micro-organismos podem sofrer variaçoes metabólicas rápidas durante as etapas de preparo da amostra (geralmente em 1-2 s), por isso, é crucial que essa coleta e manipulaçao da amostra seja rápida.5 Além disso, outro passo importante é a realizaçao do quenching metabólico, que visa a interrupçao rápida da atividade enzimática através do uso de solventes específicos e baixa temperatura, buscando manter a célula ou o organismo intacto em todos os momentos.5 A escolha do quenching adequado depende principalmente do tipo de micro-organismo a ser investigado e, em alguns casos, da adequaçao para os metabólitos de interesse.25 Como a maioria dos estudos de metabólitos microbianos envolve um procedimento de extraçao, em grande parte dos casos, o quenching metabólico é realizado simplesmente como a etapa de congelamento da biomassa em baixas temperaturas e, em seguida, separada do meio extracelular por centrifugaçao, seguindo-se para o passo de lavagem.26-31 Para uma descriçao válida do metaboloma intracelular as etapas de lavagem e quenching metabólico sao cruciais. A lavagem deve efetivamente remover os componentes do meio extracelular e o quenching deve interromper as atividades metabólicas dentro da célula. Portanto, é importante avaliar a eficiência dos solventes tanto na etapa de lavagem quanto na etapa do quenching.32 No procedimento de extraçao, a etapa de lavagem é uma prática comum para a remoçao de metabólitos encontrados no exterior da célula.33 Em um estudo sobre parâmetros que afetam a detecçao e quantificaçao de classes de metabólitos envolvidos na extraçao metabolômica, os autores relataram que, além dos fatores temperatura e pH, a lavagem é uma prática comum e de extrema importância na remoçao de metabólitos extracelulares, melhorando nesses casos a relaçao sinal-ruído.34 Os trabalhos realizados até o presente momento têm descrito muito pouco sobre as etapas de quenching e lavagem, sendo que muitas vezes nao há uma otimizaçao clara desses procedimentos ou mesmo testes de controle para avaliar a perda de metabólitos pela ausência ou realizaçao nao significativa desses procedimentos. Entretanto, a avaliaçao e otimizaçao dessas etapas poderiam aumentar a confiabilidade dos estudos metabolômicos obtidos. Em alguns estudos, esse procedimento é realizado em combinaçao com a etapa de extraçao, onde utilizam-se solventes ou misturas de solventes à baixa temperatura no processo de extraçao.35-38 Além disso, mediante a importância desse procedimento, em alguns casos também sao realizados testes com diferentes tipos de quenching a fim de avaliar o melhor método a ser utilizado. Um estudo de perfil metabólico utilizando a levedura Pichia pastoris empregou diferentes metodologias de quenching, demonstrando, assim, duas abordagens para metabólitos extracelulares e intracelulares.39 Na avaliaçao dos metabólitos extracelulares, as amostras foram rapidamente centrifugadas e decantadas antes de serem congeladas em gelo seco. Com relaçao à avaliaçao dos metabólitos intracelulares, foi utilizado um procedimento de quenching total (células + meio de cultura) com 60% de metanol aquoso e bicarbonato de amônio para interromper o metabolismo.39 A mistura metanol-água à baixa temperatura (aproximadamente -40 °C) é um dos procedimentos mais relatados na literatura,40 inclusive nos estudos lipidômicos,41,42 sendo aplicado também em estudos metabólicos do fungo Fusarium oxysporum,43 e da bactéria Gluconacetobacter xylinus,44 dentre outros.45,46 A mistura tem se apresentado extremamente eficiente nesses estudos alcançando ótimos resultados, mas ainda vale ressaltar que os experimentos simples de otimizaçoes podem viabilizar uma escolha ainda mais confiável do método empregado. Após o quenching metabólico, frequentemente é realizada a separaçao dos metabólitos extracelulares dos intracelulares, dependendo da abordagem empregada.31 Para identificar e quantificar metabólitos intracelulares, é necessário extrair esses compostos do compartimento intracelular protegido pela membrana celular. Isso é alcançado usando solventes de extraçao (orgânicos, inorgânicos nao aquosos ou uma mistura dos dois), que tornam a membrana celular porosa ou permeável, permitindo a penetraçao dos solventes no meio intracelular e uma maior recuperaçao desses metabólitos intracelulares.47 Por outro lado, os metabólitos presentes no ambiente extracelular sao originalmente parte da composiçao do meio de cultura, compostos secretados pelas células ou produto das lises celulares e, por isso, sao mais facilmente extraídos.25 A extraçao dos metabólitos é uma etapa de extrema importância na metabolômica microbiana, embora em alguns casos acabe sendo negligenciada.25 Em geral, a etapa de extraçao é dependente da estratégia do estudo metabolômico empregado e do tipo de análise.48 Para análises nao direcionadas, por exemplo, o objetivo é extrair o maior número de metabólitos de muitas classes químicas de forma quantitativa e nao tendenciosa, com perdas mínimas de metabólitos. Portanto, as amostras sao analisadas priorizando o preparo de amostras mínimo, como precipitaçao de proteínas, remoçao de sais, filtraçao e diluiçao.15,48 A avaliaçao global dos metabólitos extracelulares geralmente envolve somente etapas de extraçao do meio extracelular e diluiçoes. Para os metabólitos intracelulares, usualmente sao empregados procedimentos preliminares que possibilitem a lise celular e em seguida o uso de extraçoes líquido-líquido a fim de minimizar a distinçao de classes químicas para a análise global49,50 e, em alguns casos, utilizam-se etapas de agitaçao e centrifugaçao.35,37,51-54 Em geral, estudos recentes têm utilizado esse tipo de extraçao variando os solventes empregados, dependendo principalmente do micro-organismo em estudo. A Tabela 1 sumariza alguns dos principais estudos encontrados em análises metabolômicas de micro-organismos, destacando os principais solventes e proporçoes utilizadas, bem como outros sistemas de extraçao.
No que se refere aos sistemas extratores modernos, plataformas microfluídicas foram empregados em análises metabolômicas de micro-organismos a fim de miniaturizar as etapas de extraçao. O sistema desenvolvido pelos autores60 utiliza um sistema bifásico aberto em que o solvente orgânico é guiado sobre um ambiente de cultura microbiana, permitindo a extraçao integrada e passiva de metabólitos. O dispositivo foi aplicado em análises de metabólitos provenientes de diversos fungos e bactérias, possibilitando também a avaliaçao de co-culturas. Esse tipo de abordagem para extraçao de metabólitos miniaturizada parece ser uma tendência na área, uma vez que facilita a exploraçao de microambientes de cultura e possibilita a utilizaçao de solventes orgânicos incomuns para o isolamento de metabólitos o que vem a ser uma grande vantagem quando comparado aos métodos clássicos de extraçao líquido-líquido.60 Nas análises alvo-orientadas, a extraçao deve favorecer a recuperaçao quantitativa das espécies químicas de interesse e a remoçao dos interferentes, assim, classes específicas de compostos de interesse sao selecionadas, por isso, é extremamente difícil adotar uma metodologia universal. Dessa forma, os procedimentos empregados nas extraçoes em micro-organismos envolvem diversas etapas como extraçoes sequenciais com diferentes solventes, centrifugaçao, evaporaçao, filtraçao e até mesmo etapas utilizando cartuchos de extraçao em fase sólida.26,45,63-66 Para tanto, procedimentos de extraçao mais complexos sao empregados, envolvendo principalmente etapas de extraçao líquido-líquido e extraçao em fase sólida (SPE, do inglês solid phase extraction). Estudos recentes comprovaram a eficácia na utilizaçao da mistura ternária (H2O:MeOH:CH3CN) na extraçao de metabólitos de Pseudomonas syringae com o objetivo de avaliar sua resposta a adaptaçao de ambientes em diferentes temperaturas.61,62 Ao se tratar de metabólitos voláteis, a principal técnica utilizada nos estudos metabolômicos é a microextraçao em fase sólida (SPME, do inglês solid phase microextraction). Uma grande vantagem da utilizaçao da técnica de SPME nesses estudos é a possibilidade da realizaçao da análise in vivo.68 Estudos recentes demonstram o poder da ferramenta de SPME ao extrair um grande número de compostos orgânicos voláteis.69 Esses estudos têm sido aplicados nos mais diferentes gêneros de micro-organismos, tais como Lactobacillus buchneri,10 E. coli,71 Clostridium difficile,72 S. cerevisae,73 dentre outros. Utilizando a técnica de SPME, Mousavi e colaboradores avaliaram o perfil metabólico de E. coli, incluindo 500 metabólitos identificados por LC-MS e 789 componentes detectados por GCxGC-TOF-MS. Dos metabólitos detectados pela plataforma de GCxGC-TOF-MS, 125 foram identificados como metabólitos desregulados, revelando mudanças no metaboloma provocadas pela atividade antibacteriana do óleo de cravo e, em particular, o seu principal constituinte, o eugenol. A avaliaçao dos metabólitos voláteis por SPME foi extremamente eficaz e demonstrou que a variaçao nos metabólitos frente ao tratamento empregado foi facilmente detectada.71 A padronizaçao de protocolos analíticos e métodos de extraçao é um dos tópicos mais discutidos entre os membros da comunidade interessada nos estudos metabolômicos. Tais discussoes convergem quanto a inexistência de uma técnica analítica ou método de extraçao universal para determinaçao abrangente dos metabólitos de um sistema biológico. Dessa forma, é importante compreender que a combinaçao de diferentes técnicas de análise e extraçao poderao ampliar o número de metabólitos extraídos, mas a padronizaçao desses métodos se faz necessária, a fim de obter perfis de metabólitos intracelulares globais e mais precisos e, consequentemente, obter uma interpretaçao biológica mais precisa dos dados metabolômicos.15,25 Além disso, a utilizaçao de técnicas miniaturizadas também tem sido um grande atrativo no preparo de amostras em análises metabolômicas e o crescente emprego dessas ferramentas também proporcionou a aplicaçao dessas em micro-organismos.74,75 Outros tipos de extraçao e procedimentos mais detalhados podem ser encontrados em artigos de revisao que tratam especificamente dessa etapa.25,76 Ferramentas Analíticas Aplicadas à Análises metabolômicas Apesar dos grandes avanços em instrumentaçao analítica ao longo dos últimos anos, um desafio ainda enfrentado pela metabolômica microbiana é a cobertura de todos os compostos por uma única técnica analítica.22 O perfil global de metabólitos requer técnicas analíticas capazes de medir uma variedade de classes químicas de moléculas em amostras biológicas, potencialmente existentes em uma ampla gama de concentraçoes.5 Por conseguinte, a análise eficaz é atingida através da combinaçao de diferentes ferramentas analíticas para obter a maior informaçao possível sobre o perfil de metabólitos de uma populaçao de células ou organismo.22 Dentro de metabolômica de micro-organismos as principais técnicas analíticas empregadas sao a NMR, GC-MS (do inglês, Gas Chromatography-Mass Spectrometry) e LC-MS (do inglês, Liquid Chromatography-Mass Spectrometry), sendo as principais aplicaçoes dessas técnicas destacadas a seguir. GC-MS A GC-MS é uma técnica de análise muito utilizada em estudos metabolômicos, em que várias centenas de compostos podem ser analisados simultaneamente, incluindo ácidos orgânicos, aminoácidos, açúcares, álcoois, aminas aromáticas, ácidos graxos, dentre outros, onde em alguns casos a derivatizaçao é necessária.5,15 Uma grande vantagem para a aplicaçao em estudos metabolômicos é a disponibilidade de bibliotecas espectrais em GC-MS. Essas bibliotecas oferecem ótimas possibilidades para a identificaçao de biomarcadores desconhecidos.77 Além disso, a análise de compostos voláteis por headspace vem sendo bastante empregada para acessar a porçao volátil do metaboloma, possibilitando até mesmo estudos in vivo.68,78 Contudo, o grande limitante na análise metabolômica utilizando GC-MS é a volatilidade dos compostos, pois grande parte dos metabólitos que compoem o metaboloma requerem derivatizaçao para que se tornem voláteis nas temperaturas de trabalho do cromatógrafo a gás.15 As variaçoes nos experimentos de derivatizaçao e formaçao de compostos indesejados sao duas questoes fundamentais que podem influenciar diretamente a análise. Dessa forma, métodos de derivatizaçao reprodutíveis e eficientes sao elementos essenciais para análises utilizando GC-MS.77 As reaçoes de derivatizaçao permitiram análises de ácidos graxos e outros lipídios em micro-organismos.79 Análises de perfis de fosfolípidios de Penicillium chrysogenum foram empregadas para avaliar essa variaçao em diferentes condiçoes de cultivo do micro-organismo.80 A GC-MS também foi empregada na análise de ácidos graxos submetidos à derivatizaçao81,82 tanto para avaliaçoes de perfil lipídico82,83 quanto para identificaçoes específicas.84-87 O estudo da produçao metabólica em diferentes meios de cultura foi realizado para os micro-organismos Escherichia coli e Saccharomyces cerevisiae onde GC-TOF-MS (do inglês, Gas Chromatography - Time of Flight Mass Spectrometry) foi utilizada na identificaçao e quantificaçao dos metabólitos intracelulares.35 Além da utilizaçao da abordagem alvo-orientada 38 em GC-MS, as abordagens globais também têm sido empregadas em análises de diversos micro--organismos.43,44,46,52 Um estudo realizado com as cepas de Candida albicans e Staphylococcus aureus buscou avaliar o perfil global dos metabólitos intracelulares e extracelulares dos micro-organismos utilizando para tal análise um GC-Orbitrap-MS.52 Tanto o GC-TOF-MS quanto o GC-Orbitrap-MS sao sistemas que possuem analisadores de massas de alta resoluçao, permitindo assim a obtençao de massa exata e possibilidade de identificaçao ainda mais confiável para esses metabólitos, o que vem a ser um grande atrativo. Como já explicitado, a utilizaçao de uma única plataforma para investigaçao metabolômica acaba limitando o número de metabólitos alcançados e a distinçao entre classes químicas. Por isso, tem se tornado cada vez mais comum observar estudos nos quais diferentes abordagens sao empregadas, principalmente técnicas cromatográficas como GC-MS e LC-MS.45,88,89 A avaliaçao do exometaboloma de Saccharomyces cerevisiae utilizando ambas as plataformas permitiram uma investigaçao ampla do perfil metabólico no processo fermentativo da levedura correlacionando os metabólitos identificados com as diferentes vias bioquímicas das células do micro-organismo,88 observando dessa forma, mudanças metabólicas e fisiológicas em resposta à hipóxia. Diante da alta complexidade da amostra e do grande número de metabólitos presentes em micro-organismos, a cromatografia gasosa bidimensional abrangente (GCxGC) vem sendo utilizada como alternativa à GC-MS tradicional. As vantagens da abordagem por GCxGC sao as superiores sensibilidade analítica e capacidade de pico.90,91 Estudos avaliando o metaboloma volátil de Klebsiella pneumoniae69,92 e outros micro-organismos68,70,93 têm expandido horizontes na exploraçao de compostos voláteis provenientes de micro-organismos antes nao observados nas técnicas unidimensionais. Diversos estudos envolvendo a GCxGC têm sido empregados tanto na análise alvo quanto nao alvo. Na Tabela 1 é possível encontrar diversas aplicaçoes utilizando essa técnica, principalmente quando associada à SPME. LC-MS A cromatografia líquida acoplada à espectrometria de massas (LC-MS) tornou-se uma importante ferramenta analítica em metabolômica, e também foi aplicada em estudos sobre muitos sistemas biológicos.22,94 A LC-MS é sem dúvida a plataforma mais adequada para a análise de compostos polares e nao polares pouco voláteis, nos quais nao é viável uma reaçao de derivatizaçao.5 É uma técnica analítica que apresenta alta robustez, sensibilidade e seletividade.15 Diversas opçoes sao disponíveis para fontes de ionizaçao e analisadores de massas em sistemas LC-MS possibilitando a realizaçao tanto de análises alvo como abordagens globais, o que faz da LC-MS um grande atrativo para os estudos microbiológicos. A LC-MS permite portanto, a possibilidade de realizar tanto análises qualitativas quanto análises quantitativas, fornecendo informaçoes estruturais a partir de dados MS/MS, e quantificaçoes simultaneamente sem a necessidade de altas resoluçoes cromatográficas. Nas duas últimas décadas, várias plataformas LC-MS foram utilizadas com sucesso na análise metabolômica, sendo aplicadas à diversas matrizes micro-biológicas.95,96 O modo de monitoramento de reaçoes selecionadas (SRM, do inglês Selected Reaction Monitoring) em LC-MS/MS usando um analisador triplo quadrupolo também tem sido aplicada principalmente na identificaçao e quantificaçao de lipídios e outros metabólitos.84,97,98 As aplicaçoes para LC-MS dentro do contexto da análise metabolômica de micro-organismos sao bastante amplas, e a grande quantidade de informaçao gerada acaba tornando o uso de ferramentas de análises de dados essenciais nesses estudos.36,64,99,100 Uma abordagem recente que também tem ganhado espaço dentro das análises metabolômicas é a associaçao da espectrometria de massas com a técnica de mobilidade iônica UHPLC-TWIM-MS (do inglês, Ultra High Performance Liquid Chromatography combined with Traveling Wave Ion Mobility Mass Spectrometry). Essa abordagem tem sido empregada com sucesso para aumentar a capacidade de sinais e possibilitar a separaçao de espécies isoméricas.53,101,102 As produçoes de metabólitos e lipídios foram monitoradas em linhagens intactas e geneticamente modificadas de C. reinhardtii, com comprometimento da via de amido. Os lipídios, tais como triacilgliceróis (TAG) e diacilgliceril-N, N, N-trimetil-homosserina (DGTS), foram avaliados ao longo do tempo sob diferentes condiçoes de luz. Mais de 200 metabólitos, tais como arginina, cisteína, serina, palmitato, clorofila a, clorofila b, etc., foram detectados,101 demonstrando que a técnica empregada foi extremamente eficiente na identificaçao de metabólitos de diferentes características químicas. Uma aplicaçao da LC-MS recente e bastante interessante foi a utilizaçao de metabólitos secundários de Streptomyces (8 espécies, 14 cepas) para classificaçao quimiotaxonômica. Assim, esse estudo demonstrou que esse tipo de classificaçao baseada em metabólitos é uma ferramenta eficaz para distinguir Streptomyces spp. e para determinar os metabólitos específicos da espécie.47 Além das análises exploratórias, estudos específicos para a descoberta de novas classes de compostos, como antibióticos produzidos a partir de micro-organismos também têm sido desenvolvidos, sendo que a LC-MS é amplamente empregada na caracterizaçao desses novos compostos.103-105 É evidente que a LC-MS é a plataforma mais empregada em estudos metabolômicos em geral, contudo, devido à alta complexidade dessas amostras a cromatografia liquida bidimensional (LCxLC) também têm ganhado destaque nos estudos metabolômicos. Apesar de ainda ser pouco popular, a LCxLC compreende vantagens similares à GCxGC e possibilita ampliar ainda mais a quantidade de metabólitos investigados simultaneamente, buscando conhecer de forma detalhada esses sistemas e suas possíveis variaçoes. Imageamento por MS Dentre outras técnicas baseadas em MS, a ionizaçao e dessorçao à laser assistida por matriz (MALDI, do inglês matrix-assisted laser desorption/ionization) também tem sido usada nos estudos metabólicos de diferentes cepas de micro-organismos.60 Além das diversas aplicaçoes em estudos metabólicos gerais,41,87,106,107 vale destacar a utilizaçao de MALDI na análise de impressoes digitais lipídicas de vírus intactos108 e também em aplicaçoes de imageamento.20,109 O imageamento por espectrometria de massas (MSI, do inglês Mass Spectrometry Imaging) permite o estudo da distribuiçao espacial de pequenas moléculas em amostras biológicas.110 Essa metodologia foi introduzida em 1997 por Caprioli e colaboradores, e aplicada principalmente na superfície de tecidos biológicos intactos para criar imagens 2D com base na distribuiçao espacial de peptídeos e proteínas.111 Essa técnica tem sido expandida e utilizada no estudo espacial de metabólitos e lipídios em diferentes matrizes biológicas, análise de produtos naturais microbianos,113 interaçoes microbianas entre culturas mistas114 e caracterizaçao metabólica de micro-organismos.115,116 A técnica de imageamento por MALDI-TOF por exemplo, foi utilizada a fim de caracterizar a distribuiçao espacial de metabólitos produzidos por cianobactérias de diferentes complexidades. Uma série de metabólitos produzidos pelas cianobactérias Lyngbya majuscula, Oscilatoria nigro-viridis, Lyngbya bouillonii e uma espécie Phormidium, mesmo quando estavam presentes como misturas, foram identificados. Além da jamaicamida B, um produto natural conhecido da cianobactéria Lyngbya majuscula JHB, um grande número de íons desconhecidos localizados nas diferentes cianobactérias foi detectado, possibilitando a exploraçao de novos metabólitos nos organismos investigados. Em uma abordagem recente sobre o perfil metabólico de diferentes cepas de Lysobacter, relatou-se a aplicaçao de MALDI-MSI em complemento às análises do genoma in silico frente ao estudo das interaçoes entre Lysobacter e Rhizoctonia solani, sendo esse último uma espécie de patógeno fúngico economicamente importante. Através da técnica de imageamento, foi possível identificar os compostos bioativos bem como os padroes de distribuiçao espacial desses compostos na interaçao entre os dois micro-organismos.96 Dessa forma, é possível perceber que a técnica de MALDI-MSI tem sido empregada na metabolômica microbiana, e apesar de algumas limitaçoes referentes às condiçoes experimentais imposta pela técnica, essa ainda tem demonstrado excelentes resultados principalmente na identificaçao de micro-organismos e também em promover diversidade química. O avanço das técnicas de imageamento por espectrometria de massas de alta resoluçao também viabilizou a possibilidade de informaçoes detalhadas e elementares em sistemas microbianos possibilitando o monitoramento nao invasivo e in situ dessas comunidades.117,118 As técnicas de ionizaçao ambiente em imageamento por espectrometria de massas têm sido uma escolha popular para apresentar a distribuiçao espacial de moléculas in situ devido à pouca ou nenhuma preparaçao de amostra e sem interferência de matriz, onde a ionizaçao de dessorçao por electrospray (DESI, do inglês Desorption Electrospray Ionization) é o sistema mais utilizado.119,120 Vale destacar o desenvolvimento e adaptaçao de outras fontes de ionizaçao que permitam análises in situ como a ionizaçao por eletrospray com ablaçao a laser (LAESI, do inglês Laser Ablation Electrospray Ionization), análise direta em tempo real (DART, do inglês Direct Analysis in Real Time), além dos grandes avanços tecnológicos que permitiram a realizaçao de imageamento por MS, de modo que fosse possível visualizar metabólitos dentro de resoluçoes espaciais < 1 µm em pressoes atmosféricas e com alta precisao e resoluçao em massa.22 Também é importante ressaltar que outras técnicas emergentes baseadas em espectrometria de massas ainda pouco difundidas, tais como espectrometria de massas de ionizaçao evaporativa rápida (REIMS, do inglês Rapid Evaporative Ionization Mass Spectrometry) e espectrometria de massas por pulverizaçao em papel (PS-MS, do inglês Paper Spray-Mass Spectrometry) têm demonstrado eficiência nos estudos metabolômicos, a fim de facilitar o preparo de amostra e minimizar os tempos de análise, como pode ser visto na Figura 2. Essas técnicas têm sido aplicadas tanto na identificaçao de micro-organismos quanto nos estudos metabólicos individuais ou de interaçao microbiana.121
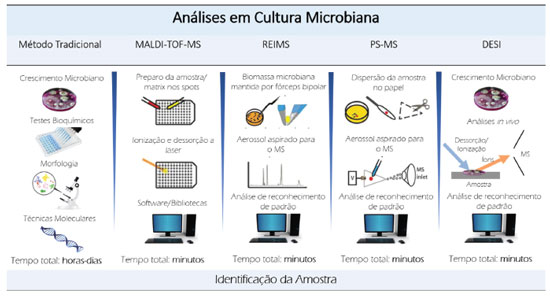 Figura 2. Fluxos de trabalho para identificaçao microbiana. Método tradicional; Ionizaçao e dessorçao a laser assistido por matriz (MALDI) espectrometria de massas (MS) por tempo de voo (TOF); Espectrometria de massas com ionizaçao evaporativa rápida (REIMS); Espectrometria de massas por pulverizaçao em papel (PS-MS); Ionizaçao de Dessorçao por Electrospray (DESI). Extraído de Luzzatto-Knaan et al. 2015.121 Copyright 2015, com autorizaçao da revista Analyst
O futuro da metabolômica microbiana certamente está relacionado com os avanços em técnicas analíticas que permitam a utilizaçao de amostras de biomassa muito pequenas. Nesse sentido, a espectrometria de massas parece ser a tecnologia com o melhor potencial para alcançar esses objetivos.22 Mediante os trabalhos avaliados, é possível observar que a utilizaçao de múltiplas plataformas é extremamente interessante nos estudos metabolômicos, possibilitando uma ampla faixa de investigaçao, levando a resultados confiáveis e com uma grande quantidade de informaçao. Processamento e análise de dados Os dados gerados a partir das análises metabolômicas sao abundantes e de alta complexidade e, por isso, ferramentas adequadas ao tratamento de dados devem ser empregadas a fim de evitar erros de interpretaçao dos resultados e manter a integridade das variaçoes biológicas inspecionadas.15 Assim, um dos maiores desafios da metabolômica envolve o pré-processamento e a análise de dados, sendo necessárias amplas opçoes de softwares e estratégias para transformar dados brutos em resultados biológicos úteis.122 As ferramentas necessárias nesse processamento incluem estratégias para processar arquivos tanto de GC-MS e LC-MS quanto de NMR, onde uma série de ferramentas de análise estatística, bancos de dados de metabólitos e, finalmente, softwares de bioinformática sao utilizados.15 Pré-processamento O pré-processamento de dados é um passo intermediário entre a aquisiçao direta e a análise dos dados.5 A maioria dos softwares de pré-processamento compartilham as funçoes gerais de deconvoluçao de sinais analíticos, filtragem de ruído, detecçao e alinhamento de picos cromatográficos, correçao da linha de base e preenchimento de lacunas (gap filling), embora as capacidades, vantagens e limitaçoes variem drasticamente dentre a grande possibilidade de softwares atualmente disponível.15,122,123 As etapas gerais de processamento podem ser resumidas na Figura 3, onde as principais etapas sao descritas, podendo haver variaçoes mediante à aplicaçao empregada e ao objetivo da análise.
 Figura 3. Etapas de processamento geral da análise de dados metabolômicos, desde a aquisiçao de dados até a análise estatística. Adaptado de A. Sussulini, ed., Metabolomics: From Fundamentals to Clinical Applications, Springer International Publishing, Cham, 2017.124 Copyright 2015, com autorizaçao da Springer
O principal objetivo da filtragem de ruído é separar o sinal proveniente de um composto na amostra biológica de um sinal de fundo proveniente de interferência química ou instrumental, facilitando a etapa de detecçao de picos125,126 A detecçao de pico/deconvoluçao compreende a identificaçao da forma correta de um sinal que potencialmente é de um composto, ou seja, detecta cada íon medido em uma amostra e atribui a um sinal cromatográfico (par m/z-RT (do inglês, retention time).123,124 O objetivo da detecçao e deconvoluçao de pico é identificar e quantificar os sinais correspondentes às moléculas nas amostras; assim, um método de detecçao de pico eficiente deve identificar os sinais verdadeiros e evitar falsos positivos. Além disso, a detecçao de picos e a deconvoluçao reduzem a complexidade dos dados e tornam a posterior análise viável.126 Outra etapa de extrema importância no pré-processamento de dados cromatográficos é o alinhamento dos picos entre as amostras. De forma resumida, o algoritmo de alinhamento visa corrigir oscilaçoes aleatórias de tempo de retençao de um mesmo pico entre diferentes amostras.124 Já o algoritmo de preenchimento de lacunas é utilizado para recuperar os sinais que podem ter sido eliminados nas etapas anteriores de pré-processamento a partir dos dados brutos.126 A normalizaçao é o processo que visa corrigir a variaçao sistemática e escalar os dados para que diferentes amostras em um estudo possam ser comparadas entre si, sendo assim, é aplicado dividindo cada linha de tabela de dados por um fator de normalizaçao.4 Duas estratégias principais sao comumente usadas para remover o viés sistemático indesejado nas medidas. A primeira é o uso de padroes internos, no qual a seleçao desses padroes pode ser baseada em regioes específicas de RT ou m/z, mas RT e m/z, contudo, nem sempre sao descritivos de todas as propriedades matriciais e químicas levando a obscurecer a variaçao de dados.126 Outra estratégia sofisticada para a normalizaçao é o uso de amostras de controle de qualidade (QC, do inglês Quality Control) em cada procedimento de aquisiçao de dados a fim de visualizar a variabilidade global de um sistema de mediçao. As amostras de QC referem-se à mistura de volumes iguais de todas as amostras envolvidas no estudo (todos os grupos avaliados).15 Assim, as amostras estudadas sao comparadas com QCs para avaliar sua reprodutibilidade, desempenho e estabilidade instrumental.4,15 As funçoes descritas anteriormente na etapa de pré-processamento sao desempenhadas automaticamente com o auxílio de diferentes softwares e plataformas online e, por isso, um grande volume de novas ferramentas vem sendo desenvolvido nos últimos anos.5,125 Contudo, é importante salientar que a etapa de pré-processamento de dados automático e eficaz continua a ser uma tarefa difícil, especialmente para a detecçao, alinhamento e deconvoluçao de picos de baixa intensidade.4 Algumas das principais plataformas/softwares empregadas no pré-processamento de dados metabolômicos foram compiladas na Tabela 2, sendo em seguida apresentadas algumas aplicaçoes para micro-organismos quando existentes. A maioria dessas ferramentas apresentadas está disponível gratuitamente e podem ser empregadas para dados de LC-MS e GC-MS.

Outras listas de softwares disponíveis podem ser encontradas em artigos de revisao focados em pré-processamento e análise de dados.4,125,126 Além disso, muitos fabricantes de instrumentos também oferecem seu próprio software, por exemplo, MarkerLynx (Waters), MassProfiler (Agilent), MarkerView (SCIEX) e SIEVE (Thermo Fisher Scientific). A plataforma MultAlign foi utilizada no pré-processamento de dados de LC-MS para análises metabolômicas de Yarrowia lipolytica antes da etapa de análise de dados.42 Em alguns casos, as etapas de pré-processamento sao integradas ao software do equipamento no qual a análise foi realizada, nao necessitando de outras plataformas externas.107,131 Além disso, a plataforma XCMS-online, que é uma ferramenta bastante difundida nos estudos metabolômicos, foi utilizada no tratamento de dados para Pseudomonas syringae,61 Streptomyces sp.,36 Candida albicans and Staphylococcus,52 e no tratamento de dados referente a um estudo de co-culturas de bactérias e fungos.60 Análise de dados As técnicas de análise estatística univariada e multivariada sao usadas para extrair informaçoes relevantes sobre o problema estudado. Além disso, ferramentas estatísticas como o teste t, análise de variância (ANOVA),66 análise de componentes principais (PCA, do inglês Principal Component Analysis) ou a análise discriminante por mínimos quadrados parciais (PLS-DA, do inglês, Partial Least Squares-Discriminant Analysis) sao geralmente componentes essenciais em estudos de metabolômica. A adequaçao de um teste estatístico para melhor detectar a expressao diferencial nos estudos de interesse é determinada pela distribuiçao de dados e problema biológico a ser resolvido ou investigado.123 Antes da análise de dados propriamente dita, em alguns casos sao utilizadas etapas de pré-tratamento, onde os dados sao submetidos a diferentes ferramentas estatísticas como centralizaçao, dimensionamento e transformaçao de dados que sao utilizadas para minimizar os erros experimentais, maximizando as variaçoes biológicas relevantes.124 A quantidade de ferramentas estatísticas que tem sido aplicada em análises metabolômicas é bastante vasta, por isso esta revisao foi direcionada às principais ferramentas aplicadas nas análises de micro-organismos, concentrando-se nas ferramentas multivariadas. Nas análises multivariadas, a classificaçao e discriminaçao de entidades ou metabólitos responsáveis por diferenciar os grupos de amostras sao realizadas através da avaliaçao do conjunto da matriz de dados extraídos na etapa de pré-processamento.4,15 Os métodos nao supervisionados podem revelar os fatores mais importantes de variabilidade que caracterizam o conjunto de dados, que normalmente sao distintos do fator especificamente investigado. Geralmente sao usados para explorar a estrutura geral de um conjunto de dados, encontrar tendências e agrupamentos dentro do conjunto, contribuindo com uma visao imparcial dos dados.4,123 Vários métodos nao supervisionados têm sido aplicados, dentre os quais a PCA e a análise de agrupamento hierárquico (HCA, do inglês Hierarchical Cluster Analysis) sao os exemplos mais utilizados na metabolômica.4 Além disso, a análise de componentes principais multimodo (MPCA, do inglês Multilinear Principal Component Analysis) também tem sido utilizada nos estudos metabolômicos de micro-organismos.68,71,132 Já os métodos supervisionados utilizam estruturas de dados conhecidas para treinar padroes com o intuito de prever a classe de novos dados. Esses métodos podem ser classificados como métodos lineares, como PLS-DA, análise discriminante linear (LDA, do inglês Linear Discriminant Analysis), projeçoes ortogonais para estruturas latentes (OPLS-DA) e métodos nao-lineares.4 O método supervisionado mais utilizado para classificaçao em quimiometria é a PLS-DA, que é uma combinaçao de regressao multivariada por PLS com a análise discriminante.133 A vantagem da PLS-DA é a sua capacidade de lidar com dados altamente colineares. Além disso, a PLS-DA pode fornecer informaçoes excelentes sobre a causa da discriminaçao, verificando o comportamento das variáveis, sendo também uma ferramenta útil na descoberta de biomarcadores.5 Uma interessante modificaçao de PLS-DA é a OPLS-DA (do inglês Orthogonal Partial Least Squares) na qual as variaçoes sistemáticas na matriz de dados X podem ser divididas em duas partes através da técnica de correçao de sinal ortogonal (OSC, do inglês Orthogonal Signal Correction): uma parte exibe os padroes multivariados correlacionados à resposta experimental, enquanto os padroes ortogonais sao representados na segunda parte. A distinçao entre as diferentes fontes de variaçao nos dados, por exemplo correlacionada versus ortogonal, impoe uma importante vantagem da OPLS-DA na visualizaçao e interpretaçao dos dados em relaçao à PLS-DA, e tem sido amplamente aplicada na modelagem e descoberta de importantes compostos na análise metabolômica de micro-organismos.26,37,44,64,67 A PCA foi empregada para avaliar a diferença no perfil lipídico de diferentes espécies de Cryptococcus sp, nos quais os principais compostos responsáveis pela diferenciaçao foram identificados.84 Tanto o pré-processamento quanto a aplicaçao de ferramentas multi-variadas foram utilizadas para a classificaçao de amostras de Bacillus sp. e Brevibacillus sp. utilizando o perfil lipídico.41 Outras aplicaçoes de PCA42,82,134 e HCA80 para a análise exploratória e diferenciaçao de micro-organismos sao encontradas na literatura utilizando o perfil lipídico como diferenciador das cepas. A PLS-DA também tem sido aplicada como método para discriminaçao de amostras e identificaçao de compostos diferenciadores na classificaçao de micro-organismos66,98 muitas vezes empregado juntamente com a PCA.38,135,136 Dessa forma, resultados mais confiáveis com relaçao a discriminaçao e classificaçao de amostras sao obtidos pela associaçao das duas estratégias. Mediante a alta complexidade dos dados metabolômicos, ferramentas como heatmaps têm sido empregadas a fim de facilitar a visualizaçao dos metabólitos identificados e condiçoes avaliadas.49 A visualizaçao dos dados se torna ainda mais explicativa quando as ferramentas de PCA e heatmaps sao empregadas em conjunto, sendo essas ferramentas extremamente úteis nesse contexto.35,49,63,100,137 Além disso, PCA e HCA também sao utilizadas em conjunto para facilitar na compreensao dos problemas biológicos investigados.35,46,92 A ferramenta de PLS-DA foi utilizada para a avaliaçao das variaçoes metabólicas de Pseudomonas syringae frente às mudanças de temperatura. Algumas amostras representativas das classes investigadas foram utilizadas para a elaboraçao do modelo estatístico que utilizou tanto dados de NMR quanto MS, fornecendo resultados satisfatórios em ambos os casos.61 Além disso, a ferramenta também foi empregada juntamente com HCA na análise de Streptomyces sp. como uma ferramenta de classificaçao quimiotaxonômica baseada em metabolômica.47 A MCR-ALS (do inglês, Multivariate Curve Resolution with Alternating Least Squares) é um método amplamente utilizado na resoluçao de curvas, essa ferramenta foi empregada com o objetivo de resolver diretamente o número máximo de perfis de eluiçao individuais e perfis espectrais de massa pura de todos os metabólitos possíveis extraídos das amostras de leveduras avaliando também o efeito da exposiçao ao Cu (II) sobre elas.29 De forma a facilitar e integrar as etapas de processamento de dados, algumas plataformas agrupam as etapas de pré-processamento com a análise estatística, como é o caso da MetaboNexus, MetaboAnalyst e MetaComp,138 que permitem ao usuário realizar PCA, PLS-DA, e outras análises multivariadas e univariadas. Além disso, elas integram a possibilidade de reportes gráficos, como gráficos de scores, gráficos de diagnóstico e heatmaps, enquanto a funçao de busca de metabólitos usa três repositórios principais de metabólitos, HMDB, MassBank e METLIN123 facilitando assim, a etapa de tratamento de dados. Identificaçao dos metabólitos A elucidaçao estrutural é outro componente essencial das análises metabolômicas e é um processo desafiador e demorado, especialmente na metabolômica microbiana.121 Isso ocorre porque os micro-organismos podem produzir uma gama muito diversificada de metabólitos em diferentes condiçoes, que podem nao ser comumente reconhecidos em bibliotecas padrao.5 Embora seja possível realizar a elucidaçao estrutural de um único metabólito utilizando métodos baseados em NMR ou MS, essa tarefa se torna extremamente inviável para os estudos metabolômicos, em que, centenas a milhares de compostos sao detectados simultaneamente. Dessa forma, a identificaçao é baseada em características de correspondência dos espectros da amostra em relaçao a uma base de dados de referência.124 Assim, em complemento aos dados de LC-MS e GC-MS comuns, informaçoes adicionais sao alcançadas a partir de espectros de massa de ordem superior, como MSn. As fragmentaçoes sofridas pelas espécies de interesse fornecem uma impressao digital da estrutura molecular que podem ser comparadas com as bibliotecas de espectros disponíveis.139 Apesar das limitaçoes, mediante o crescente interesse nos estudos metabolômicos de micro-organismos e, consequentemente, a geraçao de informaçoes, uma série de bases de dados microbianas baseada em dados de NMR e MS têm surgido, acelerando assim o processo de identificaçao dos metabólitos. Paralelamente, os pesquisadores também podem recorrer a bancos de dados de metabolômica geral que também podem fornecer informaçoes úteis.5 Os bancos de dados HMDB e METLIN foram empregados na identificaçao de metabólitos provenientes da bactéria Pseudomonas syringae a fim de investigar os metabólitos relevantes frente às variaçoes de temperatura para a espécie.61 Contudo, apesar de se tratar de um banco de dados para o metaboloma humano, o HMDB tem sido utilizado também na elucidaçao estrutural de compostos provenientes de micro-organismos.26,37 Além disso, bibliotecas como Fiehn140 e NIST69 também sao amplamente empregadas.35 Embora as bibliotecas espectrais de massas estejam crescendo, a quantidade de moléculas identificadas ainda é relativamente pequena em comparaçao ao número de compostos que poderiam estar presentes em amostras típicas, principalmente quando nao há dados de referência disponíveis, como é o caso de diversos micro-organismos. Assim, as ferramentas computacionais aliadas à MS buscam sanar esses problemas com o desenvolvimento de plataformas altamente eficazes. Essas plataformas sao capazes de propor identificaçoes de compostos desconhecidos, utilizando a consulta de diferentes bases de dados e combinaçao da informaçao extraída para o processo de identificaçao. Plataformas como MetFrag e MetFusion têm permitido o processamento de centenas de espectros de fragmentaçao de MS em poucos minutos e a adiçao de diferentes tipos de informaçoes mostrou melhorar as taxas de identificaçao de 6% até 70%, dependendo do conjunto de dados e fontes de informaçao utilizadas.139 As ferramentas de enriquecimento sao estratégias que utilizam o conhecimento de base de dados e algoritmos estatísticos para executar o processo de identificaçao. A metodologia mais utilizada para realizar essa análise é denominada enriquecimento funcional ou análise de sobre-representaçao (ORA, do inglês Over-Representation Analysis). A ferramenta ORA é muito flexível e de fácil implementaçao, por isso tem sido aplicada em diversas plataformas metabolômicas e banco de dados. A análise se inicia com uma lista de metabólitos de interesse e testa se certos grupos de metabólitos aparecem mais frequentemente do que seria esperado por chance aleatória, sendo, para isso, aplicados testes estatísticos de comparaçao como teste t ou ANOVA e os metabólitos significantes sao selecionados utilizando um limite ou critério específico.124,141 A fim de investigar a interferência do meio de cultura no metabolismo de Escherichia coli e Saccharomyces cerevisiae, uma outra ferramenta de enriquecimento (MSEA, do inglês Metabolite Set Enrichment Analysis) foi empregada utilizando a plataforma MetaboAnalyst. A MSEA utiliza diretamente o enriquecimento de grupos usando os dados de concentraçao completa sem pré-seleçao de metabólitos significativos sendo possível identificar os compostos que diferem significativamente frente a uma variaçao, nesse caso o meio de cultura.35 Uma última ferramenta com desenvolvimento recente na meta-bolômica "Big Data" baseada em algoritmos matemáticos para organizar e visualizar dados MS é o molecular networking (MN). Várias publicaçoes têm demonstrado o potencial do molecular networking em estudos de metabolômica microbiana com a capacidade de analisar conjuntos de dados em grande escala.30,50,64 O funcionamento da ferramenta baseia-se no princípio de que estruturas moleculares análogas se comportam de maneira semelhante na fase gasosa, dando origem a padroes de fragmentaçao semelhantes na espectrometria de massas. O molecular networking é aplicado a partir de algumas etapas básicas: (1) coleta de dados MS/MS; (2) cálculo de similaridade de espectros usando um algoritmo especial para gerar uma rede molecular; (3) visualizaçao da rede. Os dados sao visualizados por similaridade química no qual um nó representa um espectro MS/MS de consenso marcado pela massa original dos íons precursores e a espessura da linha que os liga representa o escore de semelhança entre os espectros, criando grupos de famílias moleculares, quanto mais próximo o valor do escore de 1, maior a semelhança no espectro de MS/MS, sugerindo que os compostos possuem uma similaridade química.121 Na plataforma GNPS (gnps.ucsd.edu) é possível carregar os dados de MS/MS online e gerar o molecular networking, podendo ser visualizado na plataforma ou utilizando softwares como o Cytoscape.142 O MN foi aplicado na elucidaçao estrutural de diversos metabólitos provenientes de comunidades microbianas multiespécies sintéticas. Nesse caso, os metabólitos conhecidos das culturas individuais auxiliaram na descoberta de diferentes metabólitos presentes nas co-culturas.64 A plataforma também foi aplicada no estudo de Clostridium acetobutylicum apresentando uma estratégia de manipulaçao do metabolismo secundário de um organismo para melhorar traços relevantes para aplicaçoes industriais.51 Devido à sua facilidade no tratamento de dados e emprego em diferentes áreas, o MN vem sendo amplamente utilizado nos estudos metabolômicos, especialmente nos estudos de micro-organismos, onde as plataformas e bancos de dados de metabólitos disponíveis ainda é escassa.30,50,143 Enquanto alguns metabolomas, como o metaboloma humano, têm sido amplamente caracterizados e disponibilizados para amplo acesso em bancos de dados, os bancos de dados de metaboloma microbiano têm se desenvolvido apenas para algumas espécies modelo. A falta de dados é um resultado direto da alta diversidade de organismos e espécies e o estudo dos metabolomas microbianos oferece muitas oportunidades para o desenvolvimento futuro.121
DESAFIOS FUTUROS Muitos desafios ainda sao encontrados no contexto da ciência metabolômica e, mais especificamente, na metabolômica microbiana. Dentre esses desafios, está a identificaçao simultânea de uma ampla gama de metabólitos intactos e a quantificaçao com precisao de cada molécula na amostra.144 Assim, a utilizaçao de múltiplas plataformas analíticas é uma tendência emergente na metabolômica, e a integraçao dos dados obtidos por NMR e MS podem auxiliar na resoluçao dessas limitaçoes.24 Diversos estudos apresentados nesta revisao demonstraram o uso de múltiplas plataformas nos estudos avançados de sistemas microbianos, contudo, esses estudos ainda nao limitados, tanto em relaçao ao processamento de dados quanto a verdadeira compreensao global do sistema. Em termos de preparo de amostras, também sao encontradas algumas barreiras que precisam ser superadas, como por exemplo a falta de protocolos padroes em metabolômica microbiana para o quenching instantâneo da atividade metabólica, bem como para extraçao abrangente de metabólitos e análise dos metabólitos de interesse. Os procedimentos atuais sao desenvolvidos para organismos específicos e sao incapazes de prevenir a perda de metabólitos durante o preparo. Com relaçao aos métodos de extraçao utilizados, esses ainda sao mais adequados para a análise de certas classes de metabólitos e precisam ser aperfeiçoados. Portanto, o perfil abrangente de metabólitos pode exigir a combinaçao e a integraçao de múltiplos métodos de extraçao.5,15,22 Além disso, o emprego de microtécnicas de extraçao também tem sido um grande atrativo para esse tipo de estudo. Métodos miniaturizados também apresentam eficiências de extraçao e reprodutibilidade satisfatórias, além de se apresentarem como métodos limpos com baixo consumo de solventes ou reutilizáveis. Na etapa de processamento de dados em metabolômica, sao encontrados problemas a serem superados desde a etapa de pré-processamento até a identificaçao metabólica. Primeiramente, o processamento de dados altamente eficaz e automático continua sendo uma tarefa difícil, principalmente nos casos em que as etapas preliminares precisam ser implementadas para um grande volume de dados. A identificaçao confiável de metabólitos desconhecidos a partir de dados complexos de espectros permanece um grande desafio e, embora surjam novos bancos de dados para a metabolômica microbiana, eles sao limitados a espécies específicas.4,5 No entanto, uma mudança desse cenário vem ocorrendo e tem sido sustentada pelos avanços em técnicas instrumentais, principalmente na espectrometria de massas. Além disso, uma ferramenta que tem ganhado espaço e sendo empregada em diversos contextos é o molecular networking. A confiabilidade e funcionalidade dessa ferramenta tem sido grande atrativo para os pesquisadores da área de metabolômica, e no âmbito de identificaçao de metabólitos microbianos, ela possivelmente será cada vez mais explorada. Na comunidade científica, muitas divergências ainda sao encontradas quanto a uma abordagem metabolômica genuína. No âmbito de micro-organismos, foi possível observar que grande parte dos estudos nao seguem à risca todo o fluxo de trabalho usualmente adotado em metabolômica, sendo muitas vezes apenas uma avaliaçao global dos metabólitos ou um perfil de metabólitos específicos. Apesar de serem encontrados estudos que avaliem as variaçoes metabólicas frente a mudanças específicas, ainda existem muitos casos em que apenas uma exploraçao do perfil metabólico do micro-organismo é realizada, sendo esses estudos também de extrema importância para a compreensao de uma via metabólica específica.
AGRADECIMENTOS Os autores agradecem a Fundaçao de Amparo à Pesquisa do Estado de Sao Paulo (FAPESP - Processo 2016/20547-2, Processo 2018/03670-0 e Processo 2017/24462-4) e a Coordenaçao de Aperfeiçoamento de Pessoal de Nível Superior (CAPES).
REFERENCIAS 1. Lin, C. Y.; Viant, M. R.; Tjeerdema, R. S.; J. Pestic. Sci. 2006, 31, 245. 2. Allen, J.; Davey, H. M.; Broadhurst, D.; Heald, J. K.; Rowland, J. J.; Oliver, S. G.; Kell, D. B.; Nat. Biotechnol. 2003, 21, 692. 3. Patterson, A. D.; Li, H.; Eichler, G. S.; Krausz, K. W.; Weinstein, J. N.; Fornace, A. J.; Gonzalez, F. J.; Idle, J. R.; Anal. Chem. 2008, 80, 665. 4. Yi, L.; Dong, N.; Yun, Y.; Deng, B.; Ren, D.; Liu, S.; Liang, Y.; Anal. Chim. Acta 2016, 914, 17. 5. Xu, Y. J.; Wang, C.; Ho, W. E.; Ong, C. N.; TrAC - Trends Anal. Chem. 2014, 56, 37. 6. Yang, S.; Hoggard, J. C.; Lidstrom, M. E.; Synovec, R. E. Em Metabolomics in Practice; Lammerhofer, M., Weckworth, W., eds.; Wiley: Hoboken, 2013. 7. Fiehn, O.; Plant Mol. Biol. 2002, 48, 155. 8. Goodacre, R.; Vaidyanathan, S.; Dunn, W. B.; Harrigan, G. G.; Kell, D. B.; Trends Biotechnol. 2004, 22, 245. 9. Wenk, M. R.; Lipidomics of host-pathogen interactions. FEBS Lett. 2006, 580, 5541. 10. Boots, A. W.; Smolinska, A.; Van Berkel, J. J. B. N.; Fijten, R. R. R.; Stobberingh, E. E.; Boumans, M. L. L.; Moonen, E. J.; Wouters, E. F. M.; Dallinga, J. W.; Van Schooten, F. J.; J. Breath Res. 2014, 027106. 11. Brunelli, L.; Caiola, E.; Marabese, M.; Broggini, M.; Pastorelli, R.; Oncotarget 2014, 5, 4722. 12. Raamsdonk, L. M.; Teusink, B.; Broadhurst, D.; Zhang, N.; Hayes, A.; Walsh, M. C.; Berden, J. A.; Brindle, K. M.; Kell, D. B.; Rowland, J. J.; Westerhoff, H. V; Van Dam, K.; Oliver, S. G.; Nat. Biotechnol. 2001, 19, 45. 13. Boroughs, L. K.; Deberardinis, R. J.; Nat. Cell Biol. 2015, 17, 351. 14. Peisl, B. Y. L.; Schymanski, E. L.; Wilmes, P.; Anal. Chim. Acta 2018, 1037, 13 15. Canuto, G.; Costa, J. L.; Cruz, P.; Souza, A.; Faccio, A.; Klassen, A.; Rodrigues, K.; Tavares, M.; Quim. Nova 2017, 41, 75. 16. Yang, S.; Hoggard, J. C.; Lidstrom, M. E.; Synovec, R. E.; J. Chromatogr. A 2013, 1317, 175. 17. Smart, K. F.; Aggio, R. B. M.; Van Houtte, J. R.; Villas-Boas, S. G.; Nat. Protoc. 2010, 5, 1709. 18. Dunn, W. B.; Ellis, D. I.; TrAC - Trends Anal. Chem. 2005, 24, 285. 19. Naz, S.; Vallejo, M.; Garcia, A.; Barbas, C.; J. Chromatogr. A 2014, 1353, 99. 20. Kondakova, T.; Merlet-Machour, N.; Chapelle, M.; Preterre, D.; Dionnet, F.; Feuilloley, M.; Orange, N.; Duclairoir Poc, C.; Res. Microbiol. 2015, 166, 1. 21. Gravouil, K.; Ferru-Clement, R.; Colas, S.; Helye, R.; Kadri, L.; Bourdeau, L.; Moumen, B.; Mercier, A.; Ferreira, T.; Environ. Sci. Technol. 2017, 51, 5172. 22. Beale, D. J.; Kouremenos, K. A.; Palombo, E. A.; Microbial Metabolomics, Springer International Publishing: Cham, 2016. 23. Khoomrung, S.; Wanichthanarak, K.; Nookaew, I.; Thamsermsang, O.; Seubnooch, P.; Laohapand, T.; Akarasereenont, P.; Front. Pharmacol. 2017, 8, 474. 24. Viant, M. R.; Kurland, I. J.; Jones, M. R.; Dunn, W. B.; Curr. Opin. Chem. Biol. 2017, 36, 64. 25. Pinu, F. R.; Villas-Boas, S. G.; Aggio, R.; Metabolites 2017, 7, 53. 26. Ząbek, A.; Junka, A.; Szymczyk, P.; Wojtowicz, W.; Klimek-Ochab, M.; Młynarz, P.; J. Basic Microbiol. 2017, 57, 428. 27. Carro, L.; Persson, T.; Pujic, P.; Alloisio, N.; Fournier, P.; Boubakri, H.; Pawlowski, K.; Normand, P.; Symbiosis 2016, 70, 37. 28. Johnson, W. M.; Kido Soule, M. C.; Kujawinski, E. B.; ISME J. 2016, 10, 2304. 29. Farres, M.; Pina, B.; Tauler, R.; Metallomics 2016, 8, 790. 30. Nguyen, D. D.; Melnik, A. V.; Koyama, N.; Lu, X.; Schorn, M.; Fang, J.; Aguinaldo, K.; Lincecum, T. L.; Ghequire, M. G. K.; Carrion, V. J.; Cheng, T. L.; Duggan, B. M.; Malone, J. G.; Mauchline, T. H.; Sanchez, L. M.; Kilpatrick, A. M.; Raaijmakers, J. M.; De Mot, R.; Moore, B. S.; Medema, M. H.; Dorrestein, P. C.; Nat. Microbiol. 2016, 2, 1. 31. Villas-Boas, S. G.; Mas, S.; Akesson, M.; Smedsgaard, J.; Nielsen, J.; Mass Spectrom. Rev. 2005, 24, 613. 32. Kapoore, R. V.; Coyle, R.; Staton, C. A.; Brown, N. J.; Vaid, yanathan, S.; Analyst 2017, 142, 2038. 33. Wittmann, C.; Kromer, J. O.; Kiefer, P.; Binz, T.; Heinzle, E.; Anal. Biochem. 2004, 327, 135. 34. Lorenz, M. A.; Burant, C. F.; Kennedy, R. T.; Anal. Chem. 2011, 83, 3406. 35. Kim, J.; Kim, K. H.; Process Biochem. 2017, 57, 64. 36. Shi, Y.; Pan, C.; Auckloo, B. N.; Chen, X.; Chen, C. T. A.; Wang, K.; Wu, X.; Ye, Y.; Wu, B.; Appl. Microbiol. Biotechnol. 2017, 101, 1395. 37. Li, H.; Xia, X.; Li, X.; Naren, G.; Fu, Q.; Wang, Y.; Wu, C.; Ding, S.; Zhang, S.; Jiang, H.; Li, J.; Shen, J.; J. Proteome Res. 2015, 14, 1060. 38. Bo, T.; Liu, M.; Zhong, C.; Zhang, Q.; Su, Q.-Z.; Tan, Z.-L.; Han, P.-P.; Jia, S.-R.; J. Agric. Food Chem. 2014, 62, 4454. 39. Tredwell, G. D.; Aw, R.; Edwards-Jones, B.; Leak, D. J.; Bundy, J. G.; J. Ind. Microbiol. Biotechnol. 2017, 44, 413. 40. Pluskal, T.; Yanagida, M.; Cold Spring Harb. Protoc. 2016, 2016, 1044. 41. AlMasoud, N.; Xu, Y.; Trivedi, D. K.; Salivo, S.; Abban, T.; Rattray, N. J. W.; Szula, E.; AlRabiah, H.; Sayqal, A.; Goodacre, R.; Anal. Bioanal. Chem. 2016, 408, 7865. 42. Pomraning, K. R.; Wei, S.; Karagiosis, S. A.; Kim, Y. M.; Dohnalkova, A. C.; Arey, B. W.; Bredeweg, E. L.; Orr, G.; Metz, T. O.; Baker, S. E.; PLoS One 2015, 10, 1. 43. Anasontzis, G. E.; Kourtoglou, E.; Villas-Boas, S. G.; Hatzinikolaou, D. G.; Christakopoulos, P.; Front. Microbiol. 2016, 7, 1. 44. Liu, M.; Zhong, C.; Wu, X. Y.; Wei, Y. Q.; Bo, T.; Han, P. P.; Jia, S. R.; Biochem. Eng. J. 2015, 101, 85. 45. Drapal, M.; Perez-Fons, L.; Wheeler, P. R.; Fraser, P. D.; J. Microbiol. Methods 2014, 106, 23. 46. Kim, S.; Lee, D. Y.; Wohlgemuth, G.; Park, H. S.; Fiehn, O.; Kim, K. H.; Anal. Chem. 2013, 85, 2169. 47. Lee, M. Y.; Kim, H. Y.; Lee, S.; Kim, J. G.; Suh, J. W.; Lee, C. H.; J. Microbiol. Biotechnol. 2015, 25, 1265. 48. Dunn, W. B.; Bailey, N. J. C.; Johnson, H. E.; Analyst 2005, 130, 606. 49. Soboń, A.; Szewczyk, R.; Rożalska, S.; Długoński, J.; Int. Biodeterior. Biodegrad. 2018, 127, 130. 50. Spraker, J. E.; Sanchez, L. M.; Lowe, T. M.; Dorrestein, P. C.; Keller, N. P.; ISME J. 2016, 10, 2317. 51. Herman, N. A.; Kim, S. J.; Li, J. S.; Cai, W.; Koshino, H.; Zhang, W.; Nat. Commun. 2017, 8, 1514. 52. Weidt, S.; Haggarty, J.; Kean, R.; Cojocariu, C. I.; Silcock, P. J.; Rajendran, R.; Ramage, G.; Burgess, K. E. V.; Metabolomics 2016, 12, 1. 53. Pacini, T.; Fu, W.; Gudmundsson, S.; Chiaravalle, A. E.; Brynjolfson, S.; Palsson, B. O.; Astarita, G.; Paglia, G.; Anal. Chem. 2015, 87, 2593. 54. Mandelli, F.; Couger, M. B.; Paixao, D. A. A.; Machado, C. B.; Carnielli, C. M.; Aricetti, J. A.; Polikarpov, I.; Prade, R.; Caldana, C.; Paes Leme, A. F.; Mercadante, A. Z.; Riano-Pachon, D. M.; Squina, F. M.; Extremophiles 2017, 21, 775. 55. Nizio, K. D.; Perrault, K. A.; Troobnikoff, A. N.; Ueland, M.; Shoma, S.; Iredell, J. R.; Middleton, P. G.; Forbes, S. L.; J. Breath Res. 2016, 10, 026008. 56. Rees, C. A.; Stefanuto, P.-H.; Beattie, S. R.; Bultman, K. M.; Cramer, R. A.; Hill, J. E.; J. Breath Res. 2017, 11, 036003. 57. Dunkel, T.; Gallego, de L.; S, E. L.; Schonsee, C. D.; Hesse, T.; Jochmann, M.; Wingender, J.; Denecke, M.; Water Res. 2016, 88, 510. 58. Salvador, A. C.; Baptista, I.; Barros, A. S.; Gomes, N. C. M.; Cunha, A.; Almeida, A.; Rocha, S. M.; PLoS One 2013, 8, e59338. 59. Rees, C. A.; Burklund, A.; Stefanuto, P.; Schwartzman, J. D.; Hill, J. E.; J. Breath Res. 2018, 12, 026001. 60. Barkal, L. J.; Theberge, A. B.; Guo, C.-J.; Spraker, J.; Rappert, L.; Berthier, J.; Brakke, K. A.; Wang, C. C. C.; Beebe, D. J.; Keller, N. P.; Berthier, E.; Nat. Commun. 2016, 7, 10610. 61. Jousse, C.; Dalle, C.; Canet, I.; Lagree, M.; Traikia, M.; Lyan, B.; Mendes, C.; Sancelme, M.; Amato, P.; Delort, A.-M.; Metabolomics 2018, 14, 11. 62. Smedsgaard, J.; J. Chromatogr. A 1997, 760, 264. 63. Zhong, F.; Xu, M.; Schelli, K.; Rutowski, J.; Holmen, B. A.; Zhu, J. Ecotoxicol. Environ. Saf. 2017, 142, 164. 64. Shi, Y.; Pan, C.; Wang, K.; Chen, X.; Wu, X.; Chen, C. T. A.; Wu, B.; Environ. Microbiol. 2017, 19, 3606. 65. Bohni, N.; Hofstetter, V.; Gindro, K.; Buyck, B.; Schumpp, O.; Bertrand, S.; Monod, M.; Wolfender, J.-L.; Molecules 2016, 21, 370. 66. Lau, S. K. P.; Lam, C.-W.; Curreem, S. O. T.; Lee, K.-C.; Chow, W.-N.; Lau, C. C. Y.; Sridhar, S.; Wong, S. C. Y.; Martelli, P.; Hui, S.-W.; Yuen, K.-Y.; Woo, P. C. Y.; Cell Biosci. 2015, 5, 26. 67. Bose, U.; Hewavitharana, A. K.; Ng, Y. K.; Shaw, P. N.; Fuerst, J. A.; Hodson, M. P.; Mar. Drugs 2015, 13, 249. 68. De Lima, P. F.; Furlan, M. F.; De Lima Ribeiro, F. A.; Pascholati, S. F.; Augusto, F.; J. Sep. Sci. 2015, 38, 1924. 69. Rees, C. A.; Franchina, F. A.; Nordick, K. V.; Kim, P. J.; Hill, J. E.; J. Appl. Microbiol. 2017, 122, 785. 70. Johanningsmeier, S. D.; McFeeters, R. F.; Int. J. Food Microbiol. 2015, 215, 40. 71. Mousavi, F.; Gionfriddo, E.; Carasek, E.; Souza-Silva, E. A.; Pawliszyn, J.; Metabolomics 2016, 12, 169. 72. Rees, C. A.; Shen, A.; Hill, J. E.; J. Chromatogr. B 2016, 1039, 8. 73. Alves, Z.; Melo, A.; Figueiredo, A. R.; Coimbra, M. A.; Gomes, A. C.; Rocha, S. M.; PLoS One 2015, 10, e0143641. 74. Zhang, Q. H.; Zhou, L. Di; Chen, H.; Wang, C. Z.; Xia, Z. N.; Yuan, C. S.; TrAC - Trends Anal. Chem. 2016, 80, 57. 75. Souza Silva, E. A.; Risticevic, S.; Pawliszyn, J.; TrAC - Trends Anal. Chem. 2013, 43, 24. 76. Willers, C.; Jansen van Rensburg, P. J.; Claassens, S.; J. Appl. Microbiol. 2015, 118, 1251. 77. Theodoridis, G.; Gika, H. G.; Wilson, I. D.; Mass Spectrom. Rev. 2011, 47, 987. 78. Lee, T. H.; Chang, J. S.; Wang, H. Y.; Anal. Chem. 2013, 85, 2155. 79. Li, L.; Han, J.; Wang, Z.; Liu, J.; Wei, J.; Xiong, S.; Zhao, Z.; Int. J. Mol. Sci. 2014, 15, 10492. 80. Qiao, B.; Lu, H.; Cao, Y. X.; Chen, R.; Yuan, Y. J.; Eng. Life Sci. 2013, 13, 496. 81. Takeshita, T.; Ota, S.; Yamazaki, T.; Hirata, A.; Zachleder, V.; Kawano, S.; Bioresour. Technol. 2014, 158, 127. 82. Řezanka, T.; Kolouchova, I.; Sigler, K.; Biochim. Biophys. Acta - Mol. Cell Biol. Lipids 2016, 1861, 1634. 83. Capusoni, C.; Rodighiero, V.; Cucchetti, D.; Galafassi, S.; Bianchi, D.; Franzosi, G.; Compagno, C.; Bioresour. Technol. 2017, 238, 281. 84. Singh, A.; MacKenzie, A.; Girnun, G.; Del Poeta, M.; J. Lipid Res. 2017, 58, 2017. 85. He, Y.; Zhang, P.; Huang, S.; Wang, T.; Ji, Y.; Xu, J.; Biotechnol. Biofuels 2017, 10, 275. 86. Elahee Doomun, S.; Loke, S.; O’Callaghan, S.; Callahan, D. A.; Metabolites 2016, 6, 42. 87. Kumar, M.; Morya, R.; Gnansounou, E.; Larroche, C.; Thakur, I. S.; Bioresour. Technol. 2017, 243, 893. 88. Fu, Z.; Verderame, T. D.; Leighton, J. M.; Sampey, B. P.; Appelbaum, E. R.; Patel, P. S.; Aon, J. C.; Microb. Cell Fact. 2014, 13, 32. 89. Sasaki, D.; Sasaki, K.; Tsuge, Y.; Morita, M.; Kondo, A.; Bioresour. Technol. 2014, 172, 83. 90. Wolfender, J.-L.; Marti, G.; Thomas, A.; Bertrand, S.; J. Chromatogr. A 2015, 1382, 136. 91. Dalluge, J.; Beens, J.; Brinkman, U. A. T.; J. Chromatogr. A 2003, 1000, 69. 92. Rees, C. A.; Nordick, K. V.; Franchina, F. A.; Lewis, A. E.; Hirsch, E. B.; Hill, J. E.; Metabolomics 2017, 13, 18. 93. Mohler, R. E.; Dombek, K. M.; Hoggard, J. C.; Young, E. T.; Synovec, R. E.; Anal. Chem. 2006, 78, 2700. 94. Aldridge, B. B.; Rhee, K. Y.; Curr. Opin. Microbiol. 2014, 19, 90. 95. Triebl, A.; Hartler, J.; Trotzmuller, M.; C. Kofeler, H.; Lipidomics: Biochim. Biophys. Acta - Mol. Cell Biol. Lipids 2017, 1862, 740. 96. Tripathy, K.; J. Comput. Sci. Syst. Biol. 2012, 04, 93. 97. Milker, S.; Goncalves, L. C. P.; Fink, M. J.; Rudroff, F.; Front. Microbiol. 2017, 8, 1. 98. Hunter, J. E.; Frada, M. J.; Fredricks, H. F.; Vardi, A.; Van Mooy, B. A. S.; Front. Mar. Sci. 2015, 2, 1. 99. Zawadzka, K.; Bernat, P.; Felczak, A.; Lisowska, K.; Environ. Sci. Pollut. Res. 2015, 22, 19658. 100. Feng, Y.; Zhao, Y.; Guo, Y.; Liu, S.; Water Res. 2018, 128, 402. 101. Stopka, S. a; Shrestha, B.; Marechal, E.; Falconet, D.; Vertes, A.; Analyst 2014, 139, 5945. 102. Groessl, M.; Graf, S.; Knochenmuss, R.; Analyst 2015, 140, 6904. 103. Zuniga, C.; Zaramela, L.; Zengler, K.; Microb. Biotechnol. 2017, 10, 1500. 104. Perez-Garcia, O.; Lear, G.; Singhal, N.; Front. Microbiol. 2016, 7, 673. 105. von Kamp, A.; Klamt, S.; Nat. Commun. 2017, 8, 15956. 106. Schenk, E. R.; Nau, F.; Thompson, C. J.; Tse-Dinh, Y. C.; Fernandez- Lima, F.; J. Mass Spectrom. 2015, 50, 88. 107. Calvano, C. D.; Italiano, F.; Catucci, L.; Agostiano, A.; Cataldi, T. R. I.; Palmisano, F.; Trotta, M.; BioMetals 2014, 27, 65. 108. Vitale, R.; Roine, E.; Bamford, D. H.; Corcelli, A.; Biochim. Biophys. Acta - Mol. Cell Biol. Lipids 2013, 1831, 872. 109. de Bruijn, I.; Cheng, X.; de Jager, V.; Exposito, R. G.; Watrous, J.; Patel, N.; Postma, J.; Dorrestein, P. C.; Kobayashi, D.; Raaijmakers, J. M.; BMC Genomics 2015, 16, 991. 110. Ho, Y.-N.; Shu, L.-J.; Yang, Y.-L.; Wiley Interdiscip. Rev.: Syst. Biol. Med. 2017, 9, e1387. 111. Caprioli, R. M.; Farmer, T. B.; Gile, J.; Anal. Chem. 1997, 69, 4751. 112. Andersson, M.; Groseclose, M. R.; Deutch, A. Y.; Caprioli, R. M.; Nat. Methods 2008, 5, 101. 113. Shih, C.-J.; Chen, P.-Y.; Liaw, C.-C.; Lai, Y.-M.; Yang, Y.-L.; Nat. Prod. Rep. 2014, 31, 739. 114. Sandonato, B. B.; Santos, V. G.; Luizete, M. F.; Bronzel, J. L.; Eberlin, M. N.; Milagre, H. M. S.; J. Braz. Chem. Soc. 2017, 28, 521. 115. Esquenazi, E.; Coates, C.; Simmons, L.; Gonzalez, D.; Gerwick, W. H.; Dorrestein, P. C.; Mol. Biosyst. 2008, 4, 562. 116. Simmons, T. L.; Coates, R. C.; Clark, B. R.; Engene, N.; Gonzalez, D.; Esquenazi, E.; Dorrestein, P. C.; Gerwick, W. H.; Proc. Natl. Acad. Sci. U. S. A. 2008, 105, 4587. 117. Lobete, M. M.; Fernandez, E. N.; Van Impe, J. F. M.; Front. Microbiol. 2015, 6, 1. 118. Weisener, C. G.; Reid, T.; Surf. Interface Anal. 2017, 49, 1416. 119. Sandrin, T. R.; Demirev, P. A.; Mass Spectrom. Rev. 2018, 37, 321. 120. Ho, Y.-N.; Shu, L.-J.; Yang, Y.-L.; Wiley Interdiscip. Rev.: Syst. Biol. Med. 2017, 9, e1387. 121. Luzzatto-Knaan, T.; Melnik, A. V.; Dorrestein, P. C.; Analyst 2015, 140, 4949. 122. Krastanov, A.; Biotechnol. Biotechnol. Equip. 2010, 24, 1537. 123. Misra, B. B.; van der Hooft, J. J. J.; Electrophoresis 2016, 37, 86. 124. Metabolomics: From Fundamentals to Clinical Applications; Sussulini, A., org.; Springer International Publishing: Cham, 2017. 125. Coble, J. B.; Fraga, C. G.; J. Chromatogr. A 2014, 1358, 155. 126. Castillo, S.; Gopalacharyulu, P.; Yetukuri, L.; Oresič, M.; Chemom. Intell. Lab. Syst. 2011, 108, 23. 127. Wei, X.; Sun, W.; Shi, X.; Koo, I.; Wang, B.; Zhang, J.; Yin, X.; Tang, Y.; Bogdanov, B.; Kim, S.; Zhou, Z.; McClain, C.; Zhang, X.; Anal. Chem. 2011, 83, 7668. 128. Duran, A. L.; Yang, J.; Wang, L.; Sumner, L. W.; Bioinformatics 2003, 19, 2283. 129. Hiller, K.; Hangebrauk, J.; Jäger, C.; Spura, J.; Schreiber, K.; Schomburg, D.; Anal. Chem. 2009, 81, 3429. 130. Broeckling, C. D.; Reddy, I. R.; Duran, A. L.; Zhao, X.; Sumner, L. W.; Anal. Chem. 2006, 78, 4334. 131. Allmann, S.; Mazet, M.; Ziebart, N.; Bouyssou, G.; Fouillen, L.; Dupuy, J.-W.; Bonneu, M.; Moreau, P.; Bringaud, F.; Boshart, M.; PLoS One 2014, 9, e114628. 132. Meissner-Roloff, R. J.; Koekemoer, G.; Warren, R. M.; Loots, D. T.; Metabolomics 2012, 8, 1194. 133. Lee, L. C.; Liong, C.-Y.; Jemain, A. A.; Analyst 2018, 143, 3526. 134. Liu, Y.; Zhang, J.; Nie, H.; Dong, C.; Li, Z.; Zheng, Z.; Bai, Y.; Liu, H.; Zhao, J.; Anal. Chem. 2014, 86, 7096. 135. Kouremenos, K. A.; Beale, D. J.; Antti, H.; Palombo, E. A.; J. Chromatogr. B 2014, 966, 179. 136. Chin, E. L.; Mishchuk, D. O.; Breksa, A. P.; Slupsky, C. M.; J. Agric. Food Chem. 2014, 62, 6585. 137. Szewczyk, R.; Kuśmierska, A.; Bernat, P.; Chemosphere 2018, 190, 174. 138. Zhai, P.; Yang, L.; Guo, X.; Wang, Z.; Guo, J.; Wang, X.; Zhu, H.; BMC Bioinformatics 2017, 18, 1. 139. Meier, R.; Ruttkies, C.; Treutler, H.; Neumann, S.; J. Biotechnol. 2017, 261, 137. 140. Zhai, P.; Yang, L.; Guo, X.; Wang, Z.; Guo, J.; Wang, X.; Zhu, H.; Kind, T.; Wohlgemuth, G.; Lee, D. Y.; Lu, Y.; Palazoglu, M.; Shahbaz, S.; Fiehn, O.; BMC Bioinformatics 2017, 81, 1. 141. Marco-Ramell, A.; Palau-Rodriguez, M.; Alay, A.; Tulipani, S.; Urpi-Sarda, M.; Sanchez-Pla, A.; Andres-Lacueva, C.; BMC Bioinformatics 2018, 19, 1. 142. Olivon, F.; Roussi, F.; Litaudon, M.; Touboul, D.; Anal. Bioanal. Chem. 2017, 409, 5767. 143. Yang, J. Y.; Sanchez, L. M.; Rath, C. M.; Liu, X.; Boudreau, P. D.; runs, N.; Glukhov, E.; Wodtke, A.; de Felicio, R.; Fenner, A.; Wong, W. R.; Linington, R. G.; Zhang, L.; Debonsi, H. M.; Gerwick, W. H.; Dorrestein, P. C.; J. Nat. Prod. 2013, 76, 1686. 144. German, J. B.; Gillies, L. A.; Smilowitz, J. T.; Zivkovic, A. M.; Watkins, S. M.; Curr. Opin. Lipidol. 2007, 18, 66. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access