Artigo
|
|
| Perfil metabólico dos extratos polares das folhas, galhos e cascas de Copaifera multijuga hayne (Copaíba mari-mari da Amazônia) por ESI-EM Metabolic profile of the polar extracts of the leaves, branches and barks of Copaifera multijuga hayne (Copaíba mari-mari Amazônia) by ESI-MS |
|
Davi S. OliveiraI; Leandro S. LimaI; Ananda S. AntonioI; Larissa S. M. WiedemannI; Valdir F. Veiga-JuniorI,II,*
I. Departamento de Química, Instituto de Ciências Exatas, Universidade Federal do Amazonas, Av. General Rodrigo Octávio, 6200, 69079-00 Manaus - AM, Brasil Recebido em 03/06/2019 *e-mail: valdir.veiga@gmail.com Copaifera multijuga Hayne is the main species of the genus in the Amazon region, being exploited for copaiba oil production mainly because of its therapeutic properties. The plant parts (leaves, branches, and barks) are widely used in folk medicine. However, they are little explored in terms of chemical composition. In this study, the polar extracts of leaves, branches, and barks of the species were analysed by direct infusion in ESI-MS with the aid of reference standards and MS2 fragmentation experiments. Twenty-six substances (carboxylic acids, flavonoids, condensed tannins, a diterpene and fifteen isomers of galoylquinic acids) were detected. Metabolic profiles were similar between extracts of leaves and branches, especially with the detection of galloylquinic acids and carboxylic acids. In bark extract, condensed tannins epicatechin-(4β-8)-epiafzelechin (dimer) and epicatechin-(4β-8)-epiafzelechin-(4β-8)-epiafzelechin (trimer) were detected. The PCA showed differences and similarities in the chemical composition of the copaíba mari-mari extracts. Thus, galloylquinic acids and flavonoids kaempferol, 7-O-methyl-kaempferol, quercetin-3-O-L-pentoside, and quercetin-3-O-L-rhamnoside are described for the first time in C. multijuga. The phytotherapeutic properties of teas of the plant parts of the species may be associated with the suggested polyphenols. INTRODUÇÃO Copaifera multijuga Hayne (Fabaceae), popularmente conhecida como copaibeira, pau d'óleo, copaíba mari-mari ou copaíba da folha pequena, encontra-se distribuída entre os estados do Amazonas e Pará, sendo considerada a principal espécie do gênero Copaifera na Amazônia.1 Há registros de sua ocorrência também nos estados da Bahia, Mato Grosso e Acre, alcançando o Peru e a Bolívia.1,2 A espécie é reconhecida por suas propriedades medicinais, principalmente devido ao óleo-resina, um fitoterápico natural utilizado na medicina popular para o tratamento de doenças urinárias, respiratórias, cutâneas e inflamatórias.1-3 O óleo-resina é rico em sesquiterpenos tais como, β-cariofileno, óxido de cariofileno, α-copaeno, α-humuleno e γ-muuroleno, bem como nos ácidos diterpênicos eperúico, agático, pinifólico, copálico e derivados, entre outros.1-3 Os chás das folhas e cascas do caule de C. multijuga são muito utilizados na medicina popular e podem ser preparados por infusão ou decocção para tratar doenças gástricas, digestivas e respiratórias.4 Embora a espécie apresente enorme potencial fitoterápico, pouco se conhece em relação à sua composição química. A maior parte dos conhecimentos químicos e farmacológicos de extratos do gênero (sem contar a oleoresina) se concentram na espécie C. langsdorffii Desf., que é típica do Cerrado.2 Sua composição destaca-se pela presença de substâncias fenólicas, como os 16 ácidos galoilquínicos descritos para os extratos de suas folhas,5 formando um grupo diversificado de mono-, di- ou tri-ésteres de ácido quínico nas variedades estruturais correspondentes aos ácidos gálico e 3-O-metil-gálico. Os ésteres de ácido quínico apresentam isomeria de posição entre os carbonos C3, C4 e C5 na cadeia do ácido quínico. Os glicosídeos quercetina-3-O-L-ramnosídeo e canferol-3-O-L-ramnsoídeo são derivados da estrutura da flavona e são considerados substâncias marcadoras das folhas desta espécie.6-9 A quercetina pode ser encontrada em outras formas de glicosídeos como a quercetina-3-O-L-glicosídeo e a quercetina-3-O-rutosídeo, identificados nos frutos dessa espécie.10 Os ácidos cumárico, cinâmico, benzóico e gálico, e derivados de catequinas, como: epicatequina galato, epicatequina e catequina nas formas diméricas epicatequina-(4β-8)-catequina e epicatequina-(4β-8)-epicatequina e os diterpenos nivenolídeo e os ácidos ent-caurenóico e poliáltico também foram descritos em extratos dos frutos da espécie.10,11 Em estudos realizados com os extratos das cascas de C. multijuga foram observadas propriedades antioxidantes e redução do estresse oxidativo induzido por paracetamol e o efeito antineoplásico em células tumorais de Ehrlich em testes in vitro e in vivo.12 As atividades antioxidantes podem estar relacionadas com a presença de polifenóis, como taninos condensados nas formas de dímero epicatequina-(4β-8)-epiafzelequina e trímero epicatequina-(4β-8)-epiafzelequina-(4β-8)-epiafzelequina sugeridos por experimentos de espectrometria de massas.12 Os usos medicinais das plantas nativas estão relacionados com a presença das substâncias fenólicas que possuem propriedades antioxidantes, anti-inflamatórias e gastroprotetoras, as quais proporcionam efeitos benéficos à saúde na prevenção contra radicais livres e doenças cancerígenas.7,12 A presença de polifenóis potencialmente ionizáveis descritos nos extratos das espécies de Copaifera estimulam os estudos de definição do fingerprint em análises com inserção direta em espectrômetro de massas (EM) utilizando a ionização por electrospray (ESI) associada aos experimentos de fragmentação MS2 induzida por colisão, abordagem que tem se mostrado eficiente em estudos preliminares da composição química.13 O experimento de fragmentação MS2 com analisador triplo quadrupolo (QqQ) permite caracterizar parcial e integralmente os metabólitos de um extrato a partir de seus íons moleculares e respectivos fragmentos.14 As substâncias fenólicas são eficientemente ionizadas por meio da fonte ESI, transferidas para a fase gasosa e detectadas em modo negativo como íons [M-H]-. A técnica exige pouca quantidade de amostra (mg), preparação simples, é rápida, altamente seletiva e precisa na produção de informações sobre as substâncias potencialmente ionizáveis.15 A interpretação dos dados espectrais obtidos por ESI-MS e o reconhecimento de padrões com análises estatísticas multivariadas estão cada vez mais presentes nos tratamentos dos dados, sendo a Análise de Componentes Principais (PCA) uma das técnicas mais empregadas, permitindo a distribuição das amostras em um plano bidimensional de acordo com o grau de importância das variáveis.16 O presente trabalho teve como objetivo caracterizar a composição química de diferentes extratos obtidos das partes vegetais de C. multijuga, principal espécie de copaíba da Amazônia Central e Ocidental utilizando ESI-QqQ-EM, contribuindo para o conhecimento da composição química e a promoção da segurança no consumo das partes vegetais da espécie na medicina popular.
PARTE EXPERIMENTAL Material vegetal, extração e padrões de referência As partes vegetais (folhas, galhos e cascas) de C. multijuga foram coletadas na Reserva Florestal Ducke do Instituto Nacional de Pesquisa da Amazônia (INPA), em Manaus, AM, Brasil. A exsicata do espécime foi depositada no herbário do INPA, sob o número INPA82.418. Ácido quínico, canferol e quercetina-3-O-L-ramnosídeo foram obtidos da Sigma Aldrich (EUA). O ácido ent-agático foi obtido e identificado em estudos anteriores do grupo de pesquisa Q-Bioma na Universidade Federal do Amazonas com o óleo-resina da espécie. O metanol grau P.A. (Tedia Brasil) foi utilizado como solvente extrator e grau HPLC (Tedia EUA) para as análises espectrométricas. As folhas, galhos e cascas foram secos à temperatura ambiente, triturados e submetidos, separadamente, à extração por maceração em metanol P.A. por 48 horas, as soluções foram filtradas e os extratos foram concentrados em evaporador rotativo com o auxílio de vácuo. Secos, os extratos foram padronizados em soluções em metanol na proporção de 1 mg mL-1. Alíquotas de 20 µL foram separadas e avolumadas para 1000 µL para os experimentos espectrométricos. Experimentos em espectrômetro de massas de varredura completa e fragmentação MS2 Os experimentos foram realizados em espectrômetro de massas de LCQ-Fleet (ThermoScientific, CA, EUA) equipado com uma fonte de ESI com analisador QqQ, operando em modo negativo de detecção. Os espectros de massas foram adquiridos e processados usando o software fornecido pelo fabricante (Xcalibur 2.1). As condições típicas do equipamento foram as seguintes: voltagem do capilar em 30 V, voltagem de pulverização de 5,0 kV, temperatura do capilar a 275 °C, gás de revestimento nitrogênio. Os experimentos de varredura completa e a fragmentação MS2 foram realizadas por inserção direta utilizando uma bomba de seringa a uma vazão de 15 µL min-1. A varredura foi realizada na faixa de m/z 100-1000 e os íons precursores [M-H]- com intensidades relativas superiores a 10% foram selecionados para a fragmentação MS2. A energia de colisão relativa foi padronizada em 30 V. Análise quimiométrica de dados PCA foi realizada usando o software ChemoFace. As intensidades relativas normalizadas dos íons sugeridos nas partes vegetais constituíram o conjunto de dados multivariados que foi interpretado pela PCA. O pré-processamento dos dados foi realizado por meio do auto-escalonamento por norma suprema, com o objetivo de reduzir a influência de variações indesejáveis no conjunto de dados. Foi criada uma matriz de dados (3 linhas - 16 colunas), 3 linhas correspondentes às 3 partes vegetais (folhas, galhos e cascas) e 16 colunas correspondentes às 16 substâncias sugeridas. A PCA mostrou a distribuição das partes vegetais e a importância das variáveis.
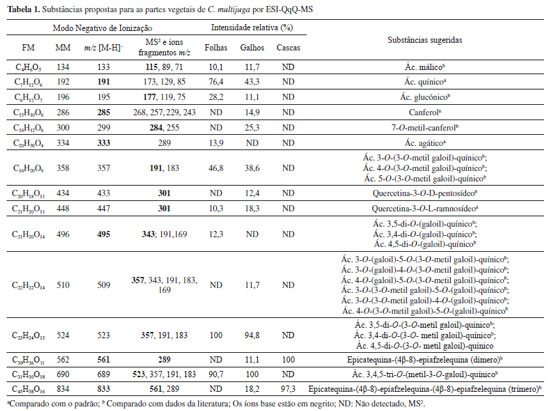
RESULTADOS E DISCUSSÃO Os extratos das folhas, galhos e cascas de C. multijuga obtidos em metanol contendo misturas complexas das substâncias polares foram investigados. Considerando as classes fenólicas de interesse, o modo negativo de ionização em ESI-EM proporcionou resultados abrangentes com alta seletividade e sensibilidade para o perfil metabólico das partes vegetais, compreendendo uma grande variedade de substâncias fenólicas, pois são facilmente desprotonadas devido ao caráter ácido e transferidas para a fase gasosa como íons [M-H]-.15 Os perfis metabólicos foram similares entre os extratos das folhas e dos galhos e ambos foram diferentes do perfil do extrato das cascas da C. multijuga. Os principais íons precursores observados nos extratos das folhas, galhos e cascas de C. multijuga foram fragmentados e os perfis de fragmentação foram confrontados com dados da literatura e com os padrões de referência. Perfis de fragmentação MS2 das substâncias detectadas nas partes vegetais A partir dos perfis de fragmentação obtidos por ESI-QqQ-MS foram sugeridos ácidos galoilquínicos, flavonóides e um diterpeno na composição química dos extratos das folhas e galhos, e no extrato das cascas a presença dos taninos condensados. Os dados da fragmentação MS2 dos íons sugeridos estão dispostos da Tabela 1.
Os íons precursores m/z 133, 195 [M-H]-, corresponderam às formas desprotonadas do ácido málico (MM: 134) e ácido glucónico (MM: 196). A fragmentação MS2 do íon precursor m/z 133 induziu a formação do íon fragmento m/z 115 [M-H2O-H]- resultado da perda de H2O. O íon fragmento m/z 71 [M-H2O-CO2-H]- surgiu da perda simultânea de H2O e CO2 e a descarboxilação direta do íon precursor gerou o íon fragmento m/z 89 [M-CO2-H]-.17 A fragmentação MS2 do íon precursor m/z 195 levou ao íon fragmento m/z 177 [M-H2O-H]- resultado da perda de H2O. Outros íons fragmentos característicos foram detectados, como os íons m/z 119 [M-C2H4O3-H]- e 75 [M-C4H8O4-H]- provenientes do Rearranjo de McLafferty a partir do íon precursor.18 O ácido málico (m/z 133) e o ácido glucónico (m/z 195) foram detectados nas folhas e nos galhos de C. multijuga. O ácido málico e o ácido glucónico estão entre os principais ácidos orgânicos encontrados nas espécies leguminosas, são mais precisamente encontrados nas folhas e frutos e são utilizados como aditivos alimentares e para fins farmacológicos.19,20 O íon precursor m/z 285 [M-H]- correspondeu ao flavonoide canferol (MM: 286) e o íon precursor 299 [M-H]- ao flavonoide 7-O-metil-canferol (MM: 300), ambos foram detectados apenas nos galhos de C. multijuga. A fragmentação MS2 do íon precursor m/z 285 resultou nos íons fragmentos m/z 257 [M-CO-H]- e que originou-se da perda de CO, o íon fragmento m/z 229 [M-2CO-H]- que resultou da segunda perda consecutiva de CO e o íon fragmento m/z 268 [M-OH-H]- como resultado da perda do radical •OH a partir do íon precursor (m/z 285).21 A fragmentação MS2 do íon precursor m/z 299 resultou na perda proeminente do radical •CH3 gerando o íon fragmento m/z 284 [M-CH3-H]- e a perda consecutiva do radical •CHO gerou o íon fragmento m/z 255 [M-CH3-CHO-H]-.21 Os íons precursores m/z 433 e 447 [M-H]- estão relacionados às formas desprotonadas de dois glicosídeos derivados da aglicona quercetina. A fragmentação MS2 dos íons precursores m/z 433 e 447 [M-H]- resultaram em um íon fragmento comum m/z 301, característico da unidade de aglicona quercetina. O íon precursor m/z 433 ao fragmentar perdeu um grupo neutro característico de pentosídeo (-133 U), o qual sugeriu-se a quercetina-3-O-L-pentosídeo (MM: 434),22 sendo detectado apenas nos galhos de C. multijuga. A fragmentação MS2 do íon precursor m/z 447 [M-H]- revelou a perda de um grupo de ramnosídeo (-147 U), logo sugeriu-se a quercetina-3-O-L-ramnosídeo.22 A quercetina-3-O-L-ramnosídeo também foi identificada nas folhas de C. langsdorffii em diversos relatos na literatura, sustentando a presença dos demais derivados da aglicona como a quercetina-3-O-L-pentosídeo identificada na C. multijuga.6-9 Os flavonóides são importantes metabólitos secundários e desempenham nas plantas a função de proteger contra raios ultravioleta, insetos, fungos, vírus e bactérias.23 São potenciais agentes terapêuticos e antioxidantes e exercem atividades antitumorais, anti-inflamatórias, antivirais, além de agir nos sistemas imunológico, circulatório, cardiovascular e nervoso.23,24 As substâncias detectadas nos extratos de Copaifera multijuga (canferol, 7-O-metil-canferol, quercetina-3-O-L-pentosídeo e quercetina-3-O-L-ramnosídeo) podem estar associadas às propriedades terapêuticas dos chás em usos medicinais dessa planta, que incluem o tratamento de enfermidades e disfunções gástricas. O íon precursor m/z 333 [M-H]- foi atribuído à forma desprotonada do diterpeno ácido ent-agático (MM: 334) em comparação com o padrão. O principal fragmento detectado em MS2 foi o íon m/z 289 [M-CO2-H]- decorrente da perda de CO2. O ácido ent-agático faz parte da composição química do óleo-resina da C. langsdorffii,25 C. paupera,26 C. reticulata.27 Este ácido diterpênico se destaca pelas atividades antileishmanial,28 e anti-trypanosoma.29 Os íons precursores m/z 191, 357, 495, 509, 523 e 689 [M-H]- foram atribuídos ao ácido quínico (MM:192) e os derivados ésteres do ácido quínico com os ácidos gálico e 3-O-metil-gálico, cujos serão considerados como ácidos galoilquínicos e foram detectados nos extratos das folhas e galhos de C. multijuga. Os ácidos galoilquínicos possuem como núcleo principal o ácido quínico, que pode ser esterificado por uma ou mais unidades de ácido gálico e/ou ácido 3-O-metil-gálico.5 As diferenças observadas nos perfis de fragmentação dos íon precursores originaram-se das variações nas perdas dos grupos galoil e/ou 3-O-metil-galoil ligados ao ácido quínico. Alguns íons detectados podem estar relacionados as diferentes séries dos ácidos galoilquínicos mono-, di- e tri-substituídos. A partir dos experimentos de fragmentação MS2 e as limitações do analisador QqQ, não foi possível estabelecer a caracterização completa dos ácidos galoilquínicos, uma vez que existem isômeros específicos para cada íon detectado de acordo com a Tabela 1.30 O íon precursor m/z 191 [M-H]- correspondeu à forma desprotonada do ácido quínico (MM: 192), sendo um importante precursor na biossíntese dos ácidos fenólicos. A fragmentação MS2 do íon precursor resultou no íon fragmento m/z 173 [M-H2O-H]- relacionado à perda de H2O. O íon fragmento m/z 129 [M-C2H2O-H]- resultou da segmentação retro Diels-Alder e a perda sequencial de CO2 resultou no íon fragmento m/z 85 [M-C2H2O-CO2-H]-.31 Já o íon precursor m/z 357 [M-H]- pode estar relacionado com a forma desprotonada do ácido 3-O-(3-O-metil-galoil)-quínico (estrutura 1, Figura 1). A fragmentação MS2 do íon precursor m/z 357 levou ao reconhecimento de íons fragmentos característicos, como o íon m/z 191 [M-C8H6O4-H]- correspondendo a forma desprotonada ácido quínico decorrente da perda do grupo 3-O-metil-galoil (-166 Da) e o íon m/z 183 [M-C7H11O5-H]- associado a forma desprotonada do ácido 3-O-metil-gálico decorrente da perda do radical quínico (-174 Da).5,30
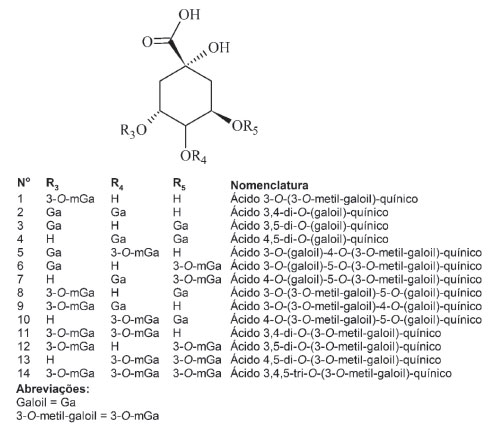 Figura 1. Derivados do ácido quínico detectados nos extratos de C. multijuga
O íon precursor m/z 495 [M-H]- pode estar associado às formas desprotonadas dos três possíveis isômeros dos ácidos di-(galoil)-quínicos (Estruturas 2-4, Figura 1).5,30 A partir da fragmentação MS2 do íon precursor m/z 495 foi possível inferir duas perdas consecutivas de radicais galoil, o íon fragmento m/z 343 [M-C7H5O4-H]- resultou da perda de um radical galoil (-152 Da) e o íon fragmento m/z 191 [M-2C7H5O4-H]- resultou da perda do segundo radical galoil (-304 Da).5,30 A detecção do íon fragmento m/z 169 está associada a forma desprotonada do ácido gálico. O íon precursor m/z 509 [M-H]- pode estar relacionado às formas desprotonadas dos seis possíveis isômeros dos ácidos (galoil)-(metil-galoil)-quínicos (Estruturas 5-10, Figura 1). A partir da fragmentação MS2 do íon precursor m/z 509 foi possível observar duas perdas características, o íon fragmento m/z 357 [M-C7H5O4-H]- resultou da perda do radical galoil (-152 Da) e o íon fragmento m/z 343 [M-C8H7O5-H]- resultou da perda do radical 3-O-metil-galoil (-166 Da).5,30 Outros íons fragmentos característicos foram detectados, como o íon m/z 183 correspondendo à forma desprotonada do ácido 3-O-metil-gálico, o íon m/z 169 ao ácido gálico desprotonado e o íon m/z 191 ao ácido quínico desprotonado. O íon precursor m/z 523 [M-H]- pode estar relacionado às formas desprotonadas dos três possíveis isômeros dos ácidos di-(metil-galoil)-quínicos (Estruturas 11-13, Figura 1). A partir da fragmentação MS2 do íon precursor m/z 523 foi possível observar duas perdas consecutivas de radicais 3-O-metil-galoil, o íon fragmento m/z 357 [M-C8H7O5-H]- resultou de uma perda de radical 3-O-metil-galoil (-166 Da) e o íon m/z 191 [M-2C8H7O5-H]- resultou da perda do segundo radical 3-O-metil-galoil (-166 Da).5,30 O íon fragmento m/z 183 pode estar relacionado a detecção da forma desprotonada do ácido 3-O-metil-gálico. O íon precursor m/z 689 [M-H]- pode estar relacionado ao ácido 3,4,5-tri-O-(3-O-metil-galoil)-quínico desprotonado (Estrutura 14, Figura 1). A fragmentação MS2 do íon precursor m/z 689 resultou em duas perdas de radicais 3-O-metil-galoil (-166 Da), a primeira perda formou o íon fragmento m/z 523 [M-C8H7O5-H]- e a segunda perda o íon fragmento m/z 357 [M-2C8H7O5-H]-.5,30 Outros fragmentos importantes foram detectados, como o íon fragmento m/z 183 e 191 que podem corresponder as formas desprotonadas dos ácidos 3-O-metil-gálico e ácido quínico, respectivamente. Os ácidos galoilquínicos sugeridos foram identificados, anteriormente, nos extratos polares das folhas de C. langsdorffii.5 Os extratos polares e os ácidos galoilquínicos isolados apresentaram atividades de gastroproteção em modelos de úlceras induzidas por etanol-HCl em camundongos.12 Os ácidos galoilquínicos reduziram significativamente a área total da lesão e aumentaram a taxa de cura em comparação com o pantoprazol e apresentaram citotoxicidade contra células de adenocarcinoma gástrico.12 Logo, os ácidos galoilquínicos sugeridos para a espécie de C. multijuga abrem novas perspectivas sobre o potencial químico e farmacológico da espécie, além de viabilizar a obtenção de novos marcadores químicos para a espécie e o gênero Copaifera. Os íons precursores de m/z 833 e 561 [M-H]- corresponderam às formas desprotonadas dos taninos condensados epicatequina-(4β-8)-epiafzelequina (dímero) e epicatequina-(4β-8)-epiafzelequina-(4β-8)-epiafzelequina (trímero), respectivamente. A fragmentação MS2 do íon precursor m/z 561 levou ao íon fragmento m/z 289 [M-C15H14O5-H]- resultado da perda de um monômero de epiafzelequina (-274 Da).12 A fragmentação MS2 do íon precursor m/z 833 resultou em perdas consecutivas de dois monômeros de epiafzelequina gerando o íon fragmento m/z 561 [M-C15H14O5-H]- e o m/z 289 [M-2C15H14O5-H]-.12 O íon fragmento m/z 289 detectado em ambos precursores pode estar relacionado à forma desprotonada da epicatequina. Os taninos condensados apresentaram boa ionização em modo negativo e foram detectados em altas intensidades nos extratos das cascas de C. multijuga, tais substâncias podem estar relacionadas às propriedades fitoterápicas dos chás das cascas na medicina popular, visto que os extratos possuem propriedades antioxidantes relatadas frente aos testes in vitro e in vivo com redução do estresse oxidativo, além do potencial efeito antineoplásico em células tumorais de Ehrlich.1 Análise dos Componentes Principais (PCA) A PCA da intensidade relativa dos íons sugeridos (Tabela 1) representou uma variância explicada dos dados nas componentes principais 1 e 2 de 61,35% e 32,75%, respectivamente (Figura 2). Por esta análise é possível observar que as amostras do extrato das cascas se separam das demais partes vegetais devido à presença de taninos condensados nas formas de dímero epicatequina-(4β-8)-epiafzelequina (m/z 561) e trímero epicatequina-(4β-8)-epiafzelequina-(4β-8)-epiafzelequina (m/z 833), detectados apenas nas cascas.
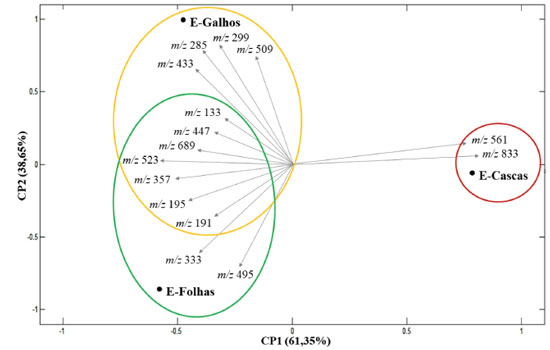 Figura 2. Gráfico de biplot: extrato das folhas (E-Folhas); extrato dos galhos (E-Galhos) e extrato das cascas (E-Cascas) de C. multijuga
Os extratos das folhas e dos galhos foram separados ao longo de CP2, as substâncias detectadas unicamente em cada parte vegetal foram cruciais para a distinção das partes: nas folhas o ácido ent-agático (m/z 333) e os ácidos di-(galoil)-quínicos, relacionados ao íon m/z 495 foram substâncias características (Estruturas 2-4, Figura 1); e nos galhos o canferol (m/z 285), 7-O-metil-canferol (m/z 299), quercetina-3-O-L-pentosídeo (m/z 433) e os seis ácidos (galoil)-(metil-galoil)-quínicos (Estruturas 5-10, Figura 1), associados ao íon m/z 509, foram característicos. Apesar da distinção entre folhas e galhos, ambas amostras compartilharam perfis metabólicos similares, ou seja, o ácido málico (m/z 133), ácido quínico (m/z 191), ácido glucônico (m/z 195), quercetina-3-O-L-ramnosídeo (m/z 447), e os ácidos galoilquínicos representados pelos íons m/z 357, 523 e 689 (Tabela 1) foram substâncias detectadas em ambas as partes vegetais.
CONCLUSÃO A técnica de fingerprint por inserção direta em espetrômetro de massas (ESI-QqQ-EM) possibilitou a distinção dos extratos dos três órgãos vegetais de C. multijuga. Foi possível realizar a identificação putativa de 26 substâncias por meio de experimentos de fragmentação MS2 dos principais íons detectados. Este é o primeiro relato dos compostos identificados nos extratos de folhas e galhos de C. multijuga, destacando-se os derivados do ácido quínico e flavonóides. Taninos condensados foram detectados unicamente no extrato das cascas de C. multijuga, podendo ser utilizados como seus marcadores químicos.
AGRADECIMENTOS Ao CNPq e à CAPES pelas bolsas concedidas. Ao INPA pelo apoio na coleta e identificação do material vegetal e à Central Analítica Multidisciplinar da UFAM pelas análises espectrométricas.
REFERÊNCIAS 1. Brun, H. D.; Camargo, J. L. C.; Ferraz, I. D. K. Copaíba-roxa, Copaifera multijuga Hayne; Ferraz, I. D. K.; Camargo, J. L. C., eds.; Manual de Sementes da Amazônia: Manaus, 2009, Fascículo 9; Patitucci, M. L.; Veiga Junior, V. F.; Pinto, A. C.; Zogbi, M. G. B.; Silva, J. R. D.; Quim. Nova 1995, 18, 262; Barbosa, P. C. S.; Medeiros, R. S.; Sampaio, P. T. B.; Vieira, G.; Wiedemann, L. S. M.; Veiga-Junior, V. F.; J. Braz. Chem. Soc. 2012, 23, 1823; Barbosa, P. C. S.; Wiedemann, L S. M.; Medeiros, R. S.; Sampaio, P. T. B.; Vieira, G.; Veiga-Junior, V. F.; Chem. Biodivers. 2013, 10, 1350. 2. Arruda, C.; Mejía, J. A. A.; Ribeiro, V. P.; Borges, C. H. G.; Martins, C. H. G.; Veneziani, R. C. S.; Ambrosío, S. R.; Bastos, J. K.; Biomed. Pharmacother. 2019, 109, 1. 3. Trindade, R.; Silva, J. K.; Setzer, W. N.; Int. J. Mol. Sci. 2018, 19, 1511; Vargas, F. S.; Almeida, P. D. O.; Aranha, E. S. P.; Boleti, A. P. A.; Newton, P.; Vasconcellos, M. C.; Junior, V. F. V.; Lima, E. S.; Molecules 2015, 20, 6194. 4. Vásquez, S. P. F.; Medonça, M. S.; Noda, S. N.; Acta Amazonica 2014, 44, 457; Leandro, Y. A. S.; Jardim, I. N.; Gavilanes, M. L.; Biodiversidade 2017, 16, 30. 5. Motta E. V.S.; Lemos, M.; Costa, J. C.; Bandero-Filho, V. C.; Sasse, A.; Sheridan H.; Bastos, J. K.; Chem-Biol. Interact. 2017, 261, 145. 6. Lemos M.; Santin, J. R.; Mizuno, C. S.; Bocing, T.; Sousa, J. P. B.; Nanayakkara, D.; Bastos, J. K.; Andrade, S. F.; Rev. Bras. Farmacogn. 2015, 25, 238. 7. Costa, A. R. M.; Freitas, L. A. P.; Mendiola, J.; Ibáñez, E.; J. Supercrit. Fluid. 2015, 100, 86. 8. Furtado, R. A.; Bernades, C. T. V.; Silva, M. N.; Zoccal, K.F.; Faccioli, L. H.; Bastos, J. K.; Biomed. Res. Int. 2015, 2015, 1. 9. Nogueira, M. S.; Furtado, R. A.; Bastos, J. K.; J. Agric. Food Chem. 2015, 63, 6939. 10. Batista, A. G.; Ferrari, A. S.; Cunha, D. C.; Silva, J. K.; Cazarin, C. B. B.; Correa, L. C.; Prado, M. A.; Silva, L. B. C.; Esteves, E. A.; Júnior, M. R. M.; Food Chem. 2016, 197, 1153. 11. Neto, J. S. L.; Gramosa, N. V.; Silveira, E. R.; Quim. Nova 2008, 31, 1078. 12. Albiero, L. R.; Nery, E. F.; Dalazen, J. C.; Kelly, T. O.; Pereira, D. L.; Sinhorin, V. D. G.; Kameno, R.; Castoldi, L.; J. Pharm. Biol. Sci. 2016, 11, 30.; Cunha, A. P. S.; Baldissera, L.; Pereira, D. L.; Albiero, L. R.; Castoldi, L.; Sinhorin, A. P.; Sinhorin, V. D. G.; Acta Amazonica 2018, 48, 347; Pereira, D. L.; Cunha, A. P. S.; Cardoso, C. R. P.; Rocha, C. Q.; Vilegas, W.; Sinhorin, A. P.; Sinhorin, V. D. G.; Acta Amazonica 2018, 48, 347. 13. Ye, Z.; Dai, J. R; Zhang, C. G.; Lu, Y.; Wu, L. L.; Gong, A. G. W.; Xu, H.; Tsim, K. W. K.; Wang, Z. T.; J. Evidence-Based Complementary Altern. Med. 2017, 2017; Quirós-Guerrero, L. Albertazzi, F.; Araya-Valverde, E.; Romero, R. M.; Villalobos, H.; Poveda, L.; Chavarría, M.; Tamayo-Castillo, G.; Metabolomics 2019, 15, 14; Taamalli, A.; Román, D. A.; Gómez, C.; Zarrouk, M.; Carretero, A. S.; J. Anal. Methods Chem. 2018, 2018. 14. Niculau, E. S. N. Freitas, S. D. L.; Sá, I. C. G.; Fernandes, J. B.; Silva, M. F. G. F.; Rev. Virtual Quim. 2016, 8, 204.; Mari, A.; Montoro, P.; D'Urso, G.; Macchia, M.; Pizza, C.; Piacente, S.; J. Pharm. Biomed. Anal. 2015, 102, 215; Klockmann, S.; Reiner, E.; Cain, N.; Fischer, M.; J. Agric. Food Chem. 2017, 65, 1456. 15. Wolfender, J. L.; Glauser, L.; Boccard J.; Raduz, S.; Nat. Prod. Commun. 2009, 10, 1417. 16. Hongry, K.; Sadanielo, V. L. M.; Junior, G. J. O.; Eng. Sci. 2015, 1, 83. 17. Roesler, R.; Catharino, R. R.; Malta, L. G.; Eberlin, M. N.; Pastore, G.; Food Chem. 2007, 104, 1048; Fernández-Fernández, R.; López-Martínez, J.; Romero-González, R.; Martínezz-Vidal, J. L.; Flores, M. I. A.; Frenich, A. G.; Chromatographia 2010, 72, 55. 18. Cádiz-Gurrea, M. L. L.; Fernández-Arroyo, S.; Joven, J.; Segura-Carretero, A.; Food Res. Int. 2013, 50, 197; Felipe, D. F.; Branbilla, L. Z. S.; Porto, C.; Pilau, E. J.; Cortez, D. A. G.; Molecules 2014, 19, 15720; Grossert, J. S.; Cook, M. C.; White, R. L.; Rapid Comm. Mass Spectrom. 2006, 20, 1511. 19. Ceylan, R.; Katanic, J.; Zengin, G.; Matic, S.; Aktumsek, A.; Boroja, T.; Stanic, S. Mihailovic, V.; Guler, G. O.; Boga, M.; Yilmaz, M. A.; Ind. Crops Prod. 2016, 84, 254. 20. Cañete-Rodríguez, A. M.; Santos-Dueñas, I. M.; Jiménez-Hornero, J. E.; Ehrenreich, A.; Liebl, W.; García-García, I.; Process Biochem. 2016, 51, 1891. 21. Justesen, U.; J. Mass Spectrom. 2001, 36, 169 March, R. E.; Miao, X. S.; J. Mass Spectrom. 2004, 231, 157. 22. Mercolini, L.; Protti, M.; Saracino, M. A.; Mandrone, M.; Antognoni, F.; Poli, F.; Phytochem. Anal. 2015, 27, 41. 23. Agrawal, A. D.; Int. J. Pharm. Sci. Nanotech. 2011, 4, 1394; Oliveira, T. T. D.; Gomes, S. M.; Nagem, T. J.; Costa, N. M. B.; Secom, P. R.; Rev. Nutr. 2002, 15, 45; Heim, K. E.; Tagliaferro, A. R.; Bobilya, D. J. J. Nutr. Biochem. 2002, 13, 572; Shashank, K.; Abhay, K.; Sci. World J. 2013, 4, 32. 24. Georgiev, V.; Ananga, A.; Tsolova, V.; Nutrients 2014, 6, 391. 25. Souza, A. B.; Martins, C. H. G.; Souza, M. G. M.; Furtado, N. A. J. C.; Heleno, V. C. G.; Sousa, J. P. B.; Rocha, E. M. P.; Bastos, J. K.; Cunha, W. R.; Veneziani, R. C. S.; Phyther. Res. 2011, 25, 215. 26. Tincusi, B. M.; Jiménez, I. A.; Bazzocchi, I. L.; Moujir, L. M.; Mamani, Z. A.; Barroso, J. P.; Ravelo, A. G.; Hernández, B. V.; Planta Med. 2002, 68, 808. 27. Geris, R.; Silva, I. G.; Silva, H. H. G.; Barison, A.; Rodrigues-Filho, E.; Ferreira, A. G.; Rev. Inst. Med. Trop. São Paulo, 2008, 50, 25. 28. Santos, A. O.; Izumi, E.; Ueda-Nakamura, T.; Dias-Filho, B. P.; da Veiga-Júnior, V. F.; Vataru-Nakamura, C.; Mem. Inst. Oswaldo Cruz 2013, 108, 59. 29. Izumi, E.; Ueda-Nakamura, T.; Veiga, V. F.; Pinto, A. C.; Nakamura, C. V.; J. Med. Chem. 2012, 55, 2994. 30. Sannomiya, M.; Santos, L. C.; Carbone, V.; Napolitano, A.; Piacente, S.; Pizza, C.; Souza-Brito, A. R. M.; Vilegas, W.; Rapid Commun. Mass Spectrom. 2007, 21, 1393. 31. Parveen, I.; Threadgill, M. D.; Hauck, B.; Donnison, I.; Winters, A.; Phytochemistry 2011, 72, 2376.; Bastos, D. H. M.; Saldanha, L. A.; Catharino, R. R.; Sawaya, A. C. H. F.; Cunha, I. B. S.; Carvalho, P. O.; Eberlin, M. N.; Molecules 2007, 12, 423. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access