Artigo
|
|
| Padronização da quantificação de glutationa redutase em Aegla singularis (Anomura, Crustacea) utilizando planejamento experimental DCCR Standardization of glutathione reductase measure in Aegla singularis (Anomura, Crustacea) using dccr experimental planning |
|
Sabrina M. AlbaniI; Ana P. BorgesI; Monik C. MartinsI; Rozane M. RestelloI; Fernanda D. CameraII; Natália ParoulIII; Rogério L. CansianIII; Albanin A. Mielniczki-PereiraI,*
I. Departamento de Ciências Biológicas, Universidade Regional Integrada do Alto Uruguai e das Missões, 99709-910 Erechim - RS, Brasil Recebido em 19/01/2020 *e-mail: albanin@uricer.edu.br Glutathione Reductase (GR) plays a key role in the maintenance of GSH homeostasis, variations in its activity may indicate damage from environmental contaminants. Experimental planning is a strategy to reduces time and cost in enzymatic assays. In this work, DCCR experimental planning was used to standardize and to validate the measure of GR in Aegla singularis bioindicator. Samples of A. singularis were collected in three watersheds with different percentages of naturalness and land uses. At all, were selected 7 streams, in which collections were performed at one upstream (most preserved) and one downstream (less preserved) point. Effects of temperature, reading time, substrate (GSSG) and protein concentration in GR reaction medium were evaluated, being that the two latter variables were optimized by DCCR. GR was not affected by temperature (15 to 30 °C) or reading time (6 and 10 minutes). Increased concentrations of proteins from biological extract had negative correlation with GR, whereas higher concentrations of GSSG resulted in higher GR activity. Environmental validation showed that GR in Aegla was influenced primarily by the watershed of origin. DCCR planning was efficient to optimizing the use of GR as a biomarker of environmental quality. INTRODUÇÃO Contaminantes ambientais podem afetar os organismos em diferentes níveis da estrutura biológica, como alterações em populações, comunidades ou alterações fisiológicas e bioquímicas de organismos individuais.1 Os biomarcadores de estresse oxidativo são ferramentas bastante utilizadas em ensaios bioquímicos de ecotoxicologia. Esses marcadores fornecem medidas robustas, pois respondem a diferentes poluentes individuais ou misturas de xenobióticos comumente encontrados em ambientes naturais.2 A enzima Glutationa Redutase (GR) é um biomarcador que catalisa a redução de glutationa oxidada (GSSG) a glutationa reduzida (GSH), utilizando NADPH como cofator.3 A GR tem papel fundamental para a reciclagem e manutenção da homeostase de GSH, especialmente em situações de exposição a xenobióticos que produzem estresse oxidativo ou que são desintoxicados em reações que utilizam GSH.4 Análises realizadas in situ ou em laboratório mostram que a atividade e/ou a expressão da GR é influenciada pela presença de medicamentos, pesticidas e metais em organismos bioindicadores como bivalves,5 peixes6 e caranguejos.7-9 Na maioria dos estudos ecotoxicológicos, a atividade da GR é determinada pelo monitoramento do consumo de NADPH a 340 nm,10,11 ou pelo aumento da absorbância a 412 nm que ocorre quando o DTNB (ácido 5,5´ dithiobis (2-nitrobenzoico)) é reduzido por GSH.12,13 A medida da GR também pode ser obtida por reação com corantes fluorescentes de alta especificidade.14 Entretanto, o uso desse sistema é menos difundido na literatura. A avaliação da GR e de outros biomarcadores em organismos bioindicadores geralmente é feita a partir da adaptação direta de metodologias descritas para outras espécies, não sendo comum a aplicação de planejamento experimental para a otimização dos ensaios. O planejamento experimental (ou design de experimentos) consiste na aplicação de análises estatísticas a priori, visando definir o efeito que variáveis conhecidas exercem sobre uma resposta de interesse. Além de reduzir o tempo e o custo da etapa experimental, esta metodologia aumenta a confiabilidade nos resultados obtidos. O Delineamento Composto Central Rotacional (DCCR) é utilizado quando se deseja avaliar o efeito de um número reduzido de variáveis previamente reconhecidas como significativas (preferencialmente).15 O planejamento experimental vem sendo aplicado em estudos ambientais que visam aprimorar métodos de tratamento de efluentes de diferentes origens,16-18 entretanto, sua aplicação em análises bioquímicas com organismos bioindicadores é praticamente inexistente. Crustáceos do gênero Aegla são bioindicadores sensíveis às variações ambientais.19,20 Embora os dados de literatura sobre biomarcadores de estresse oxidativo para o gênero sejam escassos, sabe-se que os níveis basais de peroxidação lipídica e atividade da enzima catalase variam entre machos e fêmeas, apresentando ainda oscilação sazonal.21 Metalotioneínas, sequestradores de radicais peroxil e concentração total de GSH também variam entre diferentes espécies do gênero. Por outro lado, a proporção GSH/GSSG, um indicador de capacidade antioxidante geral, é mais estável entre as espécies do que a concentração bruta de GSH.20 O objetivo deste trabalho foi utilizar o planejamento experimental DCCR para padronizar e otimizar a metodologia de medida da GR em Aegla singularis (A. singularis). Além disso, após a padronização, o método foi testado com exemplares de Aegla coletados em Bacias Hidrográficas com diferentes percentuais de naturalidade.
PARTE EXPERIMENTAL Área de estudo e coleta de organismos As coletas foram realizadas em três Bacias Hidrográficas (BH) localizadas no município de Erechim-RS, Brasil. Foram selecionados 7 riachos < 3ª ordem, sendo 3 localizados na BH do Rio Suzana, 2 na BH do Rio Ligeirinho-Leãozinho e 2 na BH do Rio Dourado (Figura 1). Essas BH diferem em relação ao percentual de uso e cobertura do solo, a saber: BH Suzana 64,65% de ocupação antrópica e 35,35% de naturalidade;22,23 BH Ligeirinho-Leãozinho 73,70% de ocupação antrópica e 26,30% de naturalidade;24 BH Dourado 41,27% de ocupação antrópica e 58,73% de naturalidade.25 Em cada riacho, os organismos foram amostrados em um trecho a montante (em direção à nascente) e outro a jusante (em direção à foz), sendo que os trechos a montante são pontualmente mais preservados (na área periférica imediata do trecho) do que os trechos a jusante, de acordo com o protocolo descrito por Callisto et al.26 Esse protocolo se baseia na avaliação de 22 parâmetros que são indicadores de (i) características dos trechos de bacia e nível de impacto antrópico e (ii) condições de habitat e nível de conservação das condições naturais.26
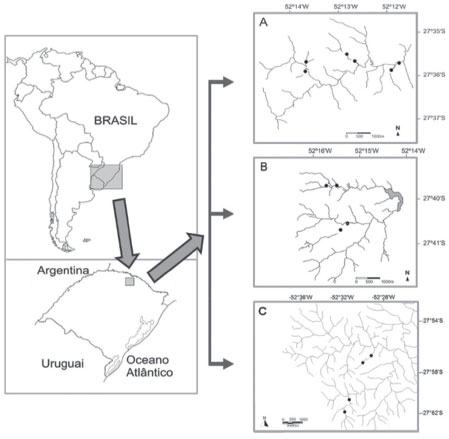 Figura 1. Mapa dos locais de coleta. A) BH do Rio Suzana; B) BH do Rio Ligeirinho-Leãozinho; C) BH do Rio Dourado
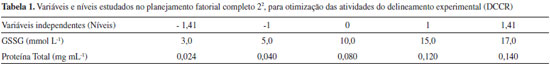
Exemplares de A. singularis foram coletados com rede do tipo puçá de 30 x 50 cm de boca, com profundidade de 60 cm e malha com abertura de 1,0 mm. Apenas fêmeas, adultas (com no mínimo 15 mm de comprimento de cefalotórax), não ovígeras, foram incluídas na coleta. A seleção do gênero foi baseada em estudo prévio21 que mostrou que as fêmeas não apresentam variação sazonal em biomarcadores de estresse oxidativo, ao contrário do que ocorre com os machos. A identificação de sexo e espécie foi feita a campo, de acordo com protocolo descrito por Melo.27 Foram coletados 3 organismos por trecho de coleta (Figura 1), durante o período de primavera, totalizando 18 organismos na BH do Rio Suzana, 12 na BH do Rio Ligeirinho-Leãozinho e 12 na BH do Rio Dourado. Após a captura, os organismos foram transportados vivos, em frascos contendo água do próprio local de coleta, até o laboratório aonde foram processados para obtenção do extrato biológico. O tempo entre a coleta e o preparo dos extratos foi de aproximadamente 20 minutos. Em todos os dias de coleta, a temperatura da água foi avaliada utilizando-se um termômetro digital (Incoterm). Preparo dos extratos biológicos O preparo dos extratos biológicos foi feito conforme descrito por Borges et al.21 Visando uma análise mais robusta e de mais fácil aplicação em estudos de biomonitoramento, os extratos biológicos foram obtidos a partir de organismos inteiros, triturados em cadinho gelado na presença de tampão fosfato e PMSF (inibidor de protease), sendo feito o descarte do exoesqueleto. Em seguida, o material biológico foi centrifugado (1600 x g, 30 min, 4 °C) e o sobrenadante foi congelado a -20 °C para ser utilizado na determinação de proteínas totais pelo método de Bradford28 e para determinação da atividade da enzima GR. Padronização do método de quantificação da Glutationa Redutase (GR) A quantificação de GR foi adaptada a partir do protocolo descrito por Ramos-Vasconcelos e Hermes-Lima.11 A base do método é a medida espectrofotométrica do consumo de NADPH (340 nm). As reações foram realizadas na presença de tampão fosfato pH 7,0 (50 mmol L-1 de Na2HPO4; 0,5 mmol L-1 de EDTA), GSSG (5 mmol L-1), NADPH (0,25 mmol L-1) e extrato biológico (concentração inicial de 0,040 mg mL-1 de proteína). Foram testados dois tempos de reação cinética, 6 e 10 minutos. Estes tempos foram estabelecidos experimentalmente por apresentarem padrão de linearidade na reação da GR em nossas condições de estudo. Nos dois casos, na primeira metade do tempo (3 ou 5 minutos, respectivamente) foi realizada a leitura do controle (branco) com tampão, NADPH e extrato. Da metade do tempo em diante, foi realizada a medida efetiva da GR, iniciada pela adição de GSSG na reação. Também foram testadas reações em diferentes temperaturas (15, 20 e 30 °C). As análises bioquímicas foram realizadas, no mínimo, em dois experimentos independentes, com triplicata (ou quadruplicata) de leituras em cada dia experimental. Os valores da atividade da GR foram expressos em unidades internacionais (U) e calculados conforme a Equação 1: 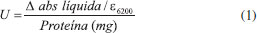 em que: U = μmol de NADPH consumido min-1 mg proteína-1; Δ abs. líquida = (Δ abs tempo total de leitura) - (Δ abs tempo de leitura do branco); ε = coeficiente de extinção molar do NADPH, no comprimento de onda de 340 nm; Proteína = conteúdo de proteína utilizado na reação (mg). A concentração de substrato (GSSG) e de proteínas no extrato biológico ótimas para a dosagem enzimática, foram avaliadas por meio de um delineamento composto central rotacional (DCCR). Para tanto, foi avaliado o efeito da concentração de GSSG na faixa de 3 a 17 mmol L-1 e o efeito da concentração de proteínas totais na faixa de 0,024 a 0,140 mg mL-1. Nessa avaliação foi utilizado como controle o tampão fosfato, extrato biológico e NADPH, com tempo de reação de 6 minutos e temperatura de 20 °C. A Tabela 1 apresenta os níveis de variáveis independentes investigadas no DCCR.
Análise dos dados A diferença entre a atividade da GR nos tempos de 6 e 10 minutos de reação foi analisada por teste-t não pareado. A comparação da atividade da GR nas diferentes temperaturas foi feita por ANOVA um fator seguida de teste de Tukey. Os resultados relativos à atividade da GR (i) em função das concentrações de GSSG e proteína total obtidos pelo planejamento experimental e (ii) em função da BH e dos trechos de coleta foram avaliados por ANOVA dois fatores. A comparação específica entre atividade da GR em organismos coletados nos trechos a montante e a jusante também foi feita utilizando-se teste t não pareado. A comparação entre os valores gerais de GR e as três BH analisadas foi realizada por ANOVA um fator seguida de teste de Tukey. Valores de p < 0,05 foram considerados significativos.
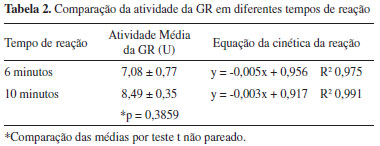
RESULTADOS E DISCUSSÃO Padronização do método de quantificação de GR proveniente de A. singularis A curva cinética da GR apresentou comportamento linear nos dois tempos analisados, não havendo diferença estatística para a atividade da enzima nesses tempos (Tabela 2). Dessa forma, os experimentos seguintes também foram realizados apenas com 6 minutos de reação.
A GR também não foi afetada significativamente pelas diferentes temperaturas de reação testadas, mantendo-se com aproximadamente 100% de atividade na faixa de 15 a 30 °C (Figura 2). Esse dado tem uma consequência experimental interessante, pois indica que, em laboratório, a GR obtida de A. singularis pode ser avaliada em uma faixa de temperatura razoável sem que isso interfira nos valores absolutos a partir de cada organismo individual.
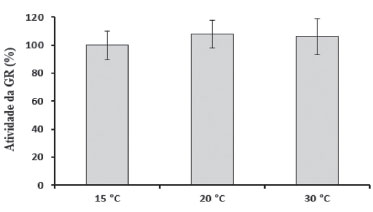 Figura 2. Efeito da temperatura sobre a atividade da GR proveniente de A. singularis. Os dados são apresentados como percentual de atividade da GR em relação à primeira temperatura testada (15 °C) e foram obtidos a partir de dois experimentos independentes (totalizando n = 8 para 15 ºC e n = 10 para 20 e 30 ºC). Os valores brutos (não percentuais) foram avaliados estatisticamente por ANOVA um fator seguida de teste de Tukey e não foram encontradas diferenças estatísticas significativas
A ausência do efeito da temperatura pode estar relacionada com a necessidade de adaptação do organismo ao ambiente natural. A temperatura da água do ambiente de coleta dos exemplares de A. singularis variou entre 15 e 22 °C ao longo do ano. Nesta faixa de temperatura, a GR é capaz de manter sua atividade constante, não comprometendo sua função para manutenção da homeostase de GSH e do sistema de defesa antioxidante. O processo de adaptação de enzimas às variações de temperatura em animais ectotérmicos é descrito na literatura dentro da bioquímica adaptativa.29 No ambiente natural, algumas possíveis estratégias para essa adaptação podem estar relacionadas com: (i) variações na sequência de aminoácidos da proteína resultando em influência sobre parâmetros cinéticos; (ii) mudança na concentração de proteínas mediante regulação da expressão gênica e alterações na taxa de renovação proteica e (iii) variações no ambiente em que a proteína funciona para promover melhores condições de manutenção de sua estabilidade.29 Neste trabalho foram testadas variações de temperatura em condições artificiais, que não permitem mecanismos de adaptação associados ao aumento da concentração proteica ou associados a adaptações internas do organismo que favoreçam a atividade enzimática. Dessa forma, a hipótese mais provável é que a GR de A. singularis seja composta por uma sequência de aminoácidos que permita adaptação em termos de cinética. Entretanto, esta hipótese precisa ser confirmada experimentalmente. A determinação da concentração de substrato (GSSG) e de extrato enzimático ótimos para a determinação da atividade da GR, nas condições maximizadas de tempo de reação (6 minutos) e temperatura (20 °C), foram definidas com base em um planejamento de experimentos. A Tabela 3 apresenta a matriz do planejamento fatorial completo (22) e os resultados da atividade enzimática da GR. Observa-se maior atividade no experimento 5 com a menor concentração de proteína total no extrato biológico (0,024 mg mL-1) e concentração intermediária de substrato (10 mmol L-1 de GSSG).
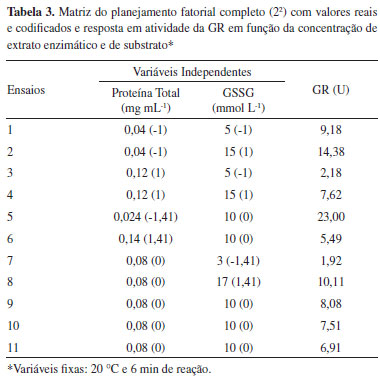
A Figura 3 apresenta o gráfico de Pareto com os efeitos estimados das variáveis estudadas para a atividade da GR, onde observa-se que a concentração de extrato e do substrato tem influência significativa na atividade da enzima. O aumento na concentração de extrato apresentou efeito negativo. Já a concentração de GSSG apresentou efeito positivo, indicando que o aumento deste substrato pode produzir aumento na atividade da GR em Aegla. A interação entre as duas variáveis avaliadas não foi significativa.
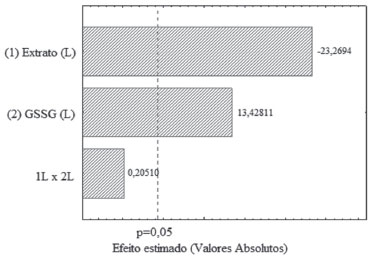 Figura 3. Gráfico de Pareto com o efeito estimado (valor absoluto) das variáveis testadas no planejamento DCCR
A influência da concentração de GSSG sobre a atividade da GR foi confirmada experimentalmente a partir de ensaios nos quais a dosagem de enzima foi realizada na presença de valores fixos de extrato proteico, mas com concentrações variáveis de GSSG (Figura 4). Especificamente, quando a concentração de proteína total no extrato foi fixada em 0,025 mg mL-1, a melhor atividade da GR foi obtida com 20 mmol L-1 de GSSG (29,42 U ± 2,89). Quando foi fixado em 0,010 mg mL-1, a melhor atividade da GR foi obtida com 8 mmol L-1 de GSSG (32,44 U ± 3,03). Comparando-se essas duas condições (0,010 e 0,025 mg mL-1), não houve diferença estatística significativa entre as atividades enzimáticas máximas.
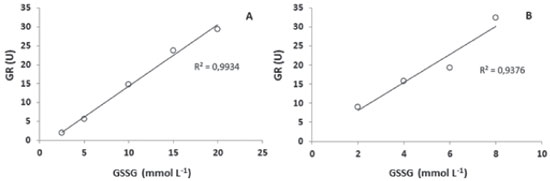 Figura 4. Efeito da concentração de GSSG sobre a atividade da GR utilizando-se 0,025 mg mL-1 (A) ou 0,010 mg mL-1 (B) de proteína total no extrato biológico
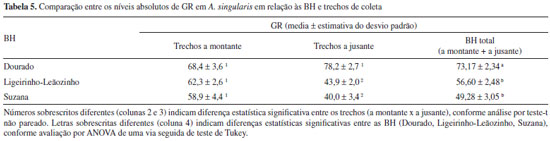
A velocidade das reações enzimáticas é proporcional à concentração dos substratos (reagentes),30 o que está de acordo com o aumento da atividade da GR em função do aumento de GSSG observado neste trabalho (Figuras 3 e 4). Além disso, sabe-se que o mecanismo de reação da GR é do tipo ping-pong.31,32 Na primeira etapa da reação, o NADPH se liga à enzima, que se torna reduzida (GRred) e libera o NADP. Na segunda etapa, a enzima transfere elétrons para a GSSG de maneira sequencial, ocorrendo a liberação de duas moléculas de GSH, uma por vez.32 Acredita-se que no ambiente celular predomina a presença GRred, devido à rápida velocidade de reação com o NADPH.32 A segunda etapa da reação é mais lenta, sendo considerada a fase limitante da reação global.31,33 Nesse sentido, maiores concentrações de GSSG podem descolar o equílibrio de ligação entre a enzima (GRred) e o substrato (GSSG), aumentando a formação de complexos GR(red)-GSSG, o que consequentemente pode resultar em maior velocidade de reação. Não existem estudos específicos de caracterização bioquímica ou molecular da GR (ou de seu gene codificante) em grupos taxonômicos próximos do gênero Aegla. Entretanto, sabe-se que a estrutura primária da enzima é bastante conservada, apresentando cerca de 50% de identidade de aminoácidos entre organismos distantes como Saccharomyces cerevisiae e Homo sapiens.34 Nesse sentido, o aumento proporcional de atividade da GR em função da concentração de GSSG observado para A. singularis está de acordo com resultados de avaliações do perfil cinético da GR proveniente de leveduras e humanos.31 Na medida que se aumenta a concentração de proteínas adicionadas ao meio reacional (Tabela 3), em tese também se está aumentando o número de moléculas de GR disponíveis na reação. Assim, a redução da atividade da GR em maiores concentrações de extrato proteico pode indicar uma proporção desfavorável entre moléculas de enzimas e moléculas de substrato (GSSG ou NADPH). Entretanto, como não houve interação entre os efeitos de GSSG e extrato protéico (Figura 3), estudos adicionais são necessários para confirmar esta hipótese. Validação do método de determinação da GR em A. singularis Para validar o método de quantificação da GR em A. singularis, a enzima foi quantificada em exemplares coletados em 7 riachos de 3 BH com características distintas em relação ao percentual de naturalidade. Em cada riacho, os organismos foram amostrados em um trecho a montante (em direção à nascente) e outro a jusante (em direção à foz, pontualmente menos preservado). A análise integrando as duas variáveis independentes (BH e trechos de coleta) mostrou que a atividade da GR é influenciada primariamente pela BH (Tabela 4), com interação entre trecho de coleta e BH. Em outras palavras, isso significa que o estado de preservação pontual do trecho de coleta terá influência sobre a GR dependendo da BH avaliada.
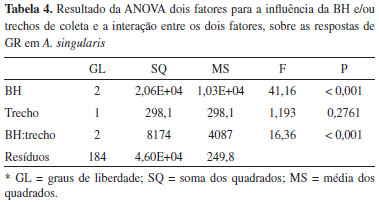
Em concordância com os dados acima, a atividade da GR foi significativamente maior em organismos coletados na BH do Rio Dourado do que nas BH dos Rios Suzana e Ligeirinho-Leãozinho, sendo que nestas duas últimas os trechos a jusante dentro de cada riacho, apresentaram níveis ainda menores de GR do que os trechos a montante (Tabela 5).
A BH do Rio Dourado, aonde a atividade da GR foi mais alta, apresenta alto percentual de vegetação arbórea nativa (58,73%), o que corresponde a uma área natural cerca de 65% e 120% maior do que as BH do Rio Suzana e Ligeirinho-Leãozinho, respectivamente.22-25 Esse fato contribui para a manutenção de um melhor estado de preservação dos riachos sob domínio da BH do Rio Dourado. Por exemplo, a presença de vegetação pode minimizar processos erosivos e de lixiviação,35,36 os quais promovem maior contaminação dos corpos hídricos por metais e resíduos agrícolas.37,38 A maior contaminação por metais provenientes de atividades antrópicas poderia explicar a menor atividade da GR nas BH dos Rios Suzana e Ligeirinho-Leãozinho, considerando que já foi demonstrado que a GR pode ser inibida por diferentes tipos de metais.39 Cossu et al.40 realizaram um estudo in situ com bivalves da espécie Unio tumidus e observaram redução de atividade da GR nos organismos coletados em trechos de riachos contaminados por misturas de diversos xenobióticos, incluindo alta concentração de metais como cromo, cobre, chumbo e mercúrio. Investigações adicionais são necessárias para confirmar se o padrão de redução da GR em Aegla está associado efetivamente ao nível de metais no ambiente de BH ou a outros fatores que afetam a qualidade da água nestes locais. O interesse pela utilização de biomarcadores fisiológicos e bioquímicos como ferramenta de monitoramento vem crescendo na última década, o que tem contribuído para o aprimoramento da determinação de risco ambiental em riachos (revisado em Rodrigues et al.).41 De forma geral, esses estudos têm uma abordagem que associa a análise de parâmetros baseados em populações e comunidades, com a análise toxicológica de biomarcadores em espécies bioindicadoras41 não apresentando, entretanto, avaliações direcionadas para o aprimoramento metodológico. Especificamente, no caso do gênero Aegla, não existem dados prévios na literatura que tenham enfoque específico na metodologia de quantificação da GR, ou mesmo informações sobre a determinação da atividade da enzima em avaliações toxicológicas in situ ou em condições laboratoriais controladas. A informação disponível na literatura é de que a proporção de GSH/GSSG é relativamente constante entre diferentes espécies de Aegla,20 indicando que o grupo filogenético tem mecanismos eficientes para reciclagem e/ou síntese de GSH.
CONCLUSÃO Foi possível maximizar a determinação da atividade da GR no organismo bioindicador A. singularis. A atividade da GR não foi afetada significativamente pelas diferentes temperaturas de reação testadas, mantendo-se com aproximadamente 100% de atividade na faixa de 15 a 30 °C. A avaliação cinética da atividade enzimática, com 6 e 10 minutos de reação, também não apresentou diferença significativa. A concentração de substrato influencia positivamente e a concentração de extrato influencia negativamente na atividade da GR. A BH foi a principal variável que influenciou a atividade da GR em A. singularis, com maior atividade da enzima quantificada em organismos coletados na BH que apresenta melhor estado de preservação da vegetação nativa. Os trechos de coleta (a montante ou a jusante) isoladamente não tiveram influência sobre a atividade da GR. Porém, apresentaram interação com a BH de origem. Isso significa que a influência do trecho de coleta sobre a GR depende da BH avaliada. No presente estudo, o uso do planejamento de experimentos para padronizar a quantificação da GR em Aegla, seguido da aplicação do método em exemplares do organismo coletados in situ, se mostrou eficiente no sentido de viabilizar e otimizar o uso da GR como biomarcador de qualidade ambiental. Esse pode contribuir para agilizar o desenvolvimento de protocolos para outros biomarcadores ecotoxicológicos, assim como para o estudo de diferentes variáveis que possam ter interferência com a própria atividade da GR.
AGRADECIMENTOS Os autores agradecem à CAPES, ao CNPq (Processo n. 473648/2013-0) e à URI pelo suporte financeiro.
REFERÊNCIAS 1. Moore, M. N.; Depledge, M. H.; Readman, J. W.; Paul Leonard, D. R.; Mutat. Res. 2004, 552, 247. 2. Halliwell, B.; Gutteridge, J. M. C.; Free radicals in biology and medicine, 4th ed.; Oxford University Press Inc.: Oxford, 2007. 3. Huber, P. C.; Almeida, W. P.; Fátima, A.; Quim. Nova 2008, 31, 1170. 4. Lv, H.; Zhen, C.; Liu, J.; Yang, P.; Hu, L.; Shang, P.; Oxid. Med. Cell Longev. (2019), doi:10.1155/2019/3150145. 5. Chen, H.; Zha, J.; Yuan, L.; Wang, Z.; Chemosphere 2015, 119, 856. 6. Franco, J. L.; Posser, T.; Mattos, J. J.; Sánchez-Chardi, A.; Trevisan, R.; Oliveira, C. S.; Carvalho, P. S. M.; Leal, R. B.; Marques, M. R. F.; Bainy, A. C. D.; Dafre, A. L.; Mar. Environ. Res. 2008, 66, 88. 7. Liu, N.; Wang, L.; Yan, B.; Li, Y.; Ye, F.; Li, J.; Wang, Q.; Aquat. Sci. 2014, 741, 3. 8. Mesquita, S. R.; Ergen, S. F.; Rodrigues, A. P.; Oliva-Teles, M. T.; Delerue-Matos, C.; Guimarães, L.; Aquat. Toxicol. 2015, 159, 225. 9. Negro, C. L.; Iturburu, F. G.; Mendieta, J.; Menone, M. L.; Collins, P.; Bull. Environ. Contam. Toxicol. 2019, 103, 405. 10. Mannervik, B.; Curr. Protoc. Toxicol. 1999, 7.2.1. 11. Ramos-Vasconcelos, G. R.; Hermes-Lima, M.; J. Exp. Biol. 2003, 206, 675. 12. Smith, I. K.; Vierheller, T. L.; Thorne, A. C. A.; Anal. Biochem. 1988, 175, 408. 13. Liu, N. A.; Wang, L.; Yan, B. O.; Li, Y.; Ye, F.; Li, J.; Wang, Q.; Hydrobiologia 2014, 741, 3. 14. Lou, X.; Hong, Y.; Chen, S.; Leung, C. W.; Zhao, N.; Situ, B.; Lam, J. W.; Tang, B. Z.; Sci. Rep. 2014, 4, 1. 15. Box, G. E. P.; Hunter, J. S.; Hunter, W. G.; Statistics for Experimenters: Design, Innovation, and Discovery, 2nd ed.; Wiley-Interscience: New York, 2005. 16. Assou, M.; Madinzi, A.; Aboulhassan, M. A.; Souabi, S.; International Journal of Civil and Environmental Engineering 2014, 36, 1290. 17. Camcioglu, S.; Pekel, L. C.; Polat, K.; Hapoglu, H.; Management of Environmental Quality 2014, 25, 86. 18. Reinehr, R. C. R.; Giordanni, P. R.; Alves, A. A. A.; Klen, M. R. F.; Toner, A. R. M.; Desalin. Water Treat. 2019, 238, 27. 19. Bond-Buckup, G.; Santos, S.; Ciênc. Ambient. 2007, 35, 47. 20. Faria, S. C.; Klein, R. D.; Corta, P. G.; Crivellaro, M. S.; Santos, S.; Bueno, S. L. S.; Bianchini, A.; Sci Rep. (2018), doi:10.1038/s41598-018-21188-1. 21. Borges, A. C. P.; Piassão, J. F. G.; Paula, M. O.; Sepp, S.; Bez, C. F. S.; Hepp, L. U.; Valduga, A. T.; Pereira, A. A. M.; Cansian, R. L.; Braz. J. Biol. 2018, 78, 61. 22. https://uploads.preferechim2.astrusweb.dataware.com.br/uploads.preferechim2.astrusweb.dataware.com.br/uploads/files/Plano_ 23. Decian, V. S.; Tese de Doutorado, Universidade Federal de São Carlos, Brasil, 2012. 24. https://uploads.preferechim2.astrusweb.dataware.com.br/uploads.preferechim2.astrusweb.dataware.com.br/uploads/files/Plano_ 25. da Silva, J. M.; Dissertação de Mestrado, Universidade Integrada do Alto Uruguai e das Missões, Brasil, 2017. 26. Callisto, M.; Ferreira, W. R.; Moreno, P.; Goulart, M.; Petrucio, M.; Acta Limnol. Bras. 2002, 14, 91. 27. Melo, G. A. S.; Manual de identificação dos Crustacea Decapoda de água doce do Brasil; Edições Loyola: São Paulo, 2003. 28. Bradford, M. M.; Anal. Biochem. 1976, 72, 248. 29. Somero, G. N.; Comp. Biochem. Physiol., Part B: Biochem. Mol. Biol. 2004, 139, 321. 30. Nelson, D. L.; Cox, M. M.; Princípios de Bioquímica de Lehninger, 6th ed.; Artmed: Porto Alegre, 2014. 31. Wong, K. K.; Vanoni, M. A.; Blanchard, J. S.; Biochemistry 1988, 27, 7091. 32. Deponte, M.; Biochim. Biophys. Acta 2013, 1830, 3217. 33. Berry, A.; Scrutton, N. S.; Perham, R. N.; Biochemistry 1989, 28, 1264. 34. Couto, N.; Wood, J.; Barber, J.; Free Radical Biol. Med. 2016, 95, 27. 35. Niyogi, D. K.; Koren, M.; Arbuckle, C. J.; Towsend, C. R.; J. Environ. Manage. 2007, 39, 213. 36. Aragão, R.; Almeida, J. A. P.; Figueiredo, E. E.; Srinivasan, V. S.; Revista Brasileira de Engenharia Agrícola e Ambiental 2011, 15, 731. 37. Bing, H.; Wu, Y.; Nahm, W. H.; Liu, E.; Environ. Earth Sci. 2013, 69, 2679. 38. Akcil, A.; Erust, C.; Ozdemiroglu, S.; Fonti, V.; Beolchini, F.; J. Cleaner Prod. 2015, 86, 24. 39. Ekinci, D.; Sentürk, M.; J. Enzyme Inhib. Med. Chem. 2013, 28, 11. 40. Cossu, C.; Doyotte, A.; Jacquin, M. C.; Babut, M.; Exinger, A.; Vasseur, P.; Ecotoxicol. Environ. Saf. 1997, 38, 122. 41. Rodrigues, C.; Guimarães, L.; Vieira, N.; Hydrobiologia (2019), doi:10.1007/s10750-019-03991-7. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access