Nota Técnica
|
|
| Otimização e validação de método enzimático semiquantitativo simples e de baixo custo para a busca de inibidores de cisteino proteases Optimization and validation of a simple and low-cost semi-quantitative enzymatic method to search for cisteine proteases inhibitors |
|
Ana Karolina de Souza Andrade; Luana Andrade Santos; Edisleide Silva Menezes; Rafael Ciro Marques Cavalcante; James Almada da Silva*
Departamento de Farmácia, Universidade Federal de Sergipe, 49400-000 Lagarto - SE, Brasil Recebido em 29/08/2020 *e-mail: jamesalmada@hotmail.com Cysteine proteases are an important class of enzymes involved in several essential biological processes. For this reason, these enzymes are important therapeutic targets, and therefore can be used in enzymatic assays for the search of hits. The most widely used methods in the search of enzyme inhibitors are spectrophotometric, however they have relatively high operational costs. For this reason, simpler, and cheaper methods can be useful in the search for enzyme inhibitors. In this work, a simple and low-cost semi-quantitative method was optimized and validated using papain and skimmed powdered milk (casein) as a substrate. The ideal condition for the method was the one that used powdered milk, 0.250%, papain solution, 9.4 mg mL-1 and incubation time, 18 h. The method validation was performed using the inhibitor E64 and herb extracts, in different concentrations. After carrying out the experiments and statistical analyses, a high reproducibility can be observed, with relative standard deviations below 2.1%. Plant extracts were used to test the method and the results indicated that these extracts are promising for the search of cysteine protease inhibitors. The method developed here has simplicity and reproducibility and has great application, as preliminary study, in the search for inhibitors of cysteine proteases. INTRODUÇÃO Biomacromoléculas envolvidas em importantes processos patológicos geralmente são utilizadas como alvos terapêuticos de sucesso no desenvolvimento de fármacos. A sua importância se dá, pois a interação de moléculas com esses importantes alvos terapêuticos pode modular a sua função e consequentemente alterar o curso da doença.1 Segundo o TTD (Therapeutic Target Database), banco de dados que valida alvos terapêuticos por meio de evidências clínicas ou patentes/artigos científicos, existem 2.954 alvos terapêuticos humanos e 465 alvos terapêuticos de agentes infecciosos validados.2 Hopkins e Groom3 classificaram esses alvos terapêuticos como: enzimas, receptores acoplados a proteína G, canais iônicos, transportadores, receptores nucleares, integrinas e DNA. As enzimas merecem destaque no desenvolvimento de fármacos, pois já foram responsáveis pela descoberta de cerca de 44% dos fármacos existentes no mercado. Além disso, 50% dos alvos terapêuticos utilizados na busca de candidatos à fármacos são representados pelas enzimas.1 As cisteíno proteases do tipo papaína (papain-like cysteine proteases) são importantes proteases expressas por animais, vegetais e microrganismos, e que estão envolvidas em diversas doenças.4 Essas enzimas possuem um sítio catalítico altamente conservado (tríade catalítica Cys, His e Asp), responsável pele hidrólise irreversível de peptídeos. Pode-se citar como exemplo: as catepsinas e calpainas, que regulam a atividade de peptídeos através da clivagem em sítios específicos.5 As catepsinas estão envolvidas em processos patológicos como, doenças musculoesqueléticas,6,7 câncer, doenças autoimunes, problemas cardíacos,8,9 doença de Alzheimer,10 entre outras. Já as calpainas regulam a atividade de diversas proteínas envolvidas com processos patológicos neuronais, como plasticidade neuronal, cognição e doenças neurodegenerativas como a doença de Alzheimer e doença de Parkinson.5 Além das cisteíno proteases, expressas por seres humanos, diversas cisteíno proteases vitais para parasitas, bactérias e vírus também são alvos terapêuticos importantes para a busca de substâncias eficazes no tratamento de doenças infecciosas.11,12 Algumas cisteíno proteases expressas pelo novo coronavírus (SARS-CoV-2) e por outros coronavírus (MERS-CoV e o SARS-CoV) responsáveis pelo surgimento de epidemias/pandemias, são essenciais para a sua sobrevivência e aumento do poder de infecção. Os coronavírus mencionados acima possuem uma proteína estrutural presente na membrana bilipídica, denominada proteína S. Essa glicoproteína é responsável pela entrada do vírus nas células do organismo infectado.13 No entanto, o vírus só consegue entrar nas células do hospedeiro e liberar o RNA após ativação mediada por uma cisteíno protesase tipo papaína, a catepsina L.14 Após a entrada do vírus nas células e liberação do RNA viral, inicia-se o processo de replicação, que é mediado por outras proteases, principalmente a cisteíno protease tipo papaína, denominada PLP ou PLpro 15,16 e a cisteíno protease tipo quimiotripsina, denominada 3CLpro.17 Dada a importância dessas enzimas para o ciclo vital do coronavírus, é de extrema importância a descoberta de inibidores enzimáticos que impeçam a entrada e/ou replicação viral, seja utilizando abordagens in sílico,17-19 ou experimentos in vitro.15,20,21 Esse é apenas um exemplo da importância em se descobrir inibidores de enzimas patologicamente importantes. As metodologias in vitro para a descoberta de inibidores de proteases utilizam substratos cromogênicos22,23 e mais frequentemente substratos fluorogênicos15,20,24-26 específicos relativamente caros. Além disso, requerem a utilização de equipamentos como espectrofotômetro ou espectrofluorímetro para a detecção do produto da reação enzimática. Técnicas espectrofotométricas podem gerar resultados não confiáveis se o extrato ou composto a ser testado absorver radiação no comprimento de onda do experimento.27 Desse modo, técnicas utilizadas para a descoberta de inibidores enzimáticos que requerem aparelhagens mais simples, substratos mais baratos e detecção visual do produto reacional são de grande relevância para o processo de descoberta de fármacos. Alguns autores28,29 utilizaram uma técnica da difusão em ágar para a detecção de proteases, utilizando enzimas da classe das serino proteases e metaloproteases, e caseína como substrato. Grenier30 estudou uma técnica similar aos autores mencionados anteriormente utilizando ágar, leite em pó e papaína para a busca de inibidores de cisteíno proteases. Esse autor utilizou faixas de papel para impregnar as amostras teste e verificar se estas amostras apresentam atividade de inibição enzimática. Ele demonstrou que a técnica pode ser utilizada para a detecção de inibidores de cisteíno proteases, entretanto, esse método não passou por processo de validação. Neste trabalho, uma técnica semelhante foi otimizada e validada utilizando a papaína como modelo de alvo terapêutico, para ser utilizada como estudo preliminar na busca de inibidores de cisteíno proteases tipo papaína. Diferente de Grenier,30 a mistura inibidor/enzima foi previamente incubada e em seguida adicionada aos poços construídos na placa contendo ágar e leite em pó. Além disso, um inibidor irreversível de cisteíno proteases (E64, trans-epoxisuccinil-L-leucilamido(4-guanidino)butano) foi utilizado para a realização da validação do método. A papaína, cisteíno protease extraída do látex do mamão (Carica papaya), foi a primeira enzima da família a ter sua estrutura tridimensional determinada por cristolagrafia de raio-X. Ela consiste em uma cadeia simples de polipeptídeo com 212 resíduos de aminoácidos.31 Para se testar o método desenvolvido, algumas espécies já avaliadas por nosso grupo de pesquisa quanto a sua atividade de inibição de uma cisteíno protease pela técnica espetrofluorimétrica foram utilizadas. As plantas utilizadas foram: gengibre (Zingiber officinale Roscoe), pequi (Caryocar brasiliense Cambess) e camélia (Camelia sinensis L. Kuntze).
PARTE EXPERIMENTAL Material e reagentes Papaina pura (Dinâmica), ágar nutriente (IONLAB) e leite em pó desnatado (Molico®) foram utilizados nos ensaios de determinação da atividade enzimática. Todos os solventes e reagentes utilizados foram de grau analítico. As plantas utilizadas para a preparação dos extratos foram gengibre (Zingiber officinale Roscoe) e pequi (Caryocar brasiliense Cambess), adquiridos em comércio local. Folhas de camélia (Camelia sinensis L. Kuntze) foram adquiridas através do site de vendas de chás e ervas medicinais, www.ervadobem.com. O mesocarpo seco de babaçu (Orbignya phalerata Mart.) foi gentilmente doado pela empresa Esmeraldo Produtos Naturais, Fortaleza, Ceará. Ensaio de inibição cisteino proteases A metodologia foi desenvolvida utilizando como base o trabalho de Morris et al.32 Esses autores detectaram a atividade de proteases de micro-organismos, em placa de petri, utilizando ágar e leite em pó desnatado. Trata-se de em um ensaio semiquantitativo por método de difusão em ágar conduzido em placas de petri. O substrato da enzima é a caseína que está presente no leite. Inicialmente, realizou-se o preparo e esterilização do ágar nutriente com leite em concentrações definidas a seguir. Em seguida, foram adicionados 50 mL do ágar/leite em placas de petri (d = 14 cm). Após a solidificação do ágar/leite, foram construídos poços com diâmetro de 5,00 mm, de maneira uniforme em toda a placa, utilizando um cilindro oco de aço para a perfuração do meio. A esses poços foram adicionadas as soluções formadas entre o inibidor irreversível E64 (trans-epoxisuccinil-L-leucilamido(4-guanidino)butano) e enzima ou extrato vegetal (100; 200 e 400 µg mL-1, dissolvidos em DMSO) e enzima. Nos poços controle (correspondente a 100% de atividade enzimática) foram adicionados solução aquosa contendo DMSO e enzima. Essas soluções foram preparadas na proporção de 1:40, v/v (inibidor/enzima ou extrato/enzima) e permaneceram incubadas por cinco minutos antes da sua adição nos poços. A concentração final de DMSO em todos os poços foi de 2,5%. A solução enzimática (concentração definida a seguir) foi preparada em tampão fosfato 10 mmol L-1 (pH 6,7), contendo 5 mmol L-1 de EDTA. Após a adição das soluções aos poços, a placa de petri foi mantida a 37 °C em estufa bacteriológica durante um período de tempo definido a seguir. A atividade enzimática foi observada pela formação de halos ao redor dos poços no ágar/leite. Para facilitar a visualização dos halos, foi adicionada 20 mL de uma solução de ácido clorídrico 0,1 mol L-1. A caseína intacta presente na placa, precipita após adição do ácido clorídrico, enquanto que aquela que não sofreu hidrólise enzimática não precipita, formando-se assim o halo. A aferição dos halos foi realizada com o auxílio de um paquímetro digital. Os ensaios foram realizados em triplicata para cada amostra. Para o cálculo da inibição percentual ocasionada pelos extratos testados foi utilizada a Equação 1. 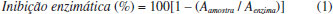 em que Aamostra é a área do halo formado em volta dos poços contendo o extrato/enzima e, Aenzima é a área do halo formado em volta dos poços contendo apenas enzima. Otimização do ensaio de inibição enzimática Para a determinação das melhores condições do ensaio utilizou-se quatro concetrações de solução enzimática, três concentrações de leite em pó e três períodos de tempo de incubação, como pode se observado na Tabela 1.
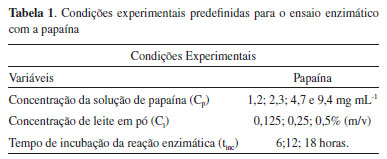
Para a determinação da melhor concentração de leite, fixou-se o tempo de incubação do experimento em 6 horas e a concentração da solução enzimática em 9,4 mg mL-1. Utilizaram-se as concentrações de leite: 0,125; 0,25; 0,5% (m/v). A concentração de leite na qual se observou o halo formado ao redor dos poços com maior nitidez foi aquela utilizada para o prosseguimento da otimização da metodologia de inibição enzimática. Em seguida, para a determinação da melhor concentração enzimática (1,2; 2,3; 4,7 e 9,4 mg mL-1) e o melhor tempo de incubação (6; 12; 18 horas), fizeram-se todas as combinações possíveis utilizando as quatro concentrações de solução de papaína e os três tempos de incubação. As melhores condições obtidas com a otimização da metodologia foram utilizadas para a validação do ensaio de inibição de cisteíno protesases, utilizando-se o inibidor irreversível E64 e extratos vegetais, em diferentes concentrações. Os ensaios foram realizados em triplicata. Após a realização dos experimentos, foram calculadas as médias das medidas dos halos obtidas, os erros padrão e os erros padrão relativo dos experimentos. Calcularam-se tambem os percentuais de inibição do inibidor E64 (10 µmol L-1) e dos extratos testados. Validação do ensaio de inibição enzimática Na validação utilizou-se as melhores condições obtidas no desenvolvimento da metodologia (Cp, Cl, tinc). As concentrações de E64 utilizadas para a validação foram: 2, 4, 6, 8, 10 µmol L-1. As soluções foram preparadas em DMSO, portanto, como controle negativo utilizou-se o mesmo solvente na concentração final de 2,5%. A validação foi conduzida com a realização de três experimentos independentes em triplicata, inter-dias. Após a realização dos experimentos, foram calculadas as médias das medidas dos halos obtidas, os desvios padrão e os desvios padrão relativo dos experimentos. Preparação dos extratos vegetais O gengibre (Z. officinale) e o mesocarpo do pequi (C. brasiliense), adquiridos em comércio local, foram secos em estufa com circulação de ar a 40 ºC por um período de 5 dias. Em seguida, o material vegetal foi triturado com o auxílio de um liquidificador doméstico. As folhas de camélia (C. sinensis) e o mesocarpo de babaçu (O. phalerata) já foram adquiridos na forma seca e pulverizada. Cada material vegetal foi submetido ao processo de extração exaustiva, por maceração (1:3; m/v), utilizando álcool etílico 99,5%. Em seguida, realizou-se a filtração em papel filtro Whatman e evaporação do solvente sob vácuo em rota-evaporador a 40 °C, obtendo-se, dessa forma, o extrato bruto.33
RESULTADOS E DISCUSSÃO Otimização do ensaio de inibição enzimática Determinação da melhor concentração de leite Três placas de petri foram preparadas utilizando ágar nutriente e leite em pó nas concentrações de 0,125%, 0,250% e 0,500%. A concentração de papaína e o tempo de incubação foram fixados em 9,4 mg mL-1 e 6 horas, respectivamente. Ao final das 6 horas de incubação a 37 °C em estufa bacteriológica adicionou-se HCl 0,1 mol L-1 e esperou-se 5 minutos para a precipitação da caseína intacta. A atividade enzimática foi observada pela formação de halos ao redor dos poços no ágar/leite, resultado da degradação da caseína presente no leite, pela enzima. A caseína intacta presente na placa precipita após adição do ácido clorídrico, enquanto aquela que não sofreu hidrólise enzimática não precipita, formando-se assim o halo. A menor concentração de leite em pó (0,125%) resultou na formação de halos pouco visíveis, enquanto a partir de 0,250% houve a formação de halos nítidos. Essa concentração foi selecionada para os estudos posteriores, uma vez que os halos formados estavam nítidos o suficiente para a sua visualização e medida dos seus diâmetros. Determinação da melhor concentração de solução enzimática e do melhor tempo de incubação para a reação enzimática Para determinação da melhor concentração de papaína e o melhor tempo de incubação foram realizadas as doze combinações possíveis, utilizando as quatro diferentes concentrações de enzima (1,2; 2,3; 4,7 e 9,4 mg mL-1) e os três diferentes tempos de incubação (6; 12; 18 h). Pode-se observar na Tabela 2 todas as combinações e os diâmetros dos halos formados em cada uma das condições.
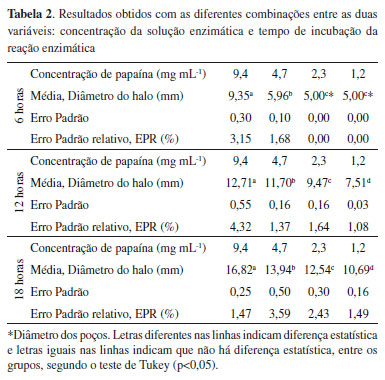
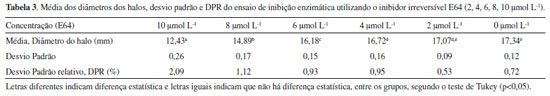
Pode-se observar claramente que, independentemente do tempo de incubação, quanto maior a concentração da solução de papaína, maior é o diâmetro do halo. Análise similar pode ser realizada, comparando-se os diâmetros dos halos nos diferentes tempos de incubação, ou seja, independente da concentração de papaína, quanto maior o tempo de incubação, maior é o diâmetro do halo. Com base nessas observações, a condição em que houve a formação do maior halo foi visualizada utilizando-se solução enzimática na concentração de 9,4 mg mL-1 e 18 horas de incubação. Portanto, as condições experimentais com a melhor visualização dos halos foi: concentração de leite em pó, 0,250%; concentração de solução de papaína, 9,4 mg mL-1; e tempo de incubação, 18 horas. Essas condições foram utilizadas para a validação da metodologia, utilizando o inibidor irreversível E64. Vale mencionar a alta reprodutibilidade dos experimentos, considerando que o EPR em todos os experimentos foi menor do que 5%. Validação do ensaio de inibição enzimática Para se avaliar a reprodutibilidade do método, os experimentos foram realizados em três dias diferentes (inter-dias). Em cada dia o experimento foi realizado em triplicata. Para a validação do método foi utilizado o inibidor irreversível de cisteíno proteases E64, nas concentrações: 2, 4, 6, 8, 10 µmol L-1 (Tabela 3).
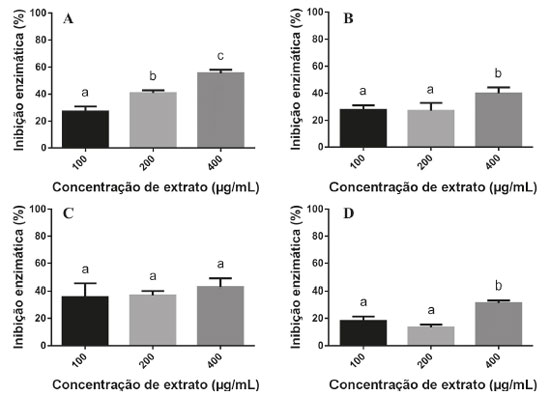
Pode-se observar que, quanto maior a concentração do E64, menor é o diâmetro dos halos, indicando um efeito de inibição enzimática dependente da concentração, situação bastante coerente em experimentos biológicos e bioquímicos. O maior valor de DPR encontrado nos experimentos de validação da metodologia foi de 2,09%, na concentração de 10 µmol L-1. Como os DPRs foram menores do que 5%, pode-se afirmar o que o método apresentou excelente reprodutibilidade. Com base na avaliação desses resultados pode-se, então, afirmar que esse método de inibição da papaína é reprodutível e confiável, podendo ser utilizado para a busca de inibidores de cisteíno proteases. Nas condições desse experimento, o inibidor E64 inibiu a atividade da papaína em 52,6% na maior concentração testada (10 µmol L-1), uma inibição muito inferior se compararmos com o seu valor de IC50 (9 nmol L-1) obtido por espetrofluorimetria.33 Essa diferença pode ser explicada pelas limitações difusionais do E64 no ágar e por se tratar de um método semiquantitativo, muito diferente operacionalmente do método quantitativo em que se utiliza espectrofluorímetro, em que a reação enzimática se processa completamente em meio líquido e com substratos fluorogênicos. Atividade de inibição enzimática de extratos vegetais frente à papaína Para os ensaios de inibição enzimática utilizou-se extratos brutos de quatro espécies vegetais (Orbygnia phalerata, Zingiber officinale, Camellia sinensis, Caryocar brasiliense), em três concentrações diferentes (100, 200 e 400 µg mL-1). A Equação 1, descrita anteriormente, foi utilizada para o cálculo da inibição enzimática (%) dos extratos. É possível observar na Figura 1 que a atividade de inibição enzimática da espécie O. phalerata aumentou à medida que a concentração dos extratos aumentou, mostrando um efeito concentração-dependente. Além disso, esse extrato foi o que apresentou a maior atividade de inibição enzimática (55,4%, na concentração de 400 µg mL-1). Os outros extratos testados também apresentaram efeito de inibição da papaína, porém, foram menos ativos do que o extrato de O. phalerata. Para os extratos das espécies Z. officinale e C. brasiliense não houve diferença estatística entre as concentrações de 100 e 200 µg mL-1, enquanto que para o extrato de C. sinensis não houve diferença significativa para as diferentes concentrações ensaiadas.
Silva,34 em seu trabalho de doutorado, realizou ensaios de inibição enzimática utilizando diversos extratos vegetais frente à catepsina K, uma cisteíno protease pertencente à superfamília da papaína. Esses ensaios foram realizados utilizando uma metodologia tradicional espectrofluorimétrica, portanto, a comparação dos resultados pode ter um grande valor na validação do método semiquantitativo realizado nesse trabalho. Dentre os extratos testados, os extratos de O. phalerata e Z. officinale apresentaram efeito inibitório frente a esta cisteino protease (>70%) na concentração de 500 µg mL-1, corroborando com o efeito inibitório encontrado nesse trabalho, apesar do percentual de inibição ter sido numericamente diferente. Dessa maneira, a papaína pode vir a ser utilizada como um modelo para a busca de inibidores de cisteino proteases em ensaios semiquantitativos. Observou-se que a seleção de extratos com potencial em inibir cisteíno proteases pode ser realizada com sucesso, da mesma forma que nas técnicas tradicionais (mais caras) e, por isso, esse método semiquantitativo apresenta grande vantagem em ensaios preliminares, reduzindo os custos nessa etapa inicial, apesar de não poder ser utilizada para a determinação da potência e do mecanismo de inibição enzimática. Até onde se sabe, não há outros trabalhos publicados demonstrando o efeito de inibição de cisteíno proteases com as espécies vegetais utilizadas neste trabalho.
CONCLUSÕES Um ensaio semiquantitativo de inibição da papaína, operacionalmente simples e de baixo custo, foi otimizado e validado com sucesso, e pode ser utilizado para a busca de inibidores de cisteíno proteases. Apresentou alta reprodutibilidade e, de acordo com os resultados obtidos, pode ser utilizado preliminarmente para a detecção de extratos com potencial na inibição de cisteíno proteases, merecendo destaque o extrato do mesocarpo de O. phalerata, que apresentou inibição da papaína acima de 50%.
MATERIAL SUPLEMENTAR Algumas imagens dos ensaios utilizados para a validação do método enzimático utilizando-se o inibidor reversível E64 estão disponíveis em http://quimicanova.sbq.org.br, na forma de arquivo PDF, com acesso livre.
AGRADECIMENTOS Agradecemos à Universidade Aberta do Brasil (UAB), do Povoado Colônia Treze, Lagarto, Sergipe, pela disponibilização dos laboratórios para a realização de todos os experimentos deste trabalho. Agradecemos também a Fundação de Apoio à Pesquisa e à Inovação Tecnológica do Estado de Sergipe (FAPITEC), pelo apoio financeiro na forma de bolsa PIBIC.
REFERÊNCIAS 1. Zheng, C. J.; Han, L. Y.; Yap, C. W.; Ji, Z. L.; Cao, Z. W.; Chen, Y. Z.; Pharmacol Rev. 2006, 58, 259. 2. Wang, Y.; Zhang, S.; Li, F.; Zhou, Y.; Zhang, Y.; Wang, Z.; Zhang, R.; Zhu, J.; Ren, Y.; Tan, Y.; Qin, C.; Li, Y.; Li, X.; Chen, Y.; Zhu, F.; Nucleic Acids Res. (2020), DOI: 10.1093/narlgkz981. 3. Hopkins, A. L.; Groom, C. R.; Nat. Rev. Drug Discov. 2002, 1, 727. 4. Brömme, D.; Nallaseth, F. S.; Turk. B.; Methods 2004, 32, 199. 5. Siklos, M.; BenAissa, M.; Thatcher, G. R. J.; Acta Pharm. Sin. B 2015, 5, 506. 6. Yasuda, Y.; Kaleta, J.; Brömme, D.; Adv. Drug Deliv. Rev. 2005, 57, 973. 7. Lu, J.; Wang, M.; Wang, Z.; Fu, Z.; Lu, A.; Zhang, G.; J. Enzyme Inhib. Med. Chem. 2018, 33, 890. 8. Aggarwal, N.; Sloane, B. F.; Proteomics: Clin. Appl. 2014, 8, 427. 9. Sena, B. F.; Figueiredo, J. L.; Aikawa, E.; Front. Cardiovasc. Med. 2018, 4, 1. 10. Hook, V.; Yoon, M.; Mosier, C.; Ito, G.; Podvin, S.; Head, B.P.; Rissman, R.; O'Donoghue, A. J.; Hook, G.; Biochim. Biophys. Acta, Proteins Proteomics 2020, 1868, 140428. 11. Kędzior, M.; Seredyński, R.; Gutowicz, J.; Med. Microbiol. Immunol. 2016, 205, 275. 12. Verma, S.; Dixit, R.; Pandey, K. C.; Front. Pharmacol. 2016, 7, 1. 13. Satarker, S.; Nampoothiri, M.; Arch. Med. Res. (2020), DOI: 10.1016/j.arcmed.2020.05.012. 14. Liu, T.; Luo, S.; Libby, P.; Shi, G-P.; Pharmacol. Ther. 2020, 213, 107587. 15. Park, J. Y.; Ko, J. A.; Kim, D. W.; Kim, M. Y.; Kwon, H.; Jeong, H. J.; Kim, C. Y.; Park, K. H.; Lee, W. S.; Ryu, Y. B.; J. Enzyme Inhib. Med. Chem. 2016, 31, 23. 16. Clemente, V.; D'arcy, P.; Bazzaro, M.; Int. J. Mol. Sci. 2020, 21, 1. 17. Tahir ul Qamar, M.; Alqahtani, S. M.; Alamri, M. A.; Chen L-L.; J. Pharm. Anal. (2020), DOI: 10.1016/j.jpha.2020.03.009. 18. Gyebi, G. A.; Ogunro, O. B.; Adegunloye, A. P.; Ogunyemi, O. M.; Afolabi, S. O.; J. Biomol. Struct. Dyn. (2020), DOI: 10.1080/07391102.2020.1764868. 19. Gurung, A. B.; Ali, M. A.; Lee J.; Farah, M. A.; Al-Anazi, K. M.; Life Sci. 2020, 255, 117831. 20. Park, J. Y.; Yuk, H. J.; Ryu, H. W.; Lim, S. H.; Kim, K. S.; Park, K. H.; Ryu, Y. B.; Lee, S.; J. Enzyme Inhib. Med. Chem. 2017, 32, 504. 21. Rut, W.; Lv, Z.; Zmudzinski, M.; Patchett, S.; Nayak, D.; Snipas, S. J.; Oualid, F. E.; Huang, T. T.; Bekes, M.; Drag, M.; Olsen, S. K.; bioRxiv 2020, doi:10.1101/2020.04.29.068890. 22. Papisova, A. I.; Javadov, A.; Rudenskaya, Y. A.; Balandina, G. N.; Zhantiev, D. R.; Korsunovskaia, O. S.; Dunaevsky, Y. E.; Rudenskaya, G. N.; Biochimie 2011, 93, 141. 23. Wang, X; Yang, Z.; Su, F.; Jin, L.; Boadi, E. O.; Cang, Y.; Wang, H.; Molecules 2020, 25, 422. 24. Assis, D. M.; Gontijo, V. S.; Pereira, I. O.; Santos, J. A. N.; Camps, I.; Nagem, T. J.; Ellena, J.; Izidoro, M. A.; Tersariol, I. L. S.; Barros, N. M. T.; Dorigueto, C. A.; Santos, H. M.; Juliano, M. A.; J. Enzyme Inhib. Med. Chem. 2013, 28, 661. 25. Marques, A. F.; Gomes, P. S. F. C.; Oliveira. P. L.; Rosenthal, P. J.; Pascutti, P. G.; Lima, L. M. T. R.; Arch. Biochem. Biophys. 2015, 573, 92. 26. Silva, T. L.; Toffano, L.; Fernandes, J. B.; Silva, M. F. G.; Sousa, L. R. F.; Vieira. P. C.; Braz. J. Microbiol. (2020), DOI:10.1007/s42770-020-00256-7. 27. Simeonov, A.; Davis, M. I. In Assay Guidance Manual; Markossian, S., Sittampalam, G. S., Grossman, A., Brimacombe, K., ed.; Eli Lilly & Company and the National Center for Advancing Translational Sciences: Bethesda, 2018. 28. Cowan, D. A.; Daniel, R. M.; J. Biochem. Biophys. Methods 1982, 6, 31. 29. Kourteva, I.; Sleigh, R. W.; Hjertén, S.; Anal. Biochem. 1987, 162, 345. 30. Grenier, D.; J. Biochem. Biophys. Methods 1991, 22, 35. 31. Drenth, J.; Jansonius, J. N.; Koekoek, R.; Wolthers, B. G.; Adv. Protein Chem. 1971, 25, 79. 32. Morris, L. S.; Evans, J.; Marchesi, J. R.; J. Microbiol. Methods 2012, 91, 144. 33. Matsumoto, K.; Mizoue, K.; Kitamura, K.; Tse, W-C.; Huber, C. P.; Ishida, T.; Peptide Sci. 1999, 51, 99. 34. Silva, J. A.; Tese de doutorado, Universidade Federal de São Carlos, Brasil, 2011. |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access