Artigo
|
|
| Monitoramento da carga viral de SARS-CoV-2 em águas residuais na cidade de goiânia: epidemiologia baseada em esgoto e um sistema de alerta precoce para COVID-19 SARS-CoV-2 viral load in goiânia wastewater: wastewater-based epidemiology for a COVID-19 early warning system |
|
Geovana de Melo MendesI,#; Paulo Felipe Neves EstrelaI,#; Marcio Neres de Souza JuniorI; Núbia Natália de BritoI; Andréa Fernandes ArrudaI; Matheus Ribeiro AugustoII; Ieda Carolina Mantovani ClaroII; Adriana Feliciano Alves DuranII; Aline Diniz CabralII; Rodrigo de Freitas BuenoII; Gabriela Rodrigues Mendes DuarteI,*
I. Instituto de Química, Universidade Federal de Goiás, 74690-900 Goiânia - GO, Brasil Recebido em: 25/02/2022 *e-mail: gabriela_duarte@ufg.br COVID-19 is currently the most critical disease in the world. Thus, several tools have been used in the last two years to contain the spread of SARS-CoV-2. In this perspective, Wastewater-Based Epidemiology (WBE) provides valuable data by including symptomatic, pre-symptomatic, and asymptomatic individuals and anticipating clinical cases. For 37 weeks, the SARS-CoV-2 viral load was monitored in sanitary sewage samples from Goiânia-Brazil. Through pre-concentration, extraction, and purification of viral RNA, followed by RT-qPCR, it was detected in 83.78% (31/37) of the weeks the presence of fragments of the genetic material SARS-CoV-2 with viral loads ranging from 105 to 108 genome copies L-1. The viral load in the wastewater samples was related to the clinical data of the municipality, demonstrating its ability to promote an Early Warning System (EWS). In early 2022, it was possible to predict the increase in clinical cases 3 weeks in advance and issue an alert note to the health authorities. Prevalence estimates ranged from 0.003 to 0.5% and showed good agreement with observed values. Thus, monitoring effluents become another essential tool that can help combat the pandemic's advance, especially in countries with scarce resources. INTRODUÇÃO A pandemia da COVID-19 se tornou o maior desafio epidemiológico dos últimos dois anos, sendo considerada a pior crise sanitária deste século. Diante disso, a Organização Mundial de Saúde (OMS) tem estabelecido constante vigilância sobre os casos clínicos em todo o mundo. Segundo os seus dados, já foram contabilizados mais de 380 milhões de casos da doença, ocasionando mais de 5,6 milhões de mortes em todo o planeta. O Brasil, que até a presente data possui cerca 25 milhões de casos reportados, ocupa o 2° lugar no ranking global em número de mortes, com mais de 627 mil vidas perdidas, o que representa aproximadamente 10% de todas as mortes causadas pelo vírus em todo mundo.1 Nesse cenário de pandemia, muitas medidas vêm sendo adotadas com o objetivo de minimizar a disseminação do vírus, como por exemplo, o uso de máscaras faciais, distanciamento social, testagem em massa e campanhas de incentivo à vacinação.2-4 Dentre essas medidas, a testagem ocupa um lugar importante, pois além de fornecer informações relacionadas ao diagnóstico individual, permite também auxiliar, desde o isolamento de pacientes contaminados, passando pelo direcionamento de políticas públicas no manejo consciente de medidas impositivas de intervenção não farmacêuticas (NPIs), até a distribuição racionalizada de vacinas.5-7 Todavia, por meio das medidas de testagem convencionalmente adotadas por alguns países, isso é, com o direcionamento de testes apenas aos pacientes sintomáticos, tem-se a presença de dados inferiores em relação ao real número de pessoas contaminadas e, portanto, um equívoco da situação epidemiológica atualizada, uma vez que há subnotificação dos casos. Os casos são subnotificados, seja pelo déficit de testes ofertados pelo setor público, principalmente em países de recursos limitados, ou ainda, pela grande quantidade de casos assintomáticos.8 A técnica considerada padrão outro para testagem da COVID-19 é a RT-qPCR (do inglês: reverse transcription- quantitative polymerase chain reaction) que é um método de amplificação e detecção de fragmentos específicos de DNA. A amplificação enzimática do gene alvo se dá através de ciclos de aquecimento e resfriamento enquanto a detecção é baseada na medida de fluorescência no decorrer da reação. A técnica baseia-se no princípio básico de que o aumento significativo da quantidade de produto da PCR (amplificação do gene de interesse) está diretamente relacionado com o aumento da fluorescência, que é medida em tempo real. Além disso, a testagem convencional fornece apenas informações no que se refere ao status momentâneo da crise sanitária, sem tempo hábil para planejamento antecipado do gerenciamento da crise. Diante desse cenário, o monitoramento de fragmentos de ácidos nucleicos de SARS-CoV-2 (que pode ser relacionado à concentração do vírus) no esgoto, passa a ser uma importante ferramenta de saúde coletiva para o acompanhamento e antecipação do curso da pandemia, possibilitando a criação de um Sistema de Alerta Precoce (SAP).9,10 A utilização dessa ferramenta compõe o conjunto de estratégias definida por epidemiologia baseada no esgoto (WBE- do inglês Mates-bastardos Epidemiology). Em geral, fragmentos do RNA do vírus podem ser encontrados no esgoto, já que todas as pessoas contaminadas, sintomáticas ou assintomáticas, excretam o vírus através de seus fluidos biológicos.11 A Figura 1 demonstra o caminho que os fragmentos do vírus percorrem até a estação de tratamento de esgoto (ETE). De modo geral, a população infectada excreta o vírus presente em seus fluidos biológicos (fluídos nasais, orais e fecais), que irão percorrer o caminho até que seus fragmentos virias cheguem até a ETE, onde podem ser detectados. A ETE recebe contribuições de fragmentos virais tanto de pacientes sintomáticos quanto de pacientes assintomáticos.
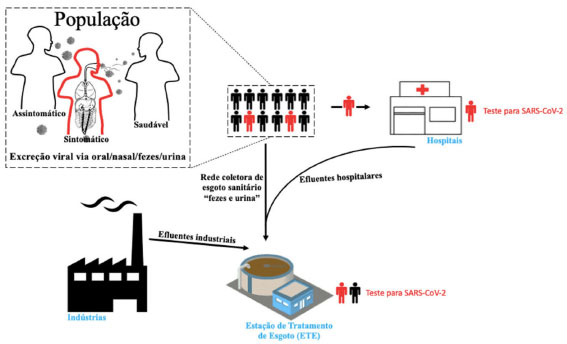 Figura 1. Desenho esquemático das principais contribuições em afluentes para uma ETE
A ferramenta WBE é de fundamental importância para a saúde pública pois serve para ampliar os dados obtidos pelos testes diagnósticos e, dessa forma, desempenha um papel crucial na otimização das tomadas de decisões de enfrentamento da pandemia, fornecendo informações relevantes tanto para fases de pós-isolamento quanto em casos de restabelecimento do isolamento da população após o aumento de casos da COVID-19. Uma das vantagens da abordagem WBE sobre os testes de diagnóstico clínico seria a capacidade de detectar o aumento de casos com antecedência. Brevemente, o conceito da epidemiologia baseada no esgoto fundamenta-se no princípio de rastrear biomarcadores no esgoto. Os biomarcadores são agentes químicos ou biológicos excretados pelo corpo humano, que podem ser coletados pela rede de esgoto e seguir até a estação de tratamento, e assim serem recuperados por métodos físico e/ou químicos. Para SARS-CoV-2, o material genético (RNA viral) tem sido amplamente aceito como biomarcador. Portanto, monitorar/quantificar as mudanças temporais nas concentrações de RNA viral presente em amostras de águas residuais comunitárias pode fornecer informações de saúde pública sendo capaz tanto de determinar a verdadeira extensão da infecção na população, quanto prever o surgimento de novos surtos virais.10,12 No Brasil, diversos grupos vêm realizando o monitoramento do SARS-CoV-2 em esgoto, mediante detecção de seus fragmentos.13-16 Nesse sentido, uma Rede de Monitoramento da COVID-19 em Águas Residuais foi criada em 2020, através da ação conjunta da Agência Nacional de Águas (ANA), Ministério de Ciência e Tecnologia e Inovações17 e Ministério da Saúde.18 A Rede de Monitoramento em Águas Residuais19 é uma iniciativa de combate ao avanço da pandemia de COVID-19 no Brasil, por meio de pesquisa epidemiológica baseada em monitoramento e mapeamento da carga viral em águas residuais, para elaboração de um sistema de alerta precoce para COVID-19. A Rede tem trabalhado na elaboração de boletins semanais de monitoramento epidemiológico em vários locais do Brasil, com o objetivo de informar a população e auxiliar as autoridades competentes na tomada de decisões em relação as ações de enfretamento da COVID-19. Na cidade de Goiânia, o Laboratório de Biomicrofluídica (IQ/UFG), que pertence a Rede, tem realizado esse monitoramento. Dessa forma, neste trabalho apresentamos os dados do monitoramento epidemiológico realizado de maio de 2021 a janeiro de 2022 em amostras da ETE Dr. Hélio Seixo de Britto em Goiânia-GO, a fim de acompanhar a circulação do vírus SARS-CoV-2, emitir alertas precoces e, assim, fornecer mais uma ferramenta auxiliar de enfrentamento à pandemia da COVID-19.
MATERIAIS E MÉTODOS Reagentes A água ultrapura para biologia molecular (livre de DNases, RNases e Proteases), o etanol PA, o tampão fosfato salino (PBS), a solução de fenol saturada com 0,1 mol L-1 de tampão de citrato (pH 4,3) e o polietilenoglicol-PEG 8000 foram adquiridos junto à Sigma-Aldrich (St Louis, MO). O cloreto de sódio PA foi obtido junto à Neon (Suzano, SP). Todos os oligonucleotídeos, incluindos os iniciadores da reação RT-qPCR (N1, N2 e RP) baseados no protocolo desenvolvido pela Centers for Disease Control and Prevention (CDC) e, também, o dsDNA de concentração conhecida utilizado para construção da curva analítica (Nº de catálogo 10006625 e 10006626), desenhados de acordo com a região dos primers, foram sintetizados junto à "Integrated DNA Technologies" - IDT (San Diego, CA). O kit de purificação de RNA e DNA viral DPK-115L foi adquirido pela Cellco Biotec (São Carlos, SP). O kit GoTaq Probe 1-Step RT-qPCR foi comprado junto à Promega (Madison, WI). Procedimento experimental As principais etapas para determinação da carga viral no esgoto estão descritas na Figura 2 e incluem: amostragem, preparo da amostra e detecção e quantificação por RT-qPCR. Em outras palavras, após a amostragem, a amostra foi concentrada utilizando PEG 8000 e em seguida o RNA foi extraído através de extração em fase sólida (SPE- do inglês: solid phase extraction) e na sequência foi realizado a RT-qPCR.
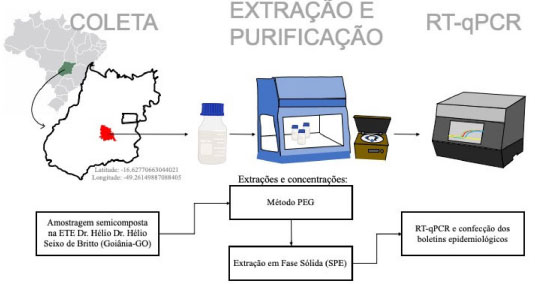 Figura 2. Principais etapas da determinação da carga de SARS-CoV-2 no esgoto da cidade de Goiânia
Amostragem As amostras de esgoto sanitário foram coletadas semanalmente (às quartas-feiras) no período de 5 de maio de 2021 a 20 de janeiro de 2022 (37 semanas - 18a a 52a semana epidemiológica de 2021 e 1a a 3a semana epidemiológica 2022) na estação de tratamento de esgoto (ETE) Dr. Hélio Seixo de Britto, localizada no município de Goiânia, Goiás, Brasil (-16.62770663044021, -49.26149887088405). A ETE Dr. Hélio Seixo de Britto foi escolhida porque recebe aproximadamente 70% do esgoto gerado na cidade de Goiânia/GO (na qual as bacias contribuintes são o ribeirão Anicuns e seus afluentes, o Ribeirão João Leite e o Caveirinha) tem capacidade de tratamento de até 2300 L s-1, recebendo uma vazão média de 1400 L s-1 de esgoto. Cada amostragem semanal foi realizada por meio da coleta e união de 10 alíquotas de 100 mL do efluente (correspondendo à amostragem semicomposta de 1000 mL), coletados em horários específicos igualmente espaçados, durante 18 horas, das 15 h às 9 h. Foi aferida a vazão para cada período amostral. Todas as amostras foram armazenadas em frascos previamente limpos e refrigeradas a temperatura de 4 °C desde a coleta até o horário da análise. Alíquotas de 50 mL de cada amostra semanal foram armazenadas a -80 °C como amostras para contraprova. Pré-concentração, extração e purificação de ácidos nucleicos em amostras de esgoto sanitário Os ácidos nucleicos presentes nas amostras foram concentrados e extraídos pelo método proposto por Claro et al.,15 com pequenas modificações, seguida pela purificação por extração em fase sólida (SPE) utilizando kit comercial. Resumidamente, 40 mL da amostra semicomposta foi incialmente centrifugada à 1500 xg por 250 min à -4° C na presença de 0,9 g de NaCl e 4,0 g de PEG 8000. Ao pellet formado, foram adicionados 0,4 mL de PBS 1X e 1 mL de fenol ácido que, após agitação, foi centrifugado à 13.000 xg por 10 minutos. A fase aquosa formada contendo os ácidos nucleicos foi submetida à extração em fase sólida utilizando o kit DPK-115L (Cellco Biotec, São Paulo), seguindo as recomendações do fabricante. Em todas as extrações, juntamente com a amostra foram realizados controles positivos através do enriquecimento de amostras de esgoto com RNA de SARS-CoV-2, para analisar a viabilidade da extração. Ao final do procedimento, os ácidos nucleicos extraídos e purificados foram imediatamente utilizados na RT-qPCR para quantificação viral do SARS-CoV-2. Quantificação viral Os ensaios de quantificação do SARS-CoV-2 foram realizados por meio da RT-qPCR (transcrição reversa seguida da Reação em Cadeia da Polimerase quantitativa) usando o kit GoTaq probe RT-qPCR one step (Promega, Wisconsin, EUA) no termociclador da Agilent AriaMx Real-Time PCR, baseado nos protocolos desenvolvidos pela Centers for Disease Control and Prevention (CDC). O protocolo desenvolvido pela CDC amplifica duas regiões distintas do SARS-CoV-2 (N1 e N2) e uma região do genoma humano (RP). O volume total de cada reação foi de 10 μL (5 μL GoTaq Probe qPCR Master Mix, 0,2 μL Go Script RT Mix, 1,55 μL água livre de nuclease, 0,75 μL Primers/Probe e 2,5 μL de amostra). Os primer/sondas usados para detecção visaram duas regiões distintas do nucleocapsídeo do novo coronavírus (N1 e N2) e uma região do DNA genômico humano, RNase P humana (controle interno). Inicialmente, para construção da curva analítica, foram realizadas diluições seriadas de 1,0x105 à 1,0 cópia μL-1 a partir de um fragmento de dsDNA contendo os alvos (N1 e N2) de concentração conhecida (padrão). Para cada concentração do fragmento de SARS-CoV-2 foram realizadas reações em triplicata e, ao final da RT-qPCR, correlacionamos cada concentração de SARS-CoV-2 com um ciclo de quantificação (Cq). O programa de amplificação consistiu em um ciclo de 45 ºC por 15 minutos para transcrição reversa, um ciclo de 95 ºC por 2 minutos para desnaturação inicial e, 50 ciclos de 95 ºC por 3 segundos e 55 ºC por 30 segundos, para amplificação. Para quantificação das amostras foi realizado a RT-qPCR, conforme descrito anteriormente. Todas as misturas reacionais foram testadas com controles positivos e negativos simultaneamente com as amostras. As amostras foram consideradas positivas quando apresentaram Cq < 40.18-21 O valor de Cq (do inglês Cycle quantification) é dependente do número inicial de moléculas de DNA disponíveis para amplificação. Quanto mais moléculas de DNA disponíveis para amplificação menos ciclos de aquecimento e resfriamento são necessários para detectar o gene alvo presente no meio. Desta forma, quanto maior a carga viral menor será o valor de Cq. Igualmente, quanto maior o valor de Cq menor a carga viral e por isso para valores de Cq> 40 a amostra é considerada negativa, como já estabelecido em outros trabalhos na literatura científica.9 A quantificação em cópias virais para cada amostra foi realizada por meio da equação da reta fornecida pela curva analítica. Estimativa de prevalência A população infectada da cidade de Goiânia e as estimativas de prevalência foram calculadas a partir da carga viral do SARS-CoV-2 quantificada nos esgotos e nos outros parâmetros apresentados nas equações 1 e 2, conforme anteriormente relatado por Claro e colaboradores.15  A concentração de RNA viral presente nas águas residuárias (CRNA, em cópias L-1) foi determinada como descrito anteriormente. A vazão volumétrica de esgoto (Q, em L.d-1) foi medida na própria ETE Dr. Hélio Seixo de Britto no momento da coleta das amostras. O valor de α se refere a massa fecal diária produzida por um ser humano em países de baixa renda, variando de 75 a 520 gramas por pessoa (com valor médio de 243 ± 130,2 g.pessoa-1.d-1).20 O valor de β se refere a quantidade de material genético do SARS-CoV-2 eliminada por um indivíduo infectado, que geralmente varia de 6,3 x 105 a 1,3 x 108 cópias g-1.11,21 Para calcular a estimativa da prevalência média do vírus SARS-CoV-2 foi considerando a população contribuinte da ETE Dr. Hélio Seixo de Britto (aproximadamente 1.100.000 habitantes, o que corresponde a 70% da população de Goiânia). A carga viral excretada por um indivíduo infectado (produto α x β) apresenta elevada variabilidade (3 logs ou mais), o que aumenta a imprecisão dos cálculos de estimativa de prevalência. Dessa forma, foi implementado o modelo estatístico probabilístico de Monte-Carlo com 10 mil amostras aleatórias, considerando-se um intervalo de confiança de 90%. Os valores e o tipo de distribuição utilizados para cada um dos parâmetros estão apresentados na Tabela 1.
RESULTADOS E DISCUSSÕES Detecção de SARS-CoV-2 em amostras de esgoto sanitário Conforme os dados já apresentados anteriormente, as amostras de esgoto sanitário, coletadas semanalmente, representam a contribuição de cerca de um milhão de pessoas e, portanto, trata-se de uma amostra representativa para a realidade do município, refletindo o comportamento da maioria dos casos clínicos reportados. O presente estudo descreve o monitoramento de longo prazo da ocorrência de fragmentos do RNA do SARS-CoV-2 no esgoto sanitário da cidade de Goiânia durante o período de 5 maio de 2021 (18a semana epidemiológica de 2021) até 20 de janeiro de 2022 (3a semana epidemiológica de 2022). Dentre as 37 semanas avaliadas, em 31 semanas (83,78%) foi detectada a presença de fragmentos do material genético do SARS-CoV-2 nas amostras de esgoto sanitário do ponto de monitoramento ETE Dr. Hélio Seixo de Britto. A Figura 3 mostra quais regiões do gene foram detectadas (N1, N2 ou N1 e N2) em cada uma das 37 semanas, assim como a faixa de Cq encontrada em cada semana epidemiológica.
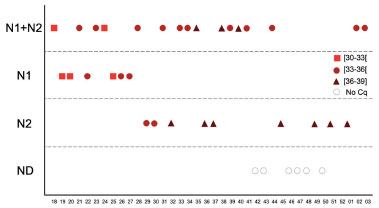 Figura 3. Região do SARS-CoV-2 (N1/N2) amplificada por RT-qPCR em cada semana epidemiológica analisada, estratificada entre No Cq e em 3 faixas de Cq [30-33[, [33-36[ e [36-39]. N1: região do nucleocapsídeo 1; N2: região do nucleocapsídeo 2; Cq: ciclo de quantificação; No Cq: ausência de ciclo de quantificação; ND: não detectado
Recentemente, muitos trabalhos realizados com amostras de ETE tem demonstrado a detecção de SARS-CoV-2 no esgoto sanitário e uma correlação com os casos clínicos.22-24 Nesse trabalho, dentre as semanas epidemiológicas avaliadas, em 51,61% das vezes foram detectados fragmentos de RNA do SARS-CoV-2 em ambas as regiões N1 e N2. Em contrapartida, em 19,35% das semanas o alvo foi detectado apenas na região N1 e, em 29,04% apenas na N2 (Figura 3). É possível observar através da Figura 3 uma tendência preferencial de amplificação da região de acordo com a data da análise (semana epidemiológica). De maio a julho (18a semana até a 27a semana de 2021) observou-se que em 72,73% das semanas, as amostras amplificaram preferencialmente na região N1, ainda que neste mesmo intervalo de tempo houvesse amplificação dos genes N1+N2, a amplificação dos dois genes só ocorreu em 27,23% das semanas. As diferenças entre os ensaios de N1 e N2 em amostras de águas residuais também foram relatadas por outros estudos recentes.24,25 Em um estudo publicado por Ai e colaboradores,26 por meio de análises estatísticas, os autores correlacionaram os casos de COVID-19 da região analisada e a concentração de 3 genes do SARS CoV-2 (N1, N2 e E) em amostras de águas residuais. As concentrações de todos os três genes do SARS-CoV-2 foram correlacionadas positivamente com os casos confirmados diários para quase todos os locais. Nesse estudo, a região N2 obteve o melhor desempenho. A robustez da região N2 na PCR quantitativa também é mostrada em outros estudos com águas residuais, relatando que a região N2 é menos propensa a mutações e, portanto, de detecção preferencial em momentos que ocorre a prevalência de circulação de variantes.26-28 Os dados da Figura 3 demonstram que, nesse estudo, a tendência em amplificar a região N2 iniciou-se a partir da 44a semana de 2021 e permaneceu até a 1ª semana de 2022. As amostras analisadas nas 37 semanas foram estratificadas em 3 intervalos de Cq (30-33, 33-36 e 36-39) e No Cq. Dentre as semanas epidemiológicas nas quais o RNA do vírus foi detectado, 16,13% das amostras apresentaram valores de Cq no intervalo de 30-33, 51,61% estão no intervalo de 33-36 e 32,26% estão no intervalo de 36-39. O intervalo de maior concentração viral (30-33) se concentra nas primeiras semanas epidemiológicas analisadas (18a a 25a semana epidemiológica de 2021), em concordância ao alto número de casos de clínicos no município de Goiânia. Em contrapartida, desde a 42a semana observou-se uma tendência de diminuição da concentração viral, com predominância de Cq no intervalo de 36-39, sendo que em algumas semanas não foi detectada a presença dos vírus (semanas 42, 43, 46, 47, 48, 50). Contudo, esse cenário se altera a partir da segunda e terceira semanas epidemiológicas de 2022, em que foi observado uma mudança no intervalo de Cq para 33-36, em uma perspectiva de aumento da concentração de SARS-CoV-2 no esgoto, indicando o aumento de casos clínicos de COVID-19 no município. Quantificação de SARS-CoV-2 em água residuais e casos clínicos A quantificação exata e confiável do vírus é o principal objetivo do WBE porque são justamente estes resultados que podem indicar o início potencial de futuras ondas e/ou epidemias. A RT-qPCR foi realizada seguindo as instruções do fabricante, como já descrito anteriormente. Cada RNA extraído foi analisado em triplicata. Foi realizada uma curva analítica para cada gene alvo (N1 e N2). Uma série de controles positivos (cinco diluições em série de 10 vezes) e negativos foram incluídos para cada RT-qPCR executado pelo alvo do genoma N1 e N2. As curvas analíticas para N1 (y =-3,221x+34,55; R2 = 0,998; Eficiência =99%) e N2 (y =-3,683x + 37,74; R2 = 0,991; Eficiência =86,85%) mostraram uma dinâmica linear e o limite de detecção (LOD) foi de 5,0 cópias do genoma. A Figura 4 apresenta os resultados do monitoramento de casos clínicos e das concentrações dos fragmentos do RNA viral no esgoto ao longo do período de monitoramento. Na Figura 4b, as concentrações são classificadas em 5 níveis: muito baixa (102 103), baixa (103-104), média (104-107), alta (107-109) e muito alta (109 1010). A definição dessas faixas de concentração foi baseada no conjunto histórico de dados desta pesquisa e de outros trabalhos conduzidos pelo nosso grupo de pesquisa.15 Com os dados de 1 ano de monitoramento do esgoto e com as informações da vigilância epidemiológica (casos clínicos notificados, número de internações e número de óbitos), foi possível verificar alguns padrões e estabelecer as faixas de concentração. De maneira geral, verificaram-se as concentrações consideradas altas (107-109) nos momentos mais críticos da pandemia (maior número de notificações da COVID-19), e as concentrações baixas (103-104) nos momentos menos críticos.
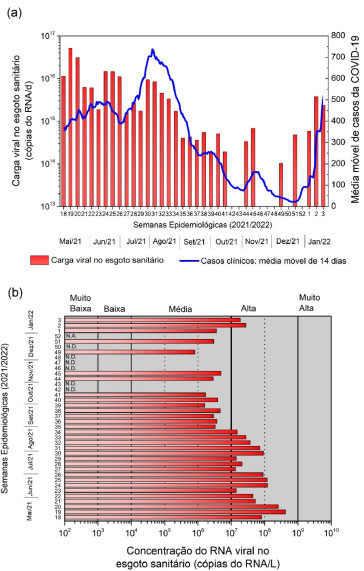 Figura 4. (a) Relação entre a carga viral de SARS-CoV-2 quantificada nas amostras de águas residuais e a média móvel dos casos clínicos na cidade de Goiânia; (b) Concentração do RNA viral no esgoto. ND: não detectado, NA: não analisado
Os dados de concentração viral no esgoto sanitário foram comparados com os dados clínicos (média móvel de 14 dias) de casos de infecção por SARS-CoV-2 da cidade de Goiânia, disponibilizados pela Secretaria Municipal de Saúde,29 como mostra a Figura 4a. Entre a 18a e 41a semana foi possível detectar a presença do RNA viral em 100% das amostras de águas residuais, sendo que entre as semanas 18 e 34 a carga viral que oscilou entre 1015 e 1016 cópias d-1, apresentou baixa variabilidade, assim como os dados epidemiológicos/clínicos. A concentração viral detectada naquele momento manteve-se em altos níveis oscilando entre 107 e 108 cópias de RNA L-1 (Figura 4b). No entanto, a partir da semana 35 observou-se uma tendência de queda na concentração do material genético do vírus no esgoto. Da mesma forma, a média móvel de casos clínicos da COVID-19 sofreu gradativa redução. A tendência de queda da carga viral no esgoto manteve-se até a primeira semana epidemiológica de 2022. Nesse período, verificaram-se as concentrações do RNA viral no esgoto sanitário em níveis médios (entre 104 e 106 cópias L-1) ou inferiores ao limite de detecção. A partir da segunda semana epidemiológica de 2022 foi observado um aumento significativo na concentração do RNA viral, que voltou a subir para patamares considerados altos (acima de 107 cópias L-1), correspondendo a maior concentração viral desde 18 de agosto de 2021. Desta forma, através da Figura 4a, observamos que os dados do esgoto apresentaram uma aparente concordância com os dados clínicos, sendo que supostamente o primeiro precedeu o segundo e nas últimas semanas epidemiológicas este efeito pode ser observado de forma mais evidente. Essas informações obtidas através do monitoramento epidemiológico são importantes pois representam uma ferramenta que permite com que agências e autoridades competentes assumam ações de vigilância epidemiológica na prevenção e controle da COVID-19. Um Sistema de Alerta Precoce (SAP) eficaz pode ser usado para identificar os pontos críticos da COVID-19 e orientar a execução e a distribuição de recursos, incluindo estratégias de testagem em massa, ampliação de assistência hospitalar e, de forma geral, rastreamento e preparo para o enfrentamento dos surtos virais.15,30,31 Com base nos dados obtidos, semanalmente foram emitidos informes via boletins epidemiológicos de livre acesso no site do Ministério da Ciência, Tecnologia e Inovações,17 relatando a carga viral presente nas amostras de águas residuais do município de Goiânia, bem como a comparação com os dados de cargas virais das semanas epidemiológicas anteriores. Além disso, diante do recente aumento na carga viral, como o ocorrido no início de 2022, foi emitido uma nota de alerta e um comunicado oficial à Secretaria de Saúde do município de Goiânia visando antecipação das ações de controle sanitário para o enfrentamento da COVID-19 devido ao possível aumento de casos clínicos nas semanas seguintes. Outro importante aspecto observado foi a relação da carga viral no esgoto sanitário com a taxa de vacinação da população goianiense e a entrada da variante Ômicron no município. A Figura 5 apresenta a variação da carga viral ao longo do tempo em relação ao avanço da imunização na cidade Goiânia.
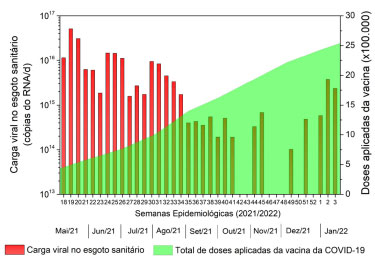 Figura 5. Relação entre a carga viral de SARS-CoV-2 quantificada nas amostras de águas residuais na cidade de Goiânia e número de doses aplicadas da vacina da COVID-19 no município
Como pode ser observado na Figura 5, o avanço da imunização refletiu na diminuição da carga viral de SARS-CoV-2 no esgoto de Goiânia, desde maio, até o início de dezembro de 2021. Todavia, no início de 2022 foi observado uma re-emergência da carga viral de SARS-CoV-2 no esgoto, provavelmente pela inserção da nova variante (ômicron) no município. Yaniv et al.32 monitoraram traços de SARS CoV-2 nas águas residuais de Beer-Sheva e observaram que após 50% da população ter sido vacinada, houve uma diminuição constante e consistente no nível de RNA de SARS-CoV-2 detectado em águas residuais, até que ficasse abaixo das concentrações detectáveis. Os autores também relataram que, mesmo a população apresentando alto índice de vacinação, o surgimento de uma nova variante no município, repercutiu em um aumento da carga viral no esgoto, assim como foi observado neste estudo. Embora a vacinação da população seja um fator preponderante para diminuição dos casos de COVID-19 e, consequentemente, da carga viral de SARS-CoV-2 no esgoto, a tendência de diminuição da carga viral no esgoto sanitário não pode ser relacionada exclusivamente com o progresso da imunização. Outros fatores como, por exemplo, as medidas restritivas de isolamento social, a complexidade da matriz biológica e os fatores ambientais podem estar correlacionados com o decréscimo da carga viral observada no esgoto dentro do período analisado. Prevalência estimada A Tabela 2 e a Figura 6 apresentam os resultados das estimativas de prevalência por mês de monitoramento, assim como os valores observados (obtidos a partir dos dados epidemiológicos do município). Os valores modelados (preditos) são apresentados como mediana e intervalo de confiança, considerando-se um nível de significância de 90% (p < 0,1). Na tabela também são apresentados os valores médios, mínimos e máximos da prevalência observada na região, determinada a partir dos casos clínicos notificados da COVID-19.
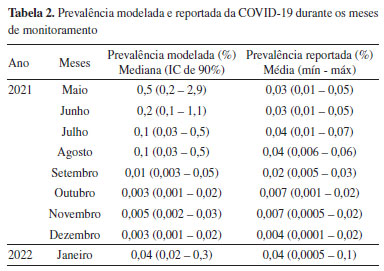
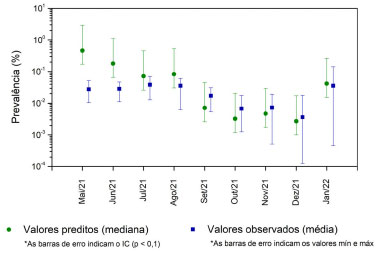 Figura 6. Relação entre as prevalências modeladas e observadas
Em geral, as prevalências modeladas apresentaram boa concordância com as reportadas. Nos meses de maio a agosto de 2021, os valores preditos foram até 10 vezes superiores aos valores observados/reportados. No entanto, nos meses subsequentes, os valores preditos foram equivalentes aos reportados, sobretudo quando os intervalos de confiança foram considerados na análise comparativa. Esse comportamento pode estar associado à redução da carga viral no esgoto, após os 4 primeiros meses (Figura 4a). Quanto maiores os valores da carga viral no esgoto (numerados da equação 1), maior será o erro associado ao modelo de estimativa de prevalência.33 Houve uma variação de 1 a 2 logs entre o início e após 4 meses de monitoramento. Esses resultados vão de encontro com outros resultados já relatados pela literatura científica, reportados para amostras de esgoto sanitário de regiões diferentes da região estudada aqui. Em Massachusetts - EUA, Wu e colaboradores22 estimaram valores de prevalência entre 0,1 e 5%, muito superiores aos valores observados na região a partir dos dados clínicos (aproximadamente 0,026%). Na região do ABC Paulista, em São Paulo, Claro et al.15 também obtiveram valores preditos cerca de 10 vezes superiores aos observados, para amostras de esgoto coletadas entre junho de 2020 e abril de 2021. Incertezas e limitações A amostragem para aplicações em WBE oferece desafios espaciais e temporais que devem ser considerados para que os resultados obtidos sejam representativos da população em estudo. Por exemplo, as diferenças entre os sistemas de águas residuais urbanas e rurais devem ser consideradas uma vez que sistemas de esgoto urbano podem fornecer amostras mais representativas da comunidade do que os sistemas rurais. Outro aspecto relevante é o tempo de amostragem, que deve considerar parâmetros como os reservatórios ambientais, locais onde o vírus é mais facilmente transportado e transmitido, o tamanho da área de captação e a possibilidade de mudanças diurnas no influxo e/ou taxas de detecção viral.34,35 Uma vez que o RNA viral é liberado pelos indivíduos infectados, ele entra na rede de esgoto e se mistura com o restante das águas residuais. Basicamente, o fator de diluição pode ser determinado assumindo uma mistura completa e homogênea do RNA viral liberado por todos os indivíduos em um dia e a vazão diária de esgoto que geralmente é obtida do operador da rede de esgoto. Mas, na prática, o processo de mistura e diluição é significativamente influenciado pela vazão diurna desigual das águas residuais e pelo tempo de descarga do vaso sanitário. Portanto, entre os muitos aspectos importantes a serem considerados destaca-se a amostragem realizada com o auxílio de amostradores automáticos, que podem são programados para realizar amostragens compostas durante um período representativo, por exemplo, 24 h usando um amostrador proporcional ao fluxo. Embora os amostradores automáticos forneçam confiabilidade e segurança, as unidades refrigeradas necessárias para prevenir a degradação viral são caras, muitos destes ainda são importados e o valor tende a inviabilizar a sua aquisição em alguns locais. Por isso, em muitas situações, a amostragem composta realizada sem o amostrador automático pode representar a abordagem mais prática, apropriada e acessível.33,35 Outra fonte de incerteza a ser destacada é a qualidade do RNA viral. A extração e concentração de amostras de efluentes geralmente contêm diversos inibidores de PCR, incluindo gorduras, proteínas e substâncias húmicas, que interferem na reação de PCR. No entanto, para vírus de RNA, como SARS-CoV-2, a RT-qPCR ainda é o método padrão ouro para obter dados quantitativos confiáveis. Em alguns casos, esses inibidores podem ser minimizados diluindo as amostras de RNA.34,35 Conforme recomendado pelo Centro de Controle e Prevenção de Doenças dos Estados Unidos,36 as estimativas de prevalência não devem ser utilizadas de forma isolada para subsidiar as tomadas de decisão, uma vez que ainda existem muitas incertezas nos cálculos. A carga viral excretada por um indivíduo infectado através das fezes (denominador da equação 1) pode variar em até 5 logs.37,38 Os valores de β (carga viral excretada nas fezes) utilizadas neste estudo foram os mesmos utilizados por Kitajima et al.11 entre 5,79 e 8,11 log10 cópias do genoma g fezes-1. No entanto, essa faixa de variação pode ser ainda maior, entre 2,56 e 7,67 log10 cópias do genoma g fezes-1.38 Outra limitação é que o modelo utilizado considera a liberação do RNA viral apenas pelas fezes. No entanto, estudos recentes mostram outras vias importantes de contribuição, como o escarro, produzido pela expectoração. Como o SARS-CoV-2 é um vírus respiratório infeccioso, é detectado principalmente em amostras do trato respiratório (70 a 100%). De acordo com Li et al.,39 a liberação de escarro nos esgotos pode aumentar as concentrações de fragmentos de RNA do SARS-CoV-2 em amostras de esgoto em até 70 vezes. No entanto, essa fonte ainda não tem sido considerada em estudos com modelagem de prevalência. O esgoto sanitário é uma matriz complexa, que fornece muitas informações sobre os hábitos e características de uma população, apresentando variada composição biológica (associada a dejetos humanos, de animais de estimação, de animais sinantrópicos, entre outros), além de compostos químicos (orgânicos e inorgânicos) oriundos de residências e efluentes industriais.15,40 Outras variáveis relevantes que devem ser consideradas são as condições ambientais, principalmente a temperatura em países de clima quente. A riqueza de constituintes químicos e biológicos presentes na matriz, aliada à processos intempéricos (temperatura e radiação solar), corroboram para degradação e, portanto, decaimento da carga viral em amostras de esgoto sanitário. Além disso, os estudos podem ser conflitantes no que se refere a como ocorre a eliminação do vírus através de um indivíduo infectado. Alguns estudos apontam que apenas 60-70% dos indivíduos infectados excretam RNA de SARS-CoV-2. Outros estudos mostraram que mesmo portadores assintomáticos e não diagnosticados também excretam o RNA de SARS-CoV-2. Também não há um consenso em qual a concentração viral excretada pelos indivíduos infectados.22,41 Além disso, a carga viral quantificada nas amostras de águas residuais é função dos métodos de amostragem, pré-concentração, extração e quantificação do RNA viral adotados por cada grupo de pesquisa, e por consequência, das incertezas desses métodos utilizados. Diante dessa realidade, as comparações absolutas entre a prevalência observada de COVID-19 e as concentrações de RNA SARS-CoV-2 em águas residuais são complexas. Todavia, por mais que seja reconhecidamente inexata devido à grandes incertezas associadas, a prevalência estimada pode fornecer um direcionamento e, portanto, ser utilizada como ferramenta de vigilância epidemiológica e combate à pandemia da COVID-19, considerando as suas limitações.
CONCLUSÕES A epidemiologia baseada no esgoto (WBE) é uma importante ferramenta que pode auxiliar no combate ao avanço da pandemia da COVID-19, uma vez que fornece informações essenciais sobre o nível de infecção da população analisada mais próxima do real, ao considerar os casos totais (sintomáticos, pré-sintomáticos e assintomáticos). Além disso, a antecipação dos casos clínicos fornece em tempo hábil, a possibilidade de tomada de decisões de políticas públicas. Dessa forma, a WBE torna-se especialmente útil em países com recursos escassos, que não possuem viabilidade de realizar testagens em massa periodicamente, uma vez que, na WBE, com poucos testes tem-se importantes dados da situação infeciosa da população. As informações obtidas neste estudo possibilitam a criação de um sistema de alerta precoce permitindo as autoridades responsáveis antecipação nas medidas de enfrentamento dos surtos virais. Contudo, ainda que os resultados apresentados neste trabalho sejam consistentes, é preciso cuidado ao analisá-los considerando a complexidade da matriz em questão, o esgoto sanitário, bem como os erros e incertezas dos métodos utilizados nas etapas de amostragem, pré-concentração, extração e quantificação do RNA. Mesmo levando em consideração as incertezas associadas, a prevalência estimada fornecer um direcionamento e por isso, portanto é uma ferramenta de vigilância epidemiológica e combate à pandemia da COVID-19. Ao analisar semanalmente as águas residuais de esgoto sanitário no município de Goiânia, detectamos em 83,78% (31/37) das semanas a presença de fragmentos do material genético do SARS CoV-2 e, observou-se uma tendência preferencial de amplificação da região do nucleocapsídeo viral de acordo com a semana epidemiológica, que condiz com a incidência de variantes no município. Quantitativamente, a carga viral variou de 1014 a 1016 cópias do genoma/dia, com exceção das semanas 42, 43, 46, 47, 48 e 50, em que não foi detectado a presença de RNA de SARS-CoV-2 no esgoto. Ademais, a estimativa de prevalência realizada indicou boa concordância entre os dados preditos e observados. Os valores estimados pelo modelo são até 10 vezes superiores aos valores observados. Através do monitoramento epidemiológico, os níveis de carga viral de SARS-CoV-2 no esgoto sanitário de Goiânia vem sendo disponibilizados gratuitamente por meio de boletins epidemiológicos publicados na página do MCTI desde 5 de maio de 2021. Esses dados fornecem à população e, principalmente, aos gestores públicos, uma ferramenta a mais de combate ao avanço da pandemia e, dessa forma, corroboram ativamente para saúde coletiva.
AGRADECIMENTOS Os autores agradecem o apoio financeiro do CNPq (processo: 402694/2020-1 e 402432/2020-7), FAPEG (processo: 202010267000273) e Ministério Público do Trabalho. Os autores agradecem à equipe da SANEAGO, ao Felipe João Carvalho Filho e ao Carlos R. A. dos Santos, que realizaram as coletas das amostras de esgoto. Esse é um trabalho desenvolvido e apoiado pela Rede de Monitoramento de COVID-19 em Águas Residuais - Agência Nacional de Águas e Saneamento Básico (ANA), Ministério da Ciência, Tecnologia e Inovações (MCTI) e Ministério da Saúde (MS).
REFERÊNCIAS 1. World Health Organization. Emergency Dashboard COVID-19. 2022. 2. Wong, M. C. S.; Ng, R. W. Y.; Chong, K. C.; Lai, C. K. C.; Huang, J.; Chen, Z.; Boon, S. S.; Chan, P. K. S.; BMJ Glob. Heal. 2020, 5, 1. [Crossref] 3. Prem, K.; Liu, Y.; Russell, T. W.; Kucharski, A. J.; Eggo, R. M.; Davies, N.; Flasche, S.; Clifford, S.; Pearson, C. A. B.; Munday, J. D.; Abbott, S.; Gibbs, H.; Rosello, A.; Quilty, B. J.; Jombart, T.; Sun, F.; Diamond, C.; Gimma, A.; van Zandvoort, K.; Funk, S.; Jarvis, C. I.; Edmunds, W. J.; Bosse, N. I.; Hellewell, J.; Jit, M.; Klepac, P.; Lancet Public Heal. 2020, 5, e261. [Crossref] 4. Gaythorpe, K. A. M.; Abbott, S.; Prem, K.; Liu, Y.; Bhatia, S.; Elsland, S. Van; Ferguson, N. M.; Wellcome Open Res. 2020, 59, 1. [Crossref] 5. Araz, O. M.; Ramirez-Nafarrate, A.; Jehn, M.; Wilson, F. A.; Heal. Syst. 2020, 9, 119. [Crossref] 6. Cheng, Z. J.; Zhan, Z.; Xue, M.; Zheng, P.; Lyu, J.; Ma, J.; Zhang, X. D.; Luo, W.; Huang, H.; Zhang, Y.; Wang, H.; Zhong, N.; Sun, B.; Clin. Rev. Allergy Immunol. 2021. [Crossref] 7. Smith, T.; Cassell, G.; Bhatnagar, A.; JAMA Heal. Forum 2021, 2, e201616. [Crossref] 8. Biswas, R. K.; Afiaz, A.; Huq, S.; Epidemiol. Infect. 2020, 148, e207. [Crossref] 9. Pezzi, L.; Charrel, R. N.; Ninove, L.; Nougairede, A.; Molle, G.; Coutard, B.; Durand, G.; Leparc-Goffart, I.; De Lamballerie, X.; Thirion, L.; Viruses 2020, 12, 1. Pezzi, L.; Charrel, R. N.; Ninove, L.; Nougairede, A.; Molle, G.; Coutard, B.; Durand, G.; Leparc-Goffart, I.; De Lamballerie, X.; Thirion, L.; Viruses 2020, 12, 1. [Crossref] 10. Bibby, K.; Bivins, A.; Wu, Z.; North, D.; Water Res. 2021, 202, 117438. [Crossref] 11. Kitajima, M.; Ahmed, W.; Bibby, K.; Carducci, A.; Gerba, C. P.; Hamilton, K. A.; Haramoto, E.; Rose, J. B.; Sci. Total Environ. 2020, 739, 139076. [Crossref] 12. Sodré, F. F.; Brandão, C. C. S.; Vizzotto, C. S.; Maldaner, E. A. O.; Quim. Nova 2020, 43, 515. [Crossref] 13. Fongaro, G.; Stoco, P. H.; Souza, D. S. M.; Grisard, E. C.; Magri, M. E.; Rogovski, P.; Schörner, M. A.; Barazzetti, F. H.; Christoff, A. P.; de Oliveira, L. F. V.; Bazzo, M. L.; Wagner, G.; Hernández, M.; Rodríguez-Lázaro, D.; Sci. Total Environ. 2021, 778, 1. [Crossref] 14. 14. Prado, T.; Fumian, T. M.; Mannarino, C. F.; Resende, P. C.; Motta, F. C.; Eppinghaus, A. L. F.; Chagas do Vale, V. H.; Braz, R. M. S.; de Andrade, J. da S. R.; Maranhão, A. G.; Miagostovich, M. P.; Water Res. 2021, 191, 116810. [Crossref] 15. Claro, I. C. M.; Cabral, A. D.; Augusto, M. R.; Duran, A. F. A.; Graciosa, M. C. P.; Fonseca, F. L. A.; Speranca, M. A.; Bueno, R. de F.; Water Res. 2021, 203, 1. [Crossref] 16. Mota, C. R.; Bressani-Ribeiro, T.; Araújo, J. C.; Leal, C. D.; Leroy-Freitas, D.; Machado, E. C.; Espinosa, M. F.; Fernandes, L.; Leão, T. L.; Chamhum-Silva, L.; Azevedo, L.; Morandi, T.; Freitas, G. T. O.; Costa, M. S.; Carvalho, B. O.; Reis, M. T. P.; Melo, M. C.; Ayrimoraes, S. R.; Chernicharo, C. A. L.; Water Res. 2021, 202. [Crossref] 17. MCTI- Ministério da Ciência Tecnologia e Inovação, 2022. 18. MS-Ministério da Saúde https://www.gov.br/saude/pt-br. 2022. 19. VIRUS, R.; Rede de Monitoramento em Águas Residuais http://redevirus.mcti.gov.br. 2022. 20. Rose, C.; Parker, A.; Jefferson, B.; Cartmell, E.; Crit. Rev. Environ. Sci. Technol. 2015, 45, 1827. [Crossref] 21. Gholipour, S.; Mohammadi, F.; Nikaeen, M.; Shamsizadeh, Z.; Khazeni, A.; Sahbaei, Z.; Mousavi, S. M.; Ghobadian, M.; Mirhendi, H.; Chemosphere 2021, 273, 129701. [Crossref] 22. Wu, F.; Zhang, J.; Xiao, A.; Gu, X.; Lee, L.; Armas, F.; Kauffman, K.; mSystems 2020, 5, 1. [Crossref] 23. Wurtzer, S.; Marechal, V.; Mouchel, J. M.; Maday, Y.; Teyssou, R.; Richard, E.; Almayrac, J. L.; Moulin, L.; medRxiv 2020. [Crossref] 24. Randazzo, W.; Truchado, P.; Cuevas-Ferrando, E.; Simón, P.; Allende, A.; Sánchez, G.; Water Res. 2020, 181. [Crossref] 25. Medema, G.; Heijnen, L.; Elsinga, G.; Italiaander, R.; Brouwer, A.; Environ. Sci. Technol. Lett. 2020, 7, 511. [Crossref] 26. Ai, Y.; Davis, A.; Jones, D.; Lemeshow, S.; Tu, H.; He, F.; Ru, P.; Pan, X.; Bohrerova, Z.; Lee, J.; Sci. Total Environ. 2021, 801, 149757. [Crossref] 27. Gonzalez, R.; Curtis, K.; Bivins, A.; Bibby, K.; Weir, M. H.; Yetka, K.; Thompson, H.; Keeling, D.; Mitchell, J.; Gonzalez, D.; Water Res. 2020, 186, 116296. [Crossref] 28. Rahman, M. S.; Islam, M. R.; Alam, A. S. M. R. U.; Islam, I.; Hoque, M. N.; Akter, S.; Rahaman, M. M.; Sultana, M.; Hossain, M. A.; J. Med. Virol. 2021, 93, 2177. [Crossref] 29. SMS- Secretaria Municipal de Saúde de Goiânia https://saude.goiania.go.gov.br/. 2022. 30. Mlejnkova, H.; Sovova, K.; Vasickova, P.; Ocenaskova, V.; Jasikova, L.; Juranova, E.; Int. J. Environ. Res. Public Health 2020, 17, 1. [Crossref] 31. Ahmed, W.; Bertsch, P. M.; Bivins, A.; Bibby, K.; Farkas, K.; Gathercole, A.; Haramoto, E.; Gyawali, P.; Korajkic, A.; McMinn, B. R.; Mueller, J. F.; Simpson, S. L.; Smith, W. J. M.; Symonds, E. M.; Thomas, K. V.; Verhagen, R.; Kitajima, M.; Sci. Total Environ. 2020, 739, 139960. [Crossref] 32. Yaniv, K.; Ozer, E.; Lewis, Y.; Kushmaro, A.; Water Res. 2021, 207, 117808. [Crossref] 33. Augusto, M. R.; Claro, I. C. M.; Siqueira, A. K.; Sousa, G. S.; Caldereiro, C. R.; Duran, A. F. A.; de Miranda, T. B.; Bomediano Camillo, L. de M.; Cabral, A. D.; de Freitas Bueno, R.; J. Environ. Chem. Eng. 2022, 10. [Crossref] 34. Gwenzi, W.; Sci. Total Environ. 2022, 806, 150680. [Crossref] 35. Polo, D.; Quintela-Baluja, M.; Corbishley, A.; Jones, D. L.; Singer, A. C.; Graham, D. W.; Romalde, J. L.; Water Res. 2020, 186, 116404. [Crossref] 36. CDC; Wastewater Surveillance Data Reporting and Analytics, Centers Dis. Control Prev https://www.cdc.gov/healthywater/surveillance/wastewater-surveillance/data-reporting-analytics.html. 2022. 37. Saththasivam, J.; El-Malah, S. S.; Gomez, T. A.; Jabbar, K. A.; Remanan, R.; Krishnankutty, A. K.; Ogunbiyi, O.; Rasool, K.; Ashhab, S.; Rashkeev, S.; Bensaad, M.; Ahmed, A. A.; Mohamoud, Y. A.; Malek, J. A.; Abu Raddad, L. J.; Jeremijenko, A.; Abu Halaweh, H. A.; Lawler, J.; Mahmoud, K. A.; Sci. Total Environ. 2021, 774, 145608. [Crossref] 38. Wölfel, R.; Corman, V. M.; Guggemos, W.; Seilmaier, M.; Zange, S.; Müller, M. A.; Niemeyer, D.; Jones, T. C.; Vollmar, P.; Rothe, C.; Hoelscher, M.; Bleicker, T.; Brünink, S.; Schneider, J.; Ehmann, R.; Zwirglmaier, K.; Drosten, C.; Wendtner, C.; Nature 2020, 581, 465. [Crossref] 39. Li, X.; Kulandaivelu, J.; Guo, Y.; Zhang, S.; Shi, J.; O'Brien, J.; Arora, S.; Kumar, M.; Sherchan, S. P.; Honda, R.; Jackson, G.; Luby, S. P.; Jiang, G.; J. Hazard. Mater. 2022, 432, 128667. [Crossref] 40. Cabral, A. D.; Claro, I. C. M.; Augusto, M. R.; Friolani, V. N.; Bezerra, C. de E.; Graciosa, M. C. P.; Fonseca, F. L. A.; Speranca, M. A.; Bueno, R. de F.; Eng. Sanit. e Ambient. 2021, 26, 1043. [Crossref] 41. Zheng, S.; Fan, J.; Yu, F.; Feng, B.; Lou, B.; Zou, Q.; Xie, G.; Lin, S.; Wang, R.; Yang, X.; Chen, W.; Wang, Q.; Zhang, D.; Liu, Y.; Gong, R.; Ma, Z.; Lu, S.; Xiao, Y.; Gu, Y.; Zhang, J.; Yao, H.; Xu, K.; Lu, X.; Wei, G.; Zhou, J.; Fang, Q.; Cai, H.; Qiu, Y.; Sheng, J.; Chen, Y.; Liang, T.; BMJ 2020, 369, 1. [Crossref] |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access





